Identificación mediante ADN barcode de peces invasores en el manglar de Tumbes (Perú)
Identification by DNA barcode of invading fish in the mangrove of Tumbes (Peru)
![]()
![]()
![]() Alberto Ordinola-Zapata1,
* ; Zoila Raquel Siccha R.2 ; Pedro S.
Castillo -Carrillo.3 ; Carlos Luque S.2
Alberto Ordinola-Zapata1,
* ; Zoila Raquel Siccha R.2 ; Pedro S.
Castillo -Carrillo.3 ; Carlos Luque S.2
1 Facultad de Ingeniería Pesquera y Ciencias del Mar de la Universidad Nacional de Tumbes, Calle Los Ceibos S/N, Puerto Pizarro, Tumbes, Perú.
2 Laboratorio Costero Tumbes del Instituto del Mar del Perú, Calle José Olaya S/N, Nueva Esperanza, Tumbes, Perú
3 Facultad de Ciencias Agrarias de la Universidad Nacional de Tumbes, La Cruz, Tumbes, Perú
![]() *Autor correspondiente: aordinolaz@untumbes.edu.pe (A. Ordinola-Zapata).
*Autor correspondiente: aordinolaz@untumbes.edu.pe (A. Ordinola-Zapata).
A. Ordinola-Zapata https://orcid.org/0000-0002-9644-0531
![]() Z. R. Siccha https://orcid.org/0000-0002-5597-3948
Z. R. Siccha https://orcid.org/0000-0002-5597-3948
![]() P.
S. Castillo-Carrillo https://orcid.org/0000-0002-0255-1047
P.
S. Castillo-Carrillo https://orcid.org/0000-0002-0255-1047
DOI: 10.17268/manglar.2019.013
Resumen
El Perú es un país megadiverso que destaca por su biodiversidad de peces, entre ellos los del manglar de Tumbes. Este ecosistema se encuentra amenazado por diversas causas, una de ellas es la introducción de especies invasoras. La investigación se realizó entre junio de 2016 y agosto de 2018, tuvo como objetivo identificar, mediante la técnica de ADN barcode, los peces invasores en el manglar de Tumbes. Se recolectaron peces en 18 puntos de muestreo, estos fueron identificados por sus características morfológicas. Para la identificación mediante ADN barcode, se obtuvo sus secuencias de un fragmento del gen de la subunidad I de la citocromo oxidasa (COI), con las cuales se realizó su identificación en las bases de datos de Blast y Bold. También se realizó análisis filogenético, construyendo árboles basados en los métodos de Neighbor joining (NJ), descubrimiento automático de brecha de barcode (ABDG) y modelo generalizado mixto de Yule y coalescencia (GMYC), con los cuales se confirmó la asignación de especies. De los análisis realizados se concluyó la presencia de cuatro especies invasoras: Poecilia reticulata, Oreochromis mossambicus, O. niloticus y O. urolepis.
Palabras clave: Poecilia; Oreochromis; peces invasores; manglar; Tumbes.
Abstract
Peru is a megadiverse country that highlight for its biodiversity of fish, including there of the mangrove of Tumbes. This ecosystem is threatened by various causes, one of them is introduction of invasive species. The investigation was carried out between June 2016 and August 2018, with the objective of identifying, through the DNA barcode technique, the invading fish in the Tumbes mangrove. Fish were collected in 18 sampling points; these were identified by their morphological characteristics. For identification by DNA barcode, its sequences were obtained from a fragment of the gene of cytochrome oxidase subunit I (COI), with which we searched for its identification in the Blast and Bold databases. Phylogenetic analysis was also carried out, constructing trees based on the methods of Neighbor joining (NJ), automatic barcode discovery gap (ABDG) and model of generalized mixed of Yule and coalescence (GMYC), with which the allocation of species was confirmed. From the analyzes carried out the presence of four invasive species was concluded: Poecilia reticulata, Oreochromis mossambicus, O. niloticus and O. urolepis.
Keywords: Poecilia; Oreochromis; invasive fish; mangrove; Tumbes.
Introducción
Perú es uno de los 10 países más megadiversos a nivel mundial, esto se debe al gran número de especies que habitan los diversos ecosistemas que se hallan en su territorio (Shanee et al., 2017); en el norte del país se destaca el ecosistema de manglar de Tumbes el cual alberga una gran diversidad de peces (Luque, 2008). Los ecosistemas marinos, incluyendo a los manglares, están amenazados por diversas causas, entre ellas la introducción de especies invasoras (Giakoumi et al., 2018; Gorman, 2018).
El manglar de Tumbes de igual manera está amenazado por el cambio climático y actividades antrópicas (pesca, acuicultura, contaminación) (Takahashi y Martínez, 2015), a lo cual se suma el riesgo de la introducción de especies invasoras; las cuales se definen como especies no nativas, introducidas por la actividad humana, que pueden originar un impacto ambiental o económico negativo, pudiendo incluso amenazar a la salud humana (Amador-del Ángel et al., 2014; Makowski y Finkl, 2019).
Las especies invasoras se caracterizan por poseer altas tasas reproductivas y de propagación, amplia variabilidad genética, conjuntamente con una considerable plasticidad fenotípica. De igual manera poseen una distribución amplia por su característica oportunista (Mendoza, 2018).
El efecto ambiental de las especies invasoras, no se evidencian solamente en su impacto en la biodiversidad, también en la disponibilidad de nutrientes y agua; pudiendo perjudicar a la agricultura, ganadería y acuicultura (Nentwig et al., 2018a); además puede ser muy perjudicial para las especies de peces nativas, así se les ha responsabilizado por la desaparición del 54% de los peces a nivel mundial, del 70% de los de Norteamérica y 60% de los mexicanos (Amador-del Ángel et al., 2014).
En muchos ecosistemas de manglar americanos se ha reportado la presencia de peces invasores, como por ejemplo: pez león (Pterois volitans) en las costas atlánticas de Estados Unidos, México, Centroamérica, Colombia y Venezuela (Barbour et al., 2010; Lasso-Alcalá y Posada, 2010; Arbeláez y Acero, 2011; Pimiento et al., 2015; Chávez-López y Rocha-Ramírez, 2018).
También se ha reportado presencia de Poecílidos (Poecilia spp., Gambusia spp.) en manglares de Florida, México y Costa Rica (Pimiento et al., 2015; Chávez-López y Rocha-Ramírez, 2018; Global Invasive Species Database, 2019); de tilapias (principalmente Oreochromis niloticus, además de O. mossambicus y O. aureus) en Estados Unidos, México, Cuba, Costa Rica y Colombia (Schüttler, 2009; Lowe et al., 2012).
En cuanto al manglar peruano, se ha reportado la presencia de peces invasores de las especies O. niloticus y de P. reticulata en el manglar de San Pedro de Vice en Piura (Barrionuevo y Marcial, 2006) así como en el manglar de Tumbes (Luque, 2008). Ambos estudios han basado su identificación en caracteres morfológicos utilizando claves de identificación. Sin embargo, dicha forma de identificación está limitada por la plasticidad fenotípica, el dimorfismo sexual y la variabilidad genética, además que las claves no abarcan todos los estadios de desarrollo y son complejas para su uso, por lo que requieren de expertos taxonomistas (Zhang y Hanner, 2011).
Estas limitaciones, pueden ser superadas por la identificación molecular basada en el ADN barcode, que utiliza tradicionalmente un fragmento del gen que codifica la subunidad I de la citocromo oxidasa (COI), puesto que sus secuencias son bastante conservadas al nivel de especie.
Material y métodos
La investigación se realizó de junio de 2016 a agosto de 2018 en el Laboratorio de Sanidad Acuícola del Laboratorio Costero del Instituto del Mar del Perú (La Cruz, Tumbes, Perú). La población en estudio correspondió a los peces del manglar de Tumbes. La muestra para este estudio fue de 362 peces que fueron recolectados en 18 puntos de muestreo, utilizando atarrayas, redes, anzuelos y marcos de malla de tul. Los peces fueron transportados en baldes con hielo hasta el Laboratorio Costero de Imarpe Tumbes. Los ejemplares fueron identificados por taxónomos del proyecto usando diversas claves de identificación.
De cada ejemplar se realizó la amplificación de un fragmento del gen de la subunidad I de la citocromo oxidasa (COI), que fue realizada en un volumen de reacción de 25 µl, conteniendo 16,1 µl de agua ultrapura, 2,5 µl de desoxirribonucleótidos trifosfato (dNTPs, 8 mM), 2,5 µl de buffer 10X, 1,2 µl de cada primer (10 µM) y 0,5 µl de Taq polimerasa.
Los primers utilizados en la amplificación fueron los empleados por Ward et al. (2005):
Pareja 1:
Fish F1: 5’- TCAACCAACCACAAAGACATTG GCAC -3’
Fish R1: 5’- TAGACTTCT GGGTGGCCAAAGA ATCA -3’
Pareja 2:
Fish F2: 5’- TCGACTAATCATAAAGATATC GGCAC -3’
Fish R2: 5’-ACTTCAGGGTGACCGAAGAATC AGAA -3’
Que permitieron obtener un fragmento de ADN de aproximadamente 648 pb.
La amplificación mediante reacción de cadena de la polimerasa (PCR) se realizó en un termociclador programado con desnaturalización inicial a 94 °C por 4 min; seguido de 35 ciclos de desnaturalización a 94 °C por 1 min, hibridación por 1 min a 50 - 65 °C y extensión por 2 min a 72 °C; posteriormente a dichos 35 ciclos, se dio un paso de extensión final a 72 °C por cinco min. Los amplicones fueron visualizados en gel de agarosa al 1%, posteriormente fueron envíados a secuenciar por duplicado (cadenas forward y reverse) al extranjero.
Las secuencias de ADN fueron ensambladas con el programa Geneious 5.6, luego fueron alineadas en Mega 7. Las secuencias depuradas fueron usadas para realizar búsquedas en las bases de datos Blast y Bold a fin de poder identificar mediante ADN barcode a que especie correspondieron.
De los ejemplares identificados, se seleccionó para el análisis posterior sólo las secuencias nucleotídicas de aquellos que fueron determinados como especies invasoras según el listado expuesto por Lowe et al. (2004).
Se calcularon las distancias genéticas intra e interespecíficas, construyéndose un árbol Neighbor joining (NJ) con el modelo de sustitución de Kimura 2-parámetros (K2P) implementados en Mega 7. Se construyeron árboles filogenéticos mediante análisis de descubrimiento automático de brecha de barcode (ABDG) implementado en el site http://wwwabi.snv.jussieu.fr/public/abgd/(Puillandre et al., 2012) y del modelo generalizado mixto de Yule y coalescencia (GMYC), ejecutado a través de los programas BEAST 2.5.2 y R 3.5.2.
Resultados y discusión
De los 362 ejemplares recolectados e identificados, 15 correspondieron a especies invasoras: tilapias del género Oreochromis y piponas del género Poecilia. En la tabla 1 se muestra la identificación de cada ejemplar basada en sus características morfológicas, así como por ADN barcode.
Las tilapias de acuerdo a sus características morfológicas fueron identificadas como correspondientes a dos especies: Oreochromis mossambicus y O. niloticus. Sin embargo, usando ADN barcode y la búsqueda en la base de datos de Blast se identificaron como pertenecientes a cuatro especies: O. niloticus, O. urolepis, O. aureus y O. mossambicus (todas con un porcentaje de identidad y cobertura de 100%), mientras que usando ADN barcode y la base de datos de Bold se identificó a tres especies: O. aureus, O. urolepis y O. mossambicus con identidad de 100%.
Respecto a los poecílidos, mediante características morfológicas se identificó a Poecilia reticulata, mientras que mediante ADN barcode y la base de datos de Blast, se logró identificar sólo hasta el nivel de género (Poecilia sp.), y a través de Bold, como Poecilia mexicana (identidad de 99,85%).
Al realizar el análisis filogenético de las secuencias de ADN del fragmento del gen COI de los peces invasores, se observó que los árboles construidos con los métodos de Neighbor-Joining (NJ), ABGD y GMYC mostraron una estructura similar entre ellos, no habiendo contradicción entre los mismos respecto a nodos que pertenezcan a clados distintos.
Tabla 1. Especies de peces identificadas morfológicamente y mediante ADN barcode
|
Código de ejemplar |
Identificación morfológica |
|
Identificación mediante ADN barcode |
|||
|
|
Blast* |
|
Bold |
|||
|
Especie |
|
Especie |
|
Especie |
Identidad (%) |
|
|
LCT_228 |
O. mossambicus |
|
O. niloticus |
|
O. aureus |
100,00 |
|
LCT_229 |
O. mossambicus |
|
O. urolepis |
|
O. urolepis |
100,00 |
|
LCT_262 |
O. mossambicus |
|
O. niloticus/O. aureus |
|
O. aureus |
100,00 |
|
LCT_263 |
O. mossambicus |
|
O. urolepis |
|
O. urolepis |
100,00 |
|
LCT_264 |
O. mossambicus |
|
O. aureus/O. mossambicus |
|
O. mossambicus |
100,00 |
|
|
|
|||||
|
LCT_217 |
O. niloticus |
|
O. niloticus |
|
O. aureus |
100,00 |
|
LCT_218 |
O. niloticus |
|
O. niloticus |
|
O. aureus |
100,00 |
|
LCT_219 |
O. niloticus |
|
O. niloticus |
|
O. aureus |
100,00 |
|
LCT_220 |
O. niloticus |
|
O. niloticus |
|
O. aureus |
100,00 |
|
LCT_221 |
O. niloticus |
|
O. niloticus |
|
O. aureus |
100,00 |
|
|
|
|||||
|
LCT_916 |
P. reticulata |
|
Poecilia sp. |
|
P. mexicana |
99,85 |
|
LCT_917 |
P. reticulata |
|
Poecilia sp. |
|
P. mexicana |
99,85 |
|
LCT_918 |
P. reticulata |
|
Poecilia sp. |
|
P. mexicana |
99,85 |
|
LCT_919 |
P. reticulata |
|
Poecilia sp. |
|
P. mexicana |
99,85 |
|
LCT_920 |
P. reticulata |
|
Poecilia sp. |
|
P. mexicana |
99,85 |
* La identificación y cobertura de las secuencias buscadas en Blast fue de 100%.
Para poder discriminar mejor las especies identificadas, se utilizó los datos del árbol generado en base al análisis GMYC, tratando de consensuar los datos de tal manera que los ejemplares de la misma especie compartan clados (figura 1), allí se aprecia que los ejemplares con fondo en color rojo (códigos LCT 217, 218, 219, 221 y 220) es más probable que se traten de O. niloticus pues es la identificación que les otorgan sus caracteres morfológicos y sus secuencias barcode en Blast, lo que es respaldado a su vez por el árbol construido con GMYC que confirma que este grupo de ejemplares pertenecen a una sola especie. Por otra parte, los ejemplares con fondo en color lila es más probable que se traten de O. urolepis pues es la identificación que les otorgan sus secuencias ADN barcode en Blast y Bold, lo que se respalda a su vez en que sus secuencias forman un solo clado en el árbol GMYC confirmando que pertenecen a una sola especie. Estos ejemplares, en base a sus caracteres morfológicos fueron identificados como O. mossambicus, pero esto es menos probable pues del análisis filogenético mostrado en el árbol GMYC se observa que en realidad corresponden a especies distintas.
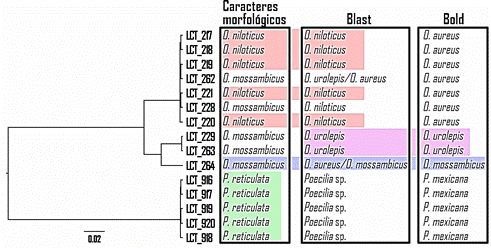
Figura 1. Comparación entre los resultados de la identificación usando caracteres morfológicos, las de ADN barcode (usando bases de datos de Blast y Bold) y el árbol filogenético usando GMYC. En fondo de colores se muestran los nodos en los que se establece un consenso de identificación ya sea por ser reconocida como pertenecientes a determinada especie por su morfología o por las bases de datos.
Tabla 2. Especies de peces invasores identificadas según consenso
|
Orden |
Familia |
Género |
Nombre científico |
Nombre común |
|
Cichliformes |
Cichlidae |
Oreochromis |
O. mossambicus |
Tilapia |
|
|
|
|
O. niloticus |
Tilapia nilótica |
|
|
|
|
O. urolepis |
Tilapia |
|
|
|
|
|
|
|
Cyprinodontiformes |
Poeciliidae |
Poecilia |
P. reticulata |
Pipona, millonario, gupi |
Con fondo de colores se muestran los nodos en los que se establece un consenso de identificación ya sea por ser reconocida como pertenecientes a determinada especie por su morfología o por las bases de datos. En el caso de los nodos LCT_262 y LCT_228, no se alcanzó un consenso, sin embargo debido a su ubicación en el árbol filogenético, lo más probable es que pertenezca al clado O. niloticus.
En el caso del clado que fue identificado como P. reticulata por sus caracteres morfológicos, no se encontró suficiente información en las bases de datos como para justificar su asignación a P. mexicana.
En el consenso logrado entre la identificación morfológica, y la de ADN barcode utilizando como bases de datos (Blast y Bold), así como con los árboles filogenéticos de NJ, ABGD y GMYC, se obtuvo que las especies de peces invasores identificadas en esta investigación fueron, las que se muestran en la tabla 2.
La identificación de peces invasores del manglar de Tumbes, difirió parcialmente entre la obtenida por la observación de caracteres morfológicos y aquella obtenida mediante ADN barcode (usando las bases de datos Blast y Bold, complementadas con los árboles filogenéticos construidos con los métodos NJ, ABGD y GMYC), esta situación ha sido observada en peces y otros grupos zoológicos (Valderrama-Aravena et al., 2014; Azmir et al., 2017; Hulley et al., 2018).
La técnica de ADN barcode permitió confirmar la identidad de algunos ejemplares; como se pudo determinar a través de la identificación en las bases de datos de Bold y Blast, así como en su posición dentro de determinados clados en los árboles filogenéticos (NJ, ABGD, GMYC). Los clados señalados mostraron una divergencia genética intraclado menor al 1%, e interclado mayor al 2%, lo cual es un indicador de un adecuado nivel de separación genética que permite establecer a los mismos como especies distintas, criterio que ha sido empleado por Zhang y Hanner (2011).
En esta investigación, el ADN barcode mostró ser útil para identificar especies de peces del manglar, hecho que es respaldado por Zhang y Hanner (2011), quienes señalan, que la identificación mediante ADN barcode (usando un fragmento del gen COI), es una herramienta adecuada para lograr una identificación confiable de especies de peces, por lo que algunos ictiólogos han propuesto la inclusión del ADN barcode como parte de la descripción formal de cada especie.
Las especies de peces invasores de los géneros Oreochromis y Poecilia, encontrados en el manglar de Tumbes, han sido reportadas en otros manglares del mundo, como por ejemplo Poecilia en manglares de Florida, México y Costa Rica (Pimiento et al., 2015; Chávez-López y Rocha-Ramírez, 2018; Global Invasive Species Database, 2019); Oreochromis en manglares de Estados Unidos, México, Cuba, Costa Rica y Colombia (Schüttler, 2009; Lowe et al., 2012). Especies de dichos géneros también han sido reportadas para los manglares del Perú como el de San Pedro de Vice (Barrionuevo y Marcial, 2006) y el manglar de Tumbes (Luque, 2008), cabe mencionar que estos dos últimos estudios utilizaron sólo identificación morfológica mediante la cual reportaron sólo las especies O. niloticus y P. reticulata, no reportando otras especies como O. mossambicus y O. urolepis.
La presencia de la especie invasora O. mossambicus en el manglar de Tumbes es preocupante, dado que la misma está incluida en la lista de las 100 especies invasoras más dañinas de Europa y del mundo (Lowe et al., 2004; Nentwig et al., 2018b); de igual manera es preocupante la presencia de P. reticulata la que si bien no ha sido incluida entre las 100 especies invasoras más dañinas del mundo, si ha sido incluida entre las 100 más dañinas en Europa (Nentwig et al., 2018b).
Conclusiones
Mediante la técnica de ADN barcode se logró confirmar la identificación a nivel de especie de ejemplares de peces invasores en el manglar de Tumbes, encontrándose ejemplares de tilapias de las especies Oreochromis niloticus, O. mossambicus y O. urolepis, así como de pipona (Poecilia reticulata).
Agradecimientos
Al Laboratorio Costero Tumbes del Instituto del Mar del Perú y a la Universidad Nacional de Tumbes, entidades que en asociación desarrollaron el Proyecto: “DNA barcode de las especies ícticas marinas, dulceacuícolas y del manglar de Tumbes”, financiado por el Fondo Nacional de Desarrollo Científico, Tecnológico y de Innovación Tecnológica según contrato Fondecyt - Imarpe –192-2015. Esta investigación se desarrolló como parte de la ejecución del citado proyecto.
Al personal técnico y científico del Laboratorio Costero Tumbes del Instituto del Mar del Perú, en particular a la Mg. Mervin Guevara Torres y Blgo. Manuel Vera Mateo, quienes colaboraron de manera significativa en la ejecución de esta investigación.
Referencias bibliográficas
Amador-del Ángel, L.E.; Wakida-Kusunoki, A.T.; Mendoza, R.; Koleff, P. 2014. Peces invasores en el sureste de México. En especies acuáticas invasoras en México, 425-433. México D.F. México.
Arbeláez, N.; Acero, A. 2011. Presencia del pez león Pterois volitans (Linnaeus) en el manglar de la bahía de Chengue, Caribe Colombiano. Bol. Invest. Mar. Cost. 40(2): 431-35.
Azmir, I.A., Esa, Y.; Amin, S.M.N.; Yasin, I.S. Md; Yusof, F.Z. Md. 2017. Identification of Larval Fish in Mangrove Areas of Peninsular Malaysia Using Morphology and DNA Barcoding Methods. Journal of Applied Ichthyology 33(5): 998-1006.
Barbour, A.; Montgomery, M.; Adamson, A.; Díaz-Ferguson, E.; Silliman, B. 2010. Mangrove Use by the Invasive Lionfish Pterois Volitans. Marine Ecology Progress Series 401: 291-94.
Barrionuevo, R.; Marcial, R. 2006. Ecología Tró-fica de la fauna acuática en el Manglar de San Pedro - Sechura. Universalia 11(2): 44-56.
Chávez-López, R.; Rocha-Ramírez, A. 2018. Report of Poecilia mexicana in a fish assemblage from a Gulf of Mexico blind estuary. Poeciliid Research 8(1): 6-12.
Giakoumi, S.; Pey, A.; Di Franco, A.; Francour, P.; Zafer, K.; Arda, Y.; Raybaud, V.; Guidetti, P. 2018. Exploring the relationships between marine protected areas and invasive fish in the world’s most invaded sea. Open Science Framework 9(1): e01809.
Global Invasive Species Database. 2019. Species profile: Poecilia reticulata. Disponible en:
http://www.iucngisd.org/gisd/species.php?sc=683.
Gorman, D. 2018. Historical Losses of Mangrove Systems in South America from Human-Induced and Natural Impacts. En Threats to Mangrove Forests, 155-171. Cham: Springer International Publishing.
Hulley, E.N.; Taylor, N.D.J.; Zarnke, A.M.; Somers, C.M.; Manzon, R.G.; Wilson, J.Y.; Boreham, D. R. 2018. DNA Barcoding vs. Morphological Identification of Larval Fish and Embryos in Lake Huron: Advantages to a Molecular Approach. Journal of Great Lakes Research 44(5): 1110-16.
Lasso-Alcalá, O.; Posada, J. 2010. Presence of the invasive red lionfish, Pterois volitans (Linnaeus, 1758), on the coast of Venezuela, southeastern Caribbean Sea. Aquatic Invasions 5(Supplement 1): S53-59.
Lowe, M.R.; Wu, W.; Peterson, M.S.; Brown-Peterson, N.J.; Slack, W.T.; Schofield, P. J. 2012. Survival, Growth and Reproduction of Non-Native Nile Tilapia II: Fundamental Niche Projections and Invasion Potential in the Northern Gulf of Mexico. PLoS ONE 7(7): e41580.
Lowe, S.; Browne, M.; Boudjelas, S.; De Poorter, M. 2004. 100 de las Especies Exóticas Invasoras más dañinas del mundo. Una selección del Global Invasive Species Database. Grupo Especialista de Especies Invasoras (GEEI) de la UICN. Disponible en:
http://www.issg.org/pdf/publications/worst_100/spanish_100_worst.pdf.
Luque, C. 2008. Estudio de la diversidad hidrobiológica en Tumbes. Instituto del Mar del Perú. Tumbes, Perú. 36 pp.
Makowski, C.; Finkl, C. W. 2019. Impacts of Invasive Species on Coastal Environments: Coasts in Crisis. Vol. 29. Coastal Research Library. Cham: Springer International Publishing. Boca Ratón, USA. 482 pp.
Mendoza, E. 2018. Manejo de peces exóticos invasores en la microcuenca del Río Jalpan, Querétaro. Tesis de Maestra en Gestión Integrada de Cuencas. Universidad Autónoma de Querétaro. 120 pp.
Nentwig, W.; Bacher, S.; Kumschick, S.; Pyšek, P.; Vilà, M. 2018a. Correction to: More than “100 Worst” Alien Species in Europe. Biological Invasions 20(6): 1623-1623.
Nentwig, W.; Bacher, S.; Kumschick, S.; Pyšek, P.; Vilà, M. 2018b. More than “100 Worst” Alien Species in Europe. Biological Invasions 20(6): 1611-21.
Pimiento, C., Nifong, J.C.; Hunter, M.E.; Monaco, E.; Silliman, B.R. 2015. Habitat Use Patterns of the Invasive Red Lionfish Pterois Volitans: A Comparison between Mangrove and Reef Systems in San Salvador, Bahamas. Marine Ecology 36(1): 28-37.
Puillandre, N.; Lambert, A.; Brouillet, S.; Achaz, G. 2012. ABGD, Automatic Barcode Gap Discovery for Primary Species Delimitation: ABGD, Automatic Barcode Gap Discovery. Molecular Ecology 21(8): 1864-77.
Schüttler, E. 2009. Especies exóticas invasoras en las Reservas de Biosfera de América Latina y el Caribe: un informe técnico para fomentar el intercambio de experiencias entre las Reservas de Biosfera y promover el manejo efectivo de las invasiones biológicas. UNESCO. Montevideo, Uruguay. 305 pp.
Shanee, S.; Shanee, N.; Monteferri, B.; Allgas, N.; Alarcon, A.; Horwich, R.H. 2017. Protected Area Coverage of Threatened Vertebrates and Ecoregions in Peru: Comparison of Communal, Private and State Reserves. Journal of Environmental Management 202: 12-20.
Takahashi, K.; Martínez, A. 2015. Impacto de la variabilidad y cambio climático en el ecosistema de Manglares de Tumbes, Perú. Instituto Geofísico del Perú - Ministerio del Ambiente. Lima, Perú. 90 pp.
Valderrama-Aravena, N.; Pérez-Araneda, K.; Avaria-Llautureo, J.; Hernández, C.E.; Lee, M.; Brante, A. 2014. Diversidad de Nematodos Marinos de Chile Continental y Antártico: Una Evaluación Morfológica y Molecular. Revista de Biología Marina y Oceanografía 49(1): 147-55.
Ward, R.D.; Zemlak, T.S.; Innes, B.H.; Last, P.R.; Hebert, P.D.N. 2005. DNA Barcoding Aus-tralia’s Fish Species. Philosophical Transac-tions of the Royal Society B: Biological Sciences 360(1462): 1847-57.
Zhang, J.-B.; Hanner, R. 2011. DNA Barcoding Is a Useful Tool for the Identification of Marine Fishes from Japan. Biochemical Systematics and Ecology 39(1): 31-42.