Variabilidad Genética en Pouteria lucuma mediante marcadores ISSR
Genetic variability in Pouteria lucuma using ISSR markers
Carlos Helí
Quijano Jara*; Zulita Adriana Prieto Lara
Facultad de Ciencias Biológicas, Universidad Nacional
de Trujillo, Av. Juan Pablo II s/n – Ciudad Universitaria, Trujillo, Perú.
*Autor corresponsal: cquijano@unitru.edu.pe
(C. Quijano).
ID ORCID de los autores
C. Quijano: ![]() https://orcid.org/0000-0001-9269-9864
https://orcid.org/0000-0001-9269-9864
Z. Prieto: ![]() https://orcid.org/0000-0002-9782-1782
https://orcid.org/0000-0002-9782-1782
DOI: http://dx.doi.org/10.17268/manglar.2020.002
RESUMEN
La lúcuma es un frutal
oriundo de Sudamérica, se produce en Colombia, Ecuador, norte de Chile y Perú,
siendo los principales productores a nivel mundial con una participación de
88%. Sin embargo, existe escasa información de su base genética, los cuales son
la base para posteriores evaluaciones agronómicas, mejoramiento genético,
selección y reproducción de materiales con características deseables en un
programa de fomento y diversificación de la producción agrícola. Razón por la
cual se planteó llevar a cabo una caracterización genética de poblaciones de Pouteria lucuma “lucuma” procedentes del caserío de Naubamba
en el Distrito de Usquil. Los resultados muestran ADN
extraído de buena concentración y calidad, así como amplificación con todos los
marcadores ISSR empleados y alto número de bandas polimórficas con una
proporción de loci polimórficos altos por marcador.
Palabras clave: Pouteria lucuma; Variabilidad; ISSR; Naubamba.
ABSTRACT
Lucuma is a fruit tree native to South America, it is produced in Colombia, Ecuador, north-ern Chile, and Peru, being the main producers worldwide with an 88% participation. How-ever, there is little information on its genetic base, which is the basis for further agronomic evaluations, genetic improvement, selection and reproduction of materials with desirable characteristics in a program to promote and diversify agricultural production. Reason why it was proposed to carry out a genetic characterization of populations of Pouteria lucuma “lucuma” from the village of Naubamba in the District of Usquil. The results show DNA extracted from good concentration and quality, as well as amplification with all used ISSR markers and high number of polymorphic bands with a high polymorphic loci ratio per marker.
Keywords: Pouteria lucuma; variability; ISSR; Naubamba.
INTRODUCCIÓN
La región andina, es
uno de los mayores centros de domesticación de plantas, en concordancia con el
desarrollo de civilizaciones que generaron una agricultura autóctona con la domesticación
de un gran número de especies de plantas, siendo empleadas durante un tiempo
prolongado hasta la llegada de los españoles y el consiguiente reemplazo de
muchas de las especies nativas por especies importadas de Europa (Jacobsen,
2003).
Entre las diversas
plantas domesticadas existen numerosas especies frutales, tales como la
chirimoya (Annona cherimola),
varias granadillas (Passiflora spp.), naranjilla (Solanum
quitoense), pacae (Inga
feuillei), varias papayas de altura (Vasconcella spp.),
pepino dulce (Solanum muricatum),
tomate de árbol (Cyphomandra betacea) y lúcuma (Pouteria
lucuma) perteneciente a la familia Sapotaceae (Sanjinés, 2006).
En nuestro país la
familia Sapotaceae presenta 10 géneros y 90
especies, todos árboles, considerando a Pouteria como
el género más rico en especies (León, 2006). Dentro del género Pouteria tenemos a Pouteria
lucuma “lúcuma”, oriunda de los valles
Interandinos de Ecuador, Chile y Perú, la cual crece entre 200 y 2800 msnm,
desarrollándose bien en diversidad de suelos por lo que puede calificarse como
rustica (Huamantupa, 2008).
Es cultivada en
Colombia, Ecuador, norte de Chile y Perú, siendo los principales productores a
nivel mundial con una participación de 88%. En el Perú, Lima concentra la
producción a nivel nacional con 676 hectareas
cultivadas y 6711 toneladas producidas y en segundo lugar Ica con 166 hectáreas
y una producción de 1906 toneladas y en tercer lugar La Libertad con 119
hectáreas y una producción de 944 toneladas, existiendo producción en otros
departamentos en menor medida, según el plan nacional de cultivos 2018-2019.
La lúcuma tiene un
fruto versátil que posee un sabor distintivo; siendo la pulpa utilizada en gran
diversidad de postres, además, es un ingrediente frecuente de los batidos de
leche. En Perú y Chile se la seca para obtener una harina que se utiliza para
varios fines: refrescos, helados y dulce, además de ser una fuente de fibra, b
– caroteno, compuestos fenólicos, vitaminas A, B1, B2, B5, C y también
minerales como el calcio, hierro y fósforo (León, 2000).
Investigaciones a
cargo del Instituto Nacional de Investigación Agraria (INIA) y la Asociación de
Productores de Lúcuma (Prolúcuma) han llegado a
identificar cuatro variedades de lúcuma de seda como las más apropiadas para el
cultivo, tanto por su calidad como por su productividad. Estas variedades han
venido sembrando en los departamentos de Lima, Ica y en la costa de La
Libertad. En los otros departamentos se puede encontrar muchos cultivos
silvestres, que crecen prácticamente solos, pero dan frutos de no muy buena
calidad, mayormente de lúcuma de palo (Mostacero, 2002).
Debido al valor
potencial que representa este material genético se hace necesario estudiar la
caracterización y conservación, ya que la caracterización in situ de especies
nativas es el paso inicial para de-terminar características morfológicas y
fenológicas que conforman la variabilidad genética. Dichos estudios son la base
para posteriores evaluaciones agronómicas, mejoramiento genético, selección y
reproducción de materiales con características deseables en un programa de
fomento y diversificación de la producción agrícola (Álvarez, 2006; dos Santos et
al., 2016; Wu, 2019).
Según Utrera y
Martínez (1994), la caracterización es la descripción de un carácter, es
sinónimo de distinguir, a marcar, diferenciar, o separar en tipos, clases o
categorías, siendo los caracteres morfo-lógicos utilizados en la
caracterización de poblaciones y en la descripción de cultivares para su
inscripción. Sin embargo, en algunos casos estos no proveen una cuantificación
adecuada de la variabilidad genética. En ese sentido, la caracterización
genética se puede realizar a diferentes niveles, des-de los marcadores
cromosómicos (Arends, 1976; Majourhat,
2003); bioquímicos como las isoenzimas (Rodríguez,
1999; Becerra y Paredes, 2000), así como los marcadores basados en secuencias
de ADN los cuales tienen la ventaja de no estar influenciadas por el ambiente,
tal como los RAPDs (Random amplified polymorphic DNA) (Rolim, 2011; Puecher, 1998;
Rodríguez-Rojas, 2012).
Otro marcador
dominante es el ISSR (Inter Simple Sequence Repeats), donde se usan cebadores compuestos de
repeticiones de di, tri, tetra o penta-nucleótidos,
que se hibrida a la región de microsatélites genómicos, razón por la cual no es
necesario tener información del genoma; las secuencias diana de los cebadores
ISSR son abundantes en todo el genoma eucariota y evolucionan rápidamente,
conllevando a presentar gran cantidad de loci
polimórficos que otros marcadores dominantes como RAPD (Ansari
et al., 2012; dos Santos et al., 2016).
En Pouteria
lucuma “lúcuma”, se han realizado diversos estudios,
respecto a los metabolitos primarios y secundarios (Fuentealba, 2016), la
presencia de flavonoides, así como su rendimiento de la harina de lúcuma
(Lavado, 2012); sin embargo, no existe información respecto a caracterización
genética a nivel de su variabilidad genética lo cual permite, determinar el
comportamiento de la especie, el éxito reproductivo individual. Los datos
moleculares mejoran o aún permiten la aclaración de la filogenia, y
proporcionan el conocimiento básico para la taxonomía, la domesticación y la
evolución de comprensión lo cual hace necesario llevar a cabo estudios de
caracterización genética de Pouteria lucuma como un paso inicial, para plantear posteriores
programas de mejoramiento genético, lo cual permitirá mejorar las
características de este cultivo y por lo tanto la calidad de los productos a
exportar; por lo cual se plantea como objetivo del presente trabajo determinar
la variabilidad genética en muestras de P. lucuma
procedentes del caserío de Naubamba del Distrito de Usquil mediante marcadores ISSR.
MATERIAL Y MÉTODOS
Material
biológico
Utilizando bolsas
herméticas, se colectaron muestras hojas jóvenes de individuos de la especie Pouteria lucuma (Ruiz&Pav.) Kuntze, en el
caserío Naubamba en el Distrito de Usquil – Otuzco, las cuales
fueron almacenadas a -40 °C hasta el momento en que fueron necesarias para
ejecutar los protocolos de extracción de ADN en Pouteria
lucuma. Se seleccionaron las hojas jóvenes, sin
signos de marchitez, ni daño.
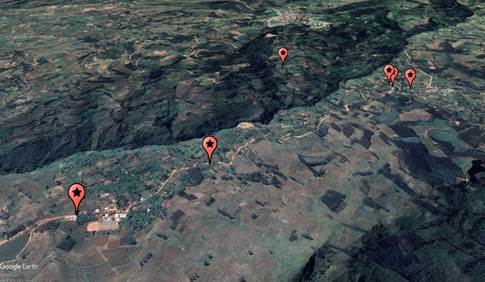
Figura 1. Mapa de la ubicación de
puntos de colecta de muestras en el caserío de Naubamba
en el Distrito de Usquil – Otuzco.
Protocolo
de extracción de ADN
Se usaron las hojas
previamente colectadas para obtener muestras de 100 mg (peso fresco). Se
trituraron las hojas hasta un polvo fino con nitrógeno líquido en un mortero
congelado y esterilizado. Después, el polvo se colocó en tubos de microcentrifuga de 1,5 mL y se
añadió 500 μL de amortiguador de extracción de
ADN (2% (w/v) CTAB (hexadeciltrimetil-amonio
bromuro), 100 mM Tris-HCI pH 7,5; 20 mM EDTA (ácido ethi-lenediaminotetracetico,
sal disodio, dihidrato-Na2); 1,4 M NaCL, 1% (w/v) polivinilpirrolidona
(PVP) esta mezcla fue mantenida a 60 °C por 30 min en termostato. Cumplido el
tiempo de incubación, la muestra se centrifugó a 14 000 rpm por 5 minutos, se
transfirió el sobrenadante a un nuevo tubo eppendorf
y después añadió un volumen de cloroformo: alcohol isoamílico
(24:1), se homogenizo mediante vortex por 5 segundos
y posteriormente se centrifugó la muestra a 14 000 rpm por 1 minuto para
separar las fases. La fase acuosa (superior) fue transferida a un nuevo tubo.
Este procedimiento de extracción se repitió hasta que la fase superior es
clara. A la fase acuosa se le agregó 0,7 volumenes de
Isopropanol helado incubándose a -20 °C durante 15
minutos. Pasado este tiempo se centrifuga la muestra a 14 000 rpm por 10
minutos, se elimina el sobrenadante y se agrega 500 ul
de etanol al 70% helado, posteriormente se elimina el etanol y se secó la
muestra en un concentrador o a temperatura ambiente; finalmente se disolvió el
pellet en 50 ul de buffer TE (Tris 10 mM pH 8, EDTA 1 mM), en una
congeladora a -20 °C hasta su utilización (Doyle y Doyle; 1990).
Determinación
de la concentración de ADN
Para determinar la
concentración y calidad de ADN, se procedió a medir las muestras, en un
espectro-fotómetro Nanodrop marca Thermo
Scientific modelo OneC del
laboratorio de servicios de la Universidad Nacional de Trujillo.
Preparación del Master Mix
En un tubo de microcentrifuga de 1,5 ml, se preparó un MasterMix (GoTaq® DNA Polymerase), mezclando todos los componentes necesarios
para llevar a cabo la PCR, posteriormente se repartió en tubos de PCR (Tubos de
0,1 ml estériles) 19,0 ul de la mezcla, finalmente se
le agregó 1ul de ADN a cada tubo de PCR, luego se colocaron los tubos de PCR en
el termociclador marca Applied
Biosystem modelo Veriti de
0,1 ml.
Tabla 1
Master Mix para muestras de ADN
de hojas de Pouteria lucuma
del caserío de Naubamba utilizando ISSR
|
Reactivo |
1X |
19.5X |
|
Buffer 5x Sin Color |
5 ul |
78 |
|
dNTPs (10mM de |
0,5 ul |
7,8 |
|
MgCl 25mM |
2,5 ul |
39 |
|
Primer F |
1 ul |
19,5 |
|
Taq pol DNA |
0,125 ul |
1,95 |
|
Agua Ultrapura |
13,88 ul |
228,15 |
|
ADN |
1 ul |
|
|
TOTAL |
20 ul |
390 |
Tabla 2
Condiciones de Amplificación para marcadores ISSR
|
ETAPA |
Etapa |
Tiempo |
Temperatura |
|
1
Ciclo inicial |
Desnaturalización |
5' |
95°C |
|
2
Ciclo (35 veces) |
Desnaturalización |
30" |
94°C |
|
Hibridación |
45" |
50 – 52 °C |
|
|
Extensión |
2´ |
72 °C |
|
|
Extensión Final |
Extensión final |
7´ |
72 °C |
Tabla 3
Nombre y secuencia de marcadores ISSR usados en la
amplificación por PCR en muestras de ADN de hojas de Pouteria
lucuma del caserío de Naubamba
|
Primer ISSR |
Secuencia de nucleótidos (5’- 3’) |
|
UBC M825 |
ACACACACACACACACT |
|
UBC M844 |
CTCTCTCTCTCTCTCTRC |
|
UBC M849 |
GTGTGTGTGTGTGTGTYA |
|
UBC M854 |
TCTCTCTCTCTCTCTCRG |
|
UBC M856 |
ACACACACACACACACYA |
Electroforesis
Se llevó a cabo
electroforesis en gel de agarosa al 1%; se utilizaron 5 ul
de cada muestra de ADN, la cual se mezcló con 2 ul de
Fluorescent DNA Loading dye marca GeneON para
posteriormente cargar la mescla en el gel de agarosa y realizar el corrido
electroforético durante 30 minutos a 100V, culminado el corrido electroforético
se procedió a visualizar las bandas correspondientes a las muestras de ADN en
el fotodocumentador CHEMIDOC XRS de la marca Biorad (Sambrook, 2001).
Análisis de datos de ISSR-PCR
Se generó una matriz
binaria de ausencia (cero) y presencia (uno) (Jedrzejczyk
y Rewers, 2018; Wu, 2019),
luego se determinaron algunos parámetros genéticos utilizando el paquete
estadístico Infogen (Balzarini,
2016).
RESULTADOS Y DISCUSIÓN
Uno de los aspectos
importantes en la caracterización en base a marcadores moleculares de ADN es la
extracción de ADN de buena concentración y calidad, siendo en vegetales un
punto crítico debido a la gran cantidad y diversidad de metabolitos secundarios
que producen, los cuales cumplen muchas funciones importantes en estos
organismos, sin embargo pueden hacer el proceso de extracción sumamente
complicado, debido a que pueden coprecipitar con el
ADN durante la extracción, los complejos de polisacáridos por ejemplo pueden
provocar que la fase acuosa sea excesivamente vis-cosa complicando la
separación del ADN, o pueden adherirse a la molécula de ADN dificultando el
acceso a las enzima como las polimerasas o las enzimas de restricción, siendo
necesario elegir un método que se adecue al material biológico con el cual se
va a trabajar (Ferreira, 1998; Sánchez-Coello et al., 2012). Uno de los parámetros
empleados para evaluar la calidad de los ácidos nucleicos es la
espectrofotometría, la cual se fundamenta en la gran capacidad de los ácidos
nucleicos para absorber el paso de la luz a una longitud de onda de 260
nanómetros, permitiendo medir la concentración de ADN
así como determinar la pureza del ADN, mediante el ratio 260/280 el cual debe
encontrarse en un rango entre 1,7 a 2,0 para ser considerado de buena calidad (Sambrook, 2001).
Tabla 4
Valores de
Concentración y calidad del ADN en muestras de hojas de Pouteria
lucuma “lucuma” del
caserío de Naubamba
|
Muestra |
Concentración ADN (ng/uL) |
A260/A280 |
|
Muestra 1 |
67,0 |
1,95 |
|
Muestra 2 |
485,6 |
2,06 |
|
Muestra 3 |
576,3 |
2,06 |
|
Muestra 4 |
188,1 |
2,00 |
|
Muestra 5 |
67,7 |
1,89 |
|
Muestra 6 |
85,5 |
2,01 |
|
Muestra 7 |
216,7 |
2,02 |
|
Muestra 8 |
66,1 |
1,96 |
|
Muestra 9 |
69,7 |
2,05 |
|
Muestra 10 |
85,1 |
2,06 |
|
Muestra 11 |
49,5 |
2,07 |
|
Muestra 12 |
76,5 |
2,03 |
|
Muestra 13 |
49,4 |
2,07 |
|
Muestra 14 |
57,2 |
2,08 |
|
Muestra 15 |
79,8 |
2,00 |
|
Muestra 16 |
65,2 |
2,00 |
|
Muestra 17 |
122,1 |
2,05 |
|
Muestra 18 |
35,5 |
1,99 |
|
Promedio |
135,7 |
2,0 |
Como se observa en los
resultados de extracción de ADN (Tabla 4) en las muestras de Pouteria lucuma
“lúcuma”, muestreadas en el caserío de Naubamba
distrito de Usquil se han obtenido valores adecua-dos
de concentración de ADN con un promedio de 135,7 ng/uL y con una calidad promedio de 2,0; estos resultados de
calidad concuerdan con los valores obtenidos por Sánchez (2017), donde se
trabajó con marcadores SSR en Solanum tuberosum, logrando, sin embargo, concentraciones de
ADN mayores (481,9 ng/ul)
pero con degradación; sin embargo no fue determinante al momento de la
amplificación de los marcadores por requerirse bajas concentraciones de ADN.
Respecto a la
amplificación mediante reacción en cadena de la polimerasa mediante el uso de
marca-dores ISSR, se eligió estos marcadores moleculares, por las ventajas que
ofrece como son: la alta variación que se detecta, así como en su
reproducibilidad, no requiere de altas concentraciones de ADN. No es necesario
conocer la secuencia del genoma del organismo en estudio. Pueden visualizarse
tan-to en geles de agarosa como de acrilamida y son sencillos de montar,
rápidos, eficientes y poco costosos (Eguiarte et al.,
2007). Asimismo, presentar una mayor reproducibilidad y un mayor porcentaje de
polimorfismo que los otros marcadores dominantes tales como los marcadores RAPDs, lo cual fue confirmado con los resultados obtenidos
(Figuras 2 a 6), observando amplificación en todos los marcadores ISSR
utilizados y polimorfismo; esto contrasta con los resultados obtenidos por Guasmi (2012) en Hordeum vulgare, donde los datos mostraron que el porcentaje de
bandas polimórficas RAPD (100%) fue mayor que el de ISSR (66,67%), además el
número medio de bandas de amplificación RAPD (5,66) fue mayor que el de ISSR
(3), siendo el número total de bandas poli-mórficas (17) detectadas por tres
cebadores RAPD fue mucho mayor que el de los tres cebadores ISSR (6),
considerando que los marcadores RAPD eran superiores a los marcadores ISSR en
la capacidad de revelar bandas más informativas en una sola amplificación
(Tabla 5).

Figura 2. Productos de
amplificación por PCR con el marcador 825 a partir de muestras de ADN de hojas
de Pouteria lucuma
del Caserío de Naubamba Distrito de Usquil.
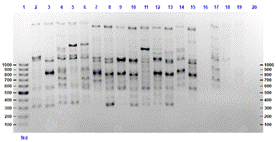
Figura 3. Productos de amplificación por PCR con el marcador 844 a partir de
muestras de ADN de hojas de Pouteria lucuma del Caserío de Naubamba
Distrito de Usquil.
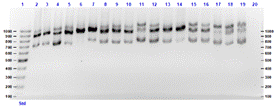
Figura 4. Productos de amplificación por PCR con el marcador 849 a partir de
muestras de ADN de hojas de Pouteria lucuma del Caserío de Naubamba
Distrito de Usquil.
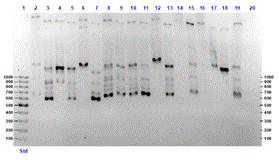
Figura 5. Productos de amplificación por PCR con el marcador 854 a partir de
muestras de ADN de hojas de Pouteria lucuma del Caserío de Naubamba
Distrito de Usquil.
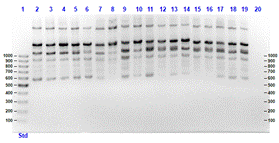
Figura 6. Productos de amplificación por PCR con el marcador 856 a partir de
muestras de ADN de hojas de Pouteria lucuma del Caserío de Naubamba
Distrito de Usquil.
El proporción de Loci Polimorficos (PMF(95),
muestran que los primers M844 y M854, presentan los
mayores valores de loci polimoficos:
respecto a la información de contenido polimórfico, fluctúa entre 0,19 y 0,32,
con un promedio de 0,26 (Tabla 6), valores bajos si lo comparamos con los
resultados en otras investigaciones en diferentes especies como Mentha L (Jedrzejczyk y Rewers, 2018) evaluada con marcadores ISSR, con un PIC
promedio de 0,434 o comparado con el PIC promedio en 30 accesiones de Solanum tuberosum,
con un promedio de 0,493 mediante marcadores microsatélites (Sánchez, 2017);
esto se podría deber al tipo de marcador empleado, a la cantidad de marcadores
o también debido a que la población no presenta gran variabilidad genética.
Tabla 5
Tabla resumen de amplificación con marcadores ISSR en
muestras de hojas de Pouteria lucuma del caserío de Naubamba
|
Resumen |
Cantidad de casos |
|
Muestras |
18 |
|
Muestras duplicadas |
0 |
|
Bandas (número) |
40 |
|
Patrón bandas duplicadas |
0 |
|
Bandas monomórficas |
9 |
|
Bandas polimorficas (%) |
77,50 |
|
Primers |
5 |
Tabla 6
Resumen por primer de
marcadores ISSR en muestras de hojas de Pouteria
lucuma del caserío de Naubamba
|
Primer |
BP |
BM |
BT |
PMF(95) |
PIC |
|
M825 |
4 |
3 |
7 |
0,57 |
0,24 |
|
M844 |
12 |
0 |
12 |
1,00 |
0,29 |
|
M849 |
2 |
1 |
3 |
0,67 |
0,19 |
|
M854 |
11 |
0 |
11 |
1,00 |
0,24 |
|
M856 |
2 |
5 |
7 |
0,29 |
0,32 |
|
Total |
31 |
9 |
40 |
0,71 |
0,26 |
Número de bandas polimórficas (BP), Número de bandas monomórficas (BM), Número de bandas totales (BT),
Proporción de loci polimórficos (PMF(95)),
Contenido de información polimórfica (PIC) promedio.
Los valores obtenidos,
además, son bajos en comparación con otras investigaciones como la realizada
por dos Santos et al. (2016) trabajando en Mimosa caesalpiniaefoli
donde a partir de 7 marcadores ISSR obtuvieron un total de 78 loci, además valores más altos de contenido de información
polimórfica promedio por marcador; sin embargo, respecto a la cantidad de loci polimórficos presen-tan valores cercanos, 41 para
Mimosa y 40 para la presente investigación, esto podría estar en función de la
población estudiada.
Recientemente se ha
analizado la variación morfológica y molecular en P. lucuma,
destacando moderada o baja variabilidad genética de las poblaciones colectadas,
probablemente por tratarse de colectas en huertas familiares, a diferencia de
la presente investigación donde evaluamos poblaciones de P. lucuma de zonas alejadas de la provincia de Otuzco, observándose de forma preliminar resultados que
demuestran variabilidad genética en las poblaciones muestreadas; pudiendo
considerarse a futuro a estas poblaciones para programas de selección de
plantas resistentes a enfermedades y producción, enmarcado en un programa de
mejoramiento genético, tal como se sugiere en la tesis (Borbor, 2017).
CONCLUSIONES
Se logró extraer ADN
de buena concentración y calidad a partir de hojas colectadas el caserío de Naubamba, distrito de Usquil. Se
logró amplificación con todos los marcadores ISSR empleados y alto número de
bandas polimórficas con una proporción de loci
polimórficos altos por marcador. Se sugiere continuar con investigaciones de
esta especie de importancia económica mediante el empleo de otros marcadores
moleculares.
AGRADECIMIENTOS
Agradecimientos al
biólogo Victor Sanchez
Cabrera por el apoyo en la colecta del material biológico y la
bachiller Silvana Carolina Ruiz Agurto por el
apoyo durante la ejecución de la investigación en la etapa de Laboratorio.
REFERENCIAS BIBLIOGRÁFICAS
Álvarez, Z.; Bravo, L.; Tagami, R. 2006. Plan de negocios para la industrialización y exportación de lúcuma de seda. Cuad. Difus. 11(21): 97-114.
Arends, J. 1976. Somatic Chromosome Number of some African Sapotaceae. Acta Bot. Neerl. 1976: 449-457.
Ansari, S.A.; Narayanan, C.; Wali,
S.A.; Kumar, R.; Shukla, N.; Rahangdale, SK. 2012.
ISSR markers for analysis of molecular diversity and genetic structure of
Indian teak (Tectona grandis
Lf) populations. Annals of Forest Research
55(1): 11-23.
Becerra, V.; Paredes,
M. 2000. Uso de marcadores bioquímicos y moleculares en estudios de diversidad
genética. Agricultura Técnica 60(3): 270-281.
Balzarini, M.; Di Rienzo, J. InfoGen.
2016. FCA, Universidad Nacional de Córdoba, Argentina. Disponible en:
www.info-gen.com.ar
Borbor, M. 2017.
Variación morfológica y molecular de la lúcuma (Pouteria
lucuma [R et. Pav] O. Kze) y su contribución al manejo sustentable de los huertos
de Yaután y Laredo. Tesis para optar por el grado de Doctoris Philosophiae en
Agricultura Sustentable. Universidad Nacional Agraria la Molina. 222 pp.
Doyle, J.; Doyle, J.
1990. Isolation
of plant DNA from fresh tissue. Focus 12: 13–15.
dos Santos, F.;
Vasconcelos, M.; de Almeida, F.; dos Santos, C.; Cardoso F. 2016. Teixeira das Chagas, Kyvia Pontes. ISSR molecular markers for the study of the
genetic diversi-ty of Mimosa caesalpiniaefolia
Benth. Idesia (Arica) 34(3): 47-52.
Eguiarte, L.; Souza, V.; Aguilar, X. 2007. Ecología Molecular. Editorial SEMARNAT.
México.
Ferreira, M.; Grattapaglia, D. 1998. Introdução
ao Uso de Marcadores Moleculares em
Análise Genética. Brasília
1: 121-123.
Fuentealba, C.; Gálvez, L.; Cobos, A.; Olaeta, J., Defilippi, B.; Chirinos, R.; Campos, D.; Pedreschi, R. 2016. Characterization of main primary and secondary metabolites and in vitro antioxidant and anti-hyperglycemic properties in themeso carp of three biotypes of Pouteria lucuma. Food Chem. 190: 403-411.
Guasmi, F.; Elfalleh,
W.; Hannachi, H.; Feres,
K.; Touil, L.; Marzougui,
N.; Triki, T.; Ferchichi,
A. 2012. The Use of ISSR and RAPD Markers for Genetic Diversity among South Tuni-sian Barley. ISRN Agronomy Article ID 952196. 10
pp.
Huamantupa, I. 2008. Frutales Nativos Silvestres consumidos en los mercados locales y
zonas rurales de la Amazonía Peruana (Departamentos de Cusco, Loreto y Madre de
Dios). Rev. Q'EUÑ 1(2): 26-31.
Jacobsen, S.; Mujica,
A.; Ortiz, R. 2003. La Importancia de los Cultivos Andinos. Rev. Vzlana. de Soc. y Ant. 13(36):
14-24.
Jedrzejczyk, I.; Rewers, M. 2018. Genome size and ISSR markers for Mentha L. (La-miaceae) genetic diversity assessment and species identification. Industrial Crops and Products 120: 171-179.
Lavado, M.; Yenque,
J.; Robles, R. 2012. Estudio de rendimiento de harina de
lúcuma a partir del fruto fresco. Revista de la Facultad de Ingeniería
Industrial 15(1): 127-130.
León, B. 2006. Sapotaceae endémicas del Perú. Rev. peru.
Biol. 13(2): 608 – 609.
León, J. 2000.
Botánica de cultivos tropicales. 3ª ed. San José: Instituto Interamericano de
Cooperación para la Agricultura.
Majourhat, K.; Jabbar, Y.; Araneda, L.; Zeinalabedini, M.; Hafidi, A.; Martínez-Gómez, P. 2007. Karyotype characterization of Argania spinosa (L.) Skeel (Sapotaceae). South African Journal of Botany 73: 661–663.
Mostacero, J.; Mejia, F.; Gamarra, E. 2002. Taxonomía de las Fanerógamas
útiles del Perú. Trujillo – Perú. Editora Normas Legales S.A.C.
Puecher, D.; Robredo, C.; Ríos, R.; Rimieri, P. 1998.
Evaluación de la variabilidad por RAPDs en Bromus catharticus Vahl. En: Congreso Argentino de Botánica. Río Cuarto.
Rodríguez, N. 1999.
Determinación de la tasa de Cruzamiento con Marcadores Bioquimicos
(Isoenzimas) en una Población de Zapote (Pouteria sapota (Jacq.) Moore Stearm) de la parte
baja del rio Hato, en San Agustín Acasaguastlan, El
Progreso. Tesis para obtener el Título de Ingeniero Agrónomo. Facultad de
Agronomía. Universidad de San Carlos de Guatemala.
Rodríguez-Rojas, T.;
Andrade-Rodríguez, M.; Alia-Tejacal, I.;
López-Martínez, V.; Espinosa-Zaragoza, S.; Esquinca-Avilés,
H. 2012. Caracterización molecular de zapote mamey (Pouteria
sapota (Jacq.) Moore & Stearn). Rev. Fac. Agron. (LUZ) 29: 339-354
Rolim, L.; Queiroz, M.; Fontes, A.; Cortopassi, G. 2011. Use of RAPD Molecular Markers on Differentiation of Brazilian and Chinese Ganoderma lucidum Strains. Braz. Arch. Biol. Technol. 54(2): 273-281.
Sánchez, A. 2017. Papa (Solanum
tuberosum L.) mediante marcadores SSRs Genetic variability study in accessions of
papa. Rev. Cien. Agri. 14(c): 67–76.
Sánchez-Coello, N.;
Luna-Rodríguez, M.; Vázquez-Torres, M.; Sánchez-Velásquez, L.; Santana-Buzzy, N.; Octavio-Aguilar, P., Iglesias-Andreu, L. 2012.
Optimización de un protocolo del aislamiento del ADN y de un sistema de
amplificación ISSR-PCR para Ceratozamia mexicana Brongn. (Zamiaceae). Revista
Chapingo serie ciencias forestales y del ambiente 18(1): 123-133.
Sambrook, J.; Fritsch, E.F.; Maniatis, T. 2001.Molecular Cloning: A Laboratory Manual. 3ª Ed.Spring Harbor LaboratoryPress,
Sanjinés, A.; Øllgaard,
B.; Balslev, H. 2006. Frutos
comestibles. Botánica Económica de los Andes Centrales. 329-346 pp.
Utrera, L.; Martínez, T. 1994. Caracterización in situ de zapote (Pouteria sapota (Jacq) Moore Stearn) en Chiquimulilla y Guazacapan, Santa Rosa, Guatemala. TIKALIA 7(2): 35-50.
Wu, W.; Chen, F.; Yeh, K.; Chen, J. 2019. ISSR analysis of genetic diversity and structure of plum varieties cultivated in Southern China. Biology 8(1): 1-13.