Caracterización
molecular de bacterias endofíticas de Elodea potamogeton (“llachu”) del
Lago Titicaca
Molecular
characterization of endophiytic bacteria of Elodea potamogeton
(“llachu”) from Titicaca Lake
Pedro Ubaldo Coila Añasco1,*;
Julio César Bernabé Ortiz2; Domingo Alberto Ruelas Calloapaza1
1 Universidad Nacional del
Altiplano – Puno, Av Floral 1153, Puno, Perú.
2 Universidad Nacional de
San Agustín Arequipa,
Santa Catalina 117, Arequipa, Perú.
*Autor
corresponsal: pcoila@unap.edu.pe (P. Coila).
ID
ORCID de los autores
P.
Coila:  https://orcid.org/0000-0002-5708-7464 J.
Bernabé:
https://orcid.org/0000-0002-5708-7464 J.
Bernabé:  https://orcid.org/0000-0003-0313-1033
https://orcid.org/0000-0003-0313-1033
D.
Ruelas:  https://orcid.org/0000-0003-0313-1033
https://orcid.org/0000-0003-0313-1033
RESUMEN
El objetivo fue aislar y caracterizar molecularmente
bacterias endofíticas del
“llachu”, planta acuática del Lago Titicaca que sobrevive en ambientes hipereutrofizados.
Para el aislamiento, las muestras fueron desinfectadas en alcohol 70%,
hipoclorito de sodio 0,5% y Tween 80%; luego, se homogenizaron y una alícuota sembrada en
medio Luria-Bertani (LB). Se eligieron tres cepas para su resembrado en agar LB
sólido. De las cepas puras, se extrajo DNA con la mezcla fenol-cloroformo-alcohol
isoamílico. Se amplificó el gen 16S rDNA por PCR; los amplicones se remitieron para su
secuenciamiento.
Las secuencias se editaron con BioEdit 7.0.0 y se eligieron secuencias
similares con BLASTn del GenBank, se hizo el alineamiento múltiple de secuencias
con Clustal W. Para las relaciones evolutivas se utilizó el MEGA7
construyéndose árboles filogenéticos por el método de Neighbor-Joining. El
análisis molecular demuestra que la cepa Sample10ped corresponde a Pantoea sp.
con una identidad superior al 76%; la cepa Sample11pe a Pseudomonas sp.
con un 100% de identidad; y, a la cepa Sample12ped a Raoultella terrigena sp.
o Klebsiella sp. con una identidad de 99%. Las secuencias de las dos
últimas, fueron registradas en el GenBank.
Palabras clave: Elodea; bacteria endofítica;
gen 16S rDNA; filogenia.
ABSTRACT
The aim was to isolate and
molecularly characterize endophytic bacteria from “llachu”, an aquatic plant
from Lake Titicaca that survives in hypereutrophic environments. For isolation,
the samples were disinfected in 70% alcohol, 0,5% sodium hypochlorite and 80%
Tween; then, they were homogenized, and an aliquot seeded in Luria-Bertani (LB)
medium. Three strains were chosen for reseeding on solid LB agar. From the pure
strains, DNA was extracted with the phenol-chloroform-isoamyl alcohol mixture.
The 16S rDNA gene was amplified by PCR; amplicons were submitted for
sequencing. The sequences were edited with BioEdit 7.0.0 and similar sequences
were chosen with BLASTn from GenBank, multiple sequence alignment was done with
Clustal W. For evolutionary relationships, MEGA7 was used, constructing
phylogenetic trees by the Neighbor-Joining method. Molecular analysis shows
that the Sample10ped strain corresponds to Pantoea sp. with an identity
greater than 76%; the Sample11pe strain to Pseudomonas sp. with 100%
identity; and, to the strain Sample12ped to Raoultella terrigena sp. or
Klebsiella sp. with an identity of 99%. The sequences of the last two were
registered in GenBank.
Keywords: Elodea;
endophytic bacteria; 16S rDNA gene; phylogeny.
Recibido: 30-06-2021.
Aceptado: 10-08-2021.
A casi 4 000 m de altitud,
el Lago Titicaca cumple un rol importante en la vida del poblador altoandino,
tanto ecológica como económicamente. Sin embargo, en los últimos tiempos, viene
sufriendo un acelerado proceso de contaminación y de eutrofización, por un mal
manejo de las aguas residuales y de residuos sólidos de las ciudades y pueblos
aledaños. Por ejemplo, Beltrán et al.
(2015),
al evaluar la calidad del agua de la bahía interior de la ciudad de Puno, a
través de determinaciones físico-químicas y microbiológicas (temperatura,
oxígeno disuelto, pH, fosfatos, nitratos, nitritos, transparencia, conductividad
eléctrica, alcalinidad y coliformes fecales) conclu-yeron que es una zona
crítica en contaminación, alcanzándose niveles hipereutróficos. Sin embargo, a
pesar de estas condiciones, muchas especies de plantas acuáticas sobreviven en
ese ambiente, siendo una de ella el “llachu” (Elodea potamogeton) una macrófita
que sirve de alimento para el ganado vacuno que se cría en todo el anillo
circunlacustre.
Es probable, que alguna
asociación simbiótica entre la planta y microorganismos rizosféricos y
endofíticos cumplan un rol importante en la vida de estas especies. Los
endofíticos (bacterias, hongos o actinomicetos) son microorganismos que
colonizan la planta, sin causar ningún signo o enfermedad aparente (Nair &
Padmavathy, 2014). Existen muchos estudios que demuestran las interacciones
simbióticas de las bacterias endofíticas con las plantas. Algunas bacterias
endofíticas promueven el crecimiento y proporcionan tolerancia al estrés
ambiental (Alvarado-Marchena
et al., 2016; Conn & Franco, 2004; Munif et al., 2012; Nair &
Padmavathy, 2014; Rosenblueth & Martínez-Romero, 2006; Sessitsch et al.,
2002; Wang et al., 2006); otras actúan como agentes de control biológico
para los fitopatógenos a través de sus metabolitos secundarios (de Oliveira
Costa et al., 2012); otras interactúan con los patógenos y aumentan la
resistencia de las plantas a las enfermedades mediante la producción de agentes
antifúngicos, antibacterianos, sideróforos o confiriendo inmunidad (Alvarado-Marchena
et al., 2016; Pérez & Chamorro, 2012), mientras que otras
aumentan la capacidad de fijación de nitrógeno y aceleran la germinación de
semillas y el crecimiento de plantas (Compant et al.,
2005; Iniguez et al., 2004; Li et al., 2008; Rosenblueth & Martínez-Romero,
2006).
En la actualidad, existe un
interés por caracterizar las bacterias endofíticas de diversas especies
vegetales con fines biotecnológicos; especialmente, en la biorremediación, en
la biofertilización y en el control biológico de los agentes patógenos de las
plantas, ya que el uso de fertilizantes químicos, pesticidas y otros, tienen un
impacto negativo sobre el medio ambiente y la salud humana y animal.
Pérez &
Chamorro (2012)
indican que el número de bacterias endofíticas depende de la especie
bacteriana, el genotipo y la etapa de desarrollo de la planta huésped, así como
las condiciones ambientales. En ese contexto, el presente estudio se diseñó con
el objetivo de aislar y caracterizar molecularmente a través de la
amplificación y análisis del gen 16S rDNA. algunas bacterias endofíticas de la
macrófita Elodea potamogeton (“llachu”) que desarrolla en aguas de la
bahía interior del Lago Titicaca, Puno, Perú.
Aislamiento,
purificación y conservación de las bacterias endofíticas
Las muestras de la planta Elodea
potamogeton, fueron colectadas en bolsas de polietileno estériles de las
orillas de la bahía interior del Lago Titicaca de la ciudad de Puno, ubicada a
3 825 m de altitud. El muestreo se realizó aleatoriamente, colectán-dose sólo
aquellas con aparente buen estado fitosanitario y sin signos de fitotoxicidad.
Las muestras fueron llevadas al laboratorio de Biología Molecular de la Escuela
Profesional de Biología de la Universidad Nacional de San Agustín, Arequipa.
En el laboratorio, la
planta fue sometida a un proceso de desinfección y esterilización superficial,
siguiendo el protocolo de Sakiyama et al.
(2001)
que consta de los siguientes pasos: 1) lavado con agua de caño y enjuague con
agua destilada por tres veces, 2) inmersión en alcohol 70% por un minuto y
enjuague con agua destilada dos veces, 3) inmersión en hipoclorito de sodio 5%
y agitación durante cinco minutos y enjuague con agua destilada, y 4) inmersión
en Tween 80% y agitación por tres minutos, para finalizar con un lavado con
agua destilada por cuatro veces.
Para el aislamiento de las
bacterias endófitas y la obtención de un cultivo puro se empleó el método de Vincent (1970). Primero, la
planta fue triturada en NaCl 0,9% hasta homogenización completa, luego
centrifugarla por cinco minutos. El sobrenadante se sembró en 10 mL de medio LB
(Luria-Bertani), para finalmente seleccionar las colonias y cultivarlas en
placa Petri con medio LB sólido a 37 °C por 24 horas.
Para el proceso de
purificación, las cepas obtenidas de los aislamientos se volvieron a cultivar
en caldo PY hasta la obtención de cepas puras. Los cultivos puros obtenidos se
conservación en viales con glicerol 15%, los que fueron congelados primero a
-20 °C y luego a -70 °C.
Caracterización molecular
de los aislados
Extracción, cuantificación
y evaluación de la calidad del DNA
La extracción de DNA de los
cultivos puros se realizó mediante la técnica descrita por Sambrook &
Russell (2006),
mediante la mezcla fenol-cloroformo-alcohol isoamílico (25:24:1), mientras que
la precipitación y purificación con alcohol isopropílico y etanol al 75% en
frio. La cuantificación de DNA se realizó por espectro-fotometría ultravioleta
(UV) a 260 nm de longitud de onda. La integridad del DNA ser comprobó mediante
electroforesis en gel de agarosa al 1% y la visualización en cámara
ultravioleta previo teñido del gel con bromuro de etidio.
Amplificación
del gen 16S rDNA
La amplificación del gen 16S rDNA de las bacterias
aisladas, se realizó mediante la técnica de reacción en cadena de la polmerasa
(PCR) utilizando los siguientes primers: 27F: 5′-AGAGTTTGATCATGGCTCAG-3′ (forward
primer) y 1492R: 5′-GGTTACCTTGTTACGACTT-3′ (reverse primer). Las
reacciones fueron realizadas en una mezcla que contenía: 2 µL de muestra
(DNA), 2 µL de primers y 29 µL del master mix conteniendo la Taq polimerasa,
MgCl2 y los dNTPs en buffer. Se utilizó el termociclador GeneAMP PCR
System bajo las siguientes condiciones de reacción: 1) desnaturalización
inicial y activación de la polimerasa a 95 °C/5 min, seguido de 30 ciclos de
desnaturalización (94 °C/1 min), anillamiento (60 °C/1 min) y extensión (72 °C/2
min); y, una extensión final a 72 °C/5 min. Los amplicones se purificaron con
reactivos Qiagen System ® y verificados por electroforesis en gel de agarosa al 1% en buffer TBE 1X.
La visualización se hizo en transiluminador ultravioleta previo teñido con
bromuro de etidio (0,5 µg/mL) (Fig.)
Secuenciamiento
Para el secuenciamiento, se
remitieron 20 µL de los productos amplificados al laboratorio Functional
Biosciences, Inc. (USA) que utiliza el método de Sanger en una plataforma Big
Dye V3.1 en instrumentos ABI 3730xl.
Análisis
bioinformático de las secuencias
Las secuencias del gen 16S
rDNA fueron editadas con el software BioEdit Versión 7.0.0© (Hall, 1999). Luego, se
buscaron secuencias similares en la base de datos del GenBanK del Centro
Nacional para Información Biotecnológica (NCBI) (http://ncbi.nlm.nih.gov/) utilizando su
herra-mienta BLASTn. El alineamiento múltiple de secuencias (AMS) entre las secuencias
que presentaron los más altos porcentajes de homología (coincidencias) del
GenBank y las secuencias obtenidas en el estudio, se realizó con el programa
Clustal W del software BioEdit version 7.0.0 ©. Finalmente, se realizó el
análisis filogenético utilizando el software MEGA7 (Molecular Evolutionary
Genetics Analysis, Version 7.0) (Kumar et al., 2016). Las distancias evolutivas se calcularon utilizando el método
de máxima verosimilitud compuesta (Tamura
et al., 2004) basada en las unidades del número de
sustituciones de bases por sitio. Para la reconstrucción filogenética se utilizó el
método de agrupamiento Neighbor-Joining con 1000 réplicas de bootstrap (Saitou &
Nei, 1987; Tamura et al., 2004). Dos de las secuencias obtenidas se registraron en la base de
datos del GenBank del National Center for Biotechnology Information (NCBI).
Se eligieron tres cultivos
puros de bacterias endofíticas, a las cuales se les denominó cepa 1 (Sample10ped 16sf), cepa 2
(Sample11ped 16sf) y cepa 3 (Sample13ped 16sf), nombres con los cuales fueron
enviados a secuenciar. Las secuencias obtenidas del gen 16S rDNA para cada una
de las cepas tuvieron una longitud de 1057, 1112 y 1115 nucleótidos, para las
cepas 1, 2 y 3, respecti-vamente.
Cepa 1
(Sample10ped 16sf)
Al compararse la secuencia del gen 16S rDNA de
esta cepaSample10 con la base de datos del GenBank, se encontraron siete
coincidencias cuyos porcentajes de similitud oscilan entre 76 y 80% con
bacterias del género Pantoea sp. (Tabla 1), lo cual indicaría que la
bacteria endofítica Sample10ped 16sf podría ser una especie de este
género.
Según la matriz de distancia genética entre las
accesiones, la especie más relacionada es con la accesión
KF202812.1_Pantoea_sp., con la cual tiene una divergencia de 0.
La Figura 1, muestra el árbol filogenético del gen
16S rDNA de la cepa Sample10ped 16sf con las siete bacterias relacionadas de la
base de datos del GenBank utilizando el método Neighbor-Joining, donde el
número en los nodos indica el valor del porcentaje bootstrap basada en 1000
réplicas y la escala indica el número de sustituciones por posición de los
nucleótidos.
Tabla 1
Secuencias que
producen alineaciones significativas con la cepa Sample10ped 16sf del GenBank
|
|
Accesiones/descripción
|
Max score
|
Total score
|
Query cover
|
E value
|
Ident
|
|
1
|
KF202734.1_Pantoea_sp.
|
163
|
163
|
56%
|
4e-36
|
78%
|
|
2
|
KF202775.1_Pantoea_sp.
|
178
|
178
|
51%
|
2e-40
|
80%
|
|
3
|
KF202784.1_Pantoea_sp.
|
159
|
159
|
50%
|
6e-35
|
79%
|
|
4
|
KF202790.1_Pantoea_sp.
|
154
|
154
|
45%
|
3e-33
|
80%
|
|
5
|
KF202791.1_Pantoea_sp.
|
189
|
189
|
68%
|
1e-37
|
76%
|
|
6
|
KF202807.1_Pantoea_sp.
|
161
|
161
|
51%
|
2e-35
|
79%
|
|
7
|
KF202812.1_Pantoea_sp.
|
161
|
161
|
51%
|
2e-35
|
79%
|
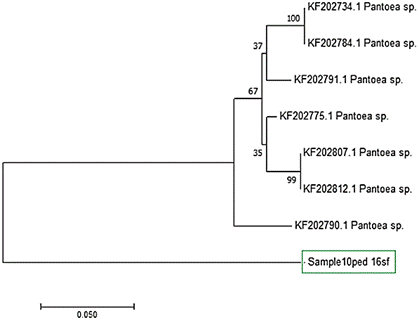
Figura 1. Árbol filogenético del
gen 16S rDNA de la cepa Sample10ped 16sf y de siete bacterias relacionadas de
la base de datos del GenBank usando el método Neighbor-Joining.
En este árbol se puede
observar que la cepa de estudio se encuentra en una rama distinta, lo que
demuestra una elevada variabilidad existente entre las cepas, indicando que se
trataría de una nueva especie. Cordero et al. (2008) y Li et al. (2008) , indican que las
bacterias endofíticas Pantoea sp., son solubilizadoras de fosfatos
incrementando la capacidad de las plantas de absorber el fósforo del agua. Como
se sabe, la mayor de fósforo se encuentra formando compuestos insolubles, poco
o nada utilizables o disponibles para las plantas, pero algunas especies
bacterianas como Pantoea sp., tienen la capacidad de solubilizar estos
compuestos insolubles de fosfatos inorgánicos como el tricálcico y el
dicálcico, la hidroxiapatita y las rocas fosfatadas en fósforo útil.
Lara-Cortés et al. (2019)
también aislaron Pantoea vagans de flores de dalia con fines de
florifagia e indican que las bacterias Pantoea son bacterias que se pueden
encontrar en el suelo, agua y plantas, así como formando parte de la microbiota
intestinal normal de muchos animales y del hombre; y, que potencialmente son patógenas pudiendo ocasionar
enfermedades gastrointestinales.
Cepa 2 (Sample11ped 16sf)
Las secuencias que producen alineaciones
significativas con la cepa Sample11ped del GenBank (100% de identidad)
corresponden a las del género
Pseudomonas sp.(Tabla 2) por lo que se infiere que la bacteria
endofítica aislada en el presente estudio pertenece a este género.
La matriz de distancias genéticas indica que la
cepa en estudio tiene una divergencia de 0 con las especies P. syringae,
P. Coronafaciens y Pseudomonas sp.
Se realizó un alineamiento
múltiple de secuencias (AMS) y la reconstrucción filogenética con las 12
secuencias obtenidas en la base de datos (Figura 2). El número en los nodos
indica el valor del porcentaje bootstrap basada en 1000 réplicas y la escala el
número de sustituciones por posición de los nucleótidos.

Figura 2. Árbol filogenético del
gen 16S rDNA de la cepa Sample11ped 16sf con las 12 bacterias relacionadas de
la base de datos del GenBank usando el método Neighbor-Joining.
Como se aprecia, la bacteria endofítica aislada en
el presente estudio (sample 11ped 16sf) quedó agrupada en una de las dos ramas
generadas en el árbol filogenético. La bacteria podría corresponder a las
especies P. syringae o P. coronafaciens, ya que con estas
especies tiene un 100% de homología.
La mayor parte de estudios indican que las Pseudomonas
son bacterias que habitan en el suelo y aguas estancadas y que pueden penetran
a las plantas por los estomas, heridas y otras aberturas produciendo
enfermedades (Montero, 2012). Sin embargo, el reciente
estudio de Devi et al. (2017), ha permitido identificar
una bacteria endofítica promotora de crecimiento de la planta Achyranthes
aspera L., designado como AL2-14B, el cual basados en sus características
fenotípicas y fisiológicas y análisis de la secuencia del gen 16S rDNA fue
identificado como Pseudomonas aeruginosa.
Tabla 2
Secuencias que
producen alineaciones significativas con la cepa Sample11ped 16sf del GenBank
|
|
Accesiones/descripción
|
Max score
|
Total score
|
Query cover
|
E value
|
Ident
|
|
1
|
KR922151.1_Pseudomonas_syringae
|
1672
|
1672
|
100%
|
0,0
|
100%
|
|
2
|
KR922077.1_Pseudomonas_sp.
|
1672
|
1672
|
100%
|
0,0
|
100%
|
|
3
|
MF591568.1_Pseudomonas_coronafaciens
|
1666
|
1666
|
100%
|
0,0
|
99%
|
|
4
|
MF669556.1_Pseudomonas_coronafaciens
|
1666
|
1666
|
99%
|
0,0
|
100%
|
|
5
|
MF668697.1_Pseudomonas_coronafaciens_pv.
|
1666
|
1666
|
100%
|
0,0
|
99%
|
|
6
|
LC164016.1_Pseudomonas_syringae
|
1666
|
1666
|
100%
|
0,0
|
99%
|
|
7
|
LC164015.1_Pseudomonas_syringae
|
1666
|
1666
|
100%
|
0,0
|
99%
|
|
8
|
KR922303.1_Pseudomonas_syringae
|
1666
|
1666
|
100%
|
0,0
|
99%
|
|
9
|
KR922143.1_Pseudomonas_syringae
|
1666
|
1666
|
100%
|
0,0
|
99%
|
|
10
|
KJ830937.1_Pseudomonas_syringae
|
1666
|
1666
|
100%
|
0,0
|
99%
|
|
11
|
JQ977255.1_Pseudomonas_sp.
|
1666
|
1666
|
100%
|
0,0
|
99%
|
|
12
|
KC311259.1_Pseudomonas_sp.
|
1666
|
1666
|
100%
|
0,0
|
99%
|
Este es el primer estudio en donde se indica que
la especie Pseudomonas es una bacteria endofítica. El mismo autor,
señala que la bacteria P. aeruginosa AL2-14B produce una buena cantidad
del sideróforo (IAA, ácido indol acético) y solubiliza fosfatos inorgánicos lo
que promueve el crecimiento y las propiedades antioxidantes de la planta
hospedera. Por lo tanto, la cepa aislada podría ser utilizado como biofertilizante
y, por su actividad antioxidante, tiene la ventaja adicional de ser utilizado
como una planta medicinal, lo cual también podría inferirse a la planta Elodea
potamogeton explorada en el presente estudio; es decir, podría ser
utilizado como biofertilizante y antioxidante.
Otros estudios han demostrado que las especies de Pseudomonas
son utilizadas para degradar detergentes en ecosistemas acuáticos, que son un
problema amenazante para la flora y microbiota acuática. Es el caso del estudio
de Guevara et al. (2013), quienes utilizaron a las
bacterias propias de aguas residuales para biodegradar detergentes,
encontrándose que las Pseudomonas son las que tienen la capacidad más
alta de utilizar los detergentes. La cepa en estudio fue registrada en el
GenBank bajo la accesión Nº SUB4288247
(cultured Prokaryotic 16S rRNA / Sample11ped).
Cepa 3 (Sample12ped 16sf)
Al realizar la búsqueda de secuencias que producen
alineaciones significativas en el GenBank, la cepa en estudio podría pertenecer
a los géneros Raoultella terrígena sp. o Klebsiella sp,
por los altos porcentajes de identidad con estas especies (99%) y un E-valor de
0 (Tabla 3).
La matriz de distancias genéticas muestra que la
cepa aislada tiene una divergencia entre 0,01 y 0,03 con las especies
indicadas.
Al realizar el alineamiento
múltiple de secuencias (AMS) y con las doce primeras secuencias del GenBank se
construyó el árbol filogenético mostrado en la Figura 3. El número en los nodos
indica el valor del porcentaje bootstrap basada en 1000 réplicas y la escala el
número de sustituciones por posición de los nucleótidos.
Brisse et al. (2006), indican que las Klebsiella
spp. se han recuperados de ambientes acuáticos contaminados y que reciben
residuos industriales y otros productos; además, señalan que son encon-trados
en una variedad de fuentes ambientales como suelo y plantas contribuyendo en
los procesos bioquímicos y geoquímicos. Arenas et al. (2009), indican que las bacterias
del género Klebsiella son organismos saprófitos, que crecen en zonas
contaminadas con herbicidas, sugiriendo que podrían tener una relación
simbiótica y benigna con su hospedero. Por lo tanto, la bacteria endofítica
aislada en el presente trabajo estaría cumpliendo un rol protector en la planta
que crece en aguas muy contaminadas, tal como lo es el “llachu”.
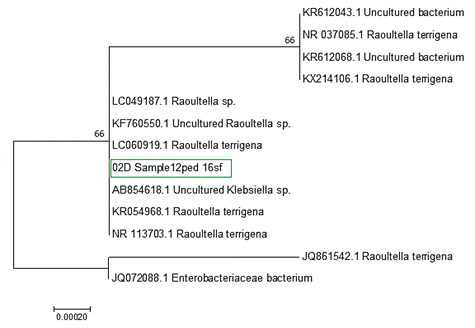
Figura 3. Árbol filogenético del
gen 16S rDNA de la cepa Sample12ped 16sf y de bacterias relacionadas de la base
de datos del GenBank usando el método Neighbor-Joining.
Schicklberger et al. (2015) aislaron la cepa R1Gly de Raoultella en medio
libre de nitrógeno y utilizando glicerol como única fuente de carbono,
encontrando que, además de los genes para la fijación biológica de nitrógeno,
la cepa aislada contenía todos los genes necesarios para la producción de
sustancias promotoras del crecimiento de la planta: ácido indol-acético,
2,3-butanodiol, entre otros.
Por otra parte, Luo et al. (2016) indican que las bacterias
fijadoras de N2 que pertenecen al género Raoultella de la
familia Enterobacteriaceae están ampliamente asociadas con las plantas.
Estos autores aislaron la cepa L03 de Raoultella sp. de las
raíces de caña de azúcar y determinaron la fijación de nitrógeno atmosférico. Los
resultados que encontraron fueron un incremento de la biomasa de la planta, del
nitrógeno total y de la clorofila, además de aliviar los signos de deficiencia
de nitrógeno de las plantas en condiciones de limitación de este elemento.
Tabla 2
Secuencias que
producen alineaciones significativas con la cepa Sample12ped 16sf del GenBank
|
|
Accesiones/descripción
|
Max score
|
Total score
|
Query cover
|
E value
|
Ident
|
|
1
|
LC060919.1_Raoultella_terrigena
|
1733
|
1733
|
100%
|
0,0
|
99%
|
|
2
|
AB854618.1_Uncultured_Klebsiella_sp.
|
1731
|
1731
|
100%
|
0,0
|
99%
|
|
3
|
KF760550.1_Uncultured_Raoultella_sp.
|
1731
|
1731
|
100%
|
0,0
|
99%
|
|
4
|
KR054968.1_Raoultella_terrigena
|
1729
|
1729
|
100%
|
0,0
|
99%
|
|
5
|
KX214106.1_Raoultella_terrigena
|
1727
|
1727
|
100%
|
0,0
|
99%
|
|
6
|
KR612068.1_Uncultured_bacterium
|
1727
|
1727
|
100%
|
0,0
|
99%
|
|
7
|
KR612043.1_Uncultured_bacterium
|
1727
|
1727
|
100%
|
0,0
|
99%
|
|
8
|
LC049187.1_Raoultella_sp.
|
1727
|
1727
|
100%
|
0,0
|
99%
|
|
9
|
JQ861542.1_Raoultella_terrigena
|
1727
|
1727
|
100%
|
0,0
|
99%
|
|
10
|
JQ072088.1_Enterobacteriaceae_bacterium
|
1727
|
1727
|
100%
|
0,0
|
99%
|
|
11
|
NR_113703.1_Raoultella_terrigena
|
1727
|
1727
|
100%
|
0,0
|
99%
|
|
12
|
NR_037085.1_Raoultella_terrigena
|
1727
|
1727
|
100%
|
0,0
|
99%
|
Este estudio, demostró por primera vez que una
bacteria Raoultella es capaz de fijar el nitrógeno atmosférico en
asociación con la planta hospedera, hecho que también estaría ocurriendo en la Elodea
potamogeton.
La cepa en estudio fue registrada en el GenBank
bajo la accesión Nº SUB4252655
(cultured Prokaryotic 16S rRNA).
De la planta acuática Elodea potamogeton
(“llachu”) proveniente de aguas eutrofizadas de la Bahía Interior de Lago
Titicaca se lograron aislar tres bacterias endofíticas, cuyo análisis molecular
a través del secuenciamiento del gen 16S rDNA, indican que pertenecen a los
siguientes géneros: cepa 1 (Sample10ped 16sf) a Pantoea sp.; cepa 2
(Sample11ped 16sf) a Pseudomonas sp. y cepa 3 (Sample12ped 16sf) a Raoultella terrígena sp. o Klebsiella sp.
Se registraron las secuencias de las cepas 11 y 12 en el GenBank del NCBI cuyas
accesiones son SUB4288247 y SUB4252655, respectivamente.
Al laboratorio “ADN
Uchumayo” de Arequipa, por habernos bridados sus instalaciones y equipos para
la extracción de ADN y amplificación del gen 16S rDNA por PCR,
REFERENCIAS
BIBLIOGRÁFICAS
Alvarado-Marchena,
L., Schmidt-Durán, A., Alvarado-Ulloa, C., Chacón-Cerdas, R., &
Flores-Mora, D. (2016). Molecular characterization of the endophytic
bacteria found in the fig crops (Ficus carica var . Brown Turkey) in Costa
Rica. ARPN Journal of Agricultural and Biological Science, 11(7),
290–297.
Arenas,
N. E., Gutiérrez, A. J., Salazar, L. M., Polanco, J. C., & Gómez, A.
(2009). Construcción de una filogenia molecular para las especies de los
géneros Klebsiella y Raoultella basada en los genes ARNr 16S y ARN polimerasa
subunidad. Revista Ciencias de La Salud, 7(2), 22–29.
Beltrán,
D. F., Palomino, R. P., Moreno, E. G., Peralta, C. G., & Montesinos-Tubée,
D. B. (2015). Calidad de agua de la bahía interior de Puno, lago Titicaca
durante el verano del 2011. Revista Peruana de Biología. Biol, 22(3),
335–340.
Brisse,
S., Grimont, F., & Grimont, P. (2006). The genus Klebsiella. In The
Prokaryotes (Vol. 6, pp. 159–196).
Compant, S., Reiter, B., Sessitsch, A., Nowak, J.,
Clément, C., & Barka, E. (2005). Endophytic Colonization of Vitis vinifera
L. by Plant Growth-Promoting Bacterium Burkholderia sp. Strain PsJN. Appl.
Environ. Microbiol., 71(4), 1685–1693.
Conn, V. M., & Franco, C. M. M. (2004).
Analysis of the Endophytic Actinobacterial Population in the Roots of Wheat
(Triticum aestivum L.) by Terminal Restriction Fragment Length Polymorphism and
Sequencing of 16S rRNA Clones. Appl. Environ. Microbiol., 70(3), 1787–1794.
Cordero,
J., Ortega-Rodes, P., & Ortega, E. (2008). La inoculación de plantas con
Pantoea sp., bacteria solubilizadora de fosfatos, incrementa la concentracion
de P en los tejidos foliares. Rev. Colomb. Biotecnol, X(1), 111–121.
de Oliveira Costa, L. E. E. de O., de Queiroz, M.
V., Borges, A. C., de Moraes, C. A., & de Araújo, E. F. (2012). Isolation and
characterization of endophytic bacteria isolated from the leaves of the common
bean (Phaseolus vulgaris). Brazilian Journal of Microbiology, 43(4),
1562–1575.
Devi, K. A., Pandey, G., Rawat, A. K. S., Sharma,
G. D., & Pandey, P. (2017). The endophytic symbiont-Pseudomonas aeruginosa
stimulates the antioxidant activity and growth of Achy-ranthes aspera L. Frontiers
in Microbiology, 8(SEP), 1–14.
Guevara,
J., Castañeda, I., Juárez, J., & Mendoza, A. (2013). Efecto de la
temperatura y del pH sobre el crecimiento de Pseudomonas aeruginosa MBLA-04 en
solución mínima de sales con detergente Ace. Revista Rebiol, 33(1),
1–8.
Hall, T. A. (1999). BioEdit: A User-Friendly
Biological Sequence Alignment Editor and Analysis Program for Windows 95/98/NT.
Nucleic Acids Symposium Series, 41, 95–98.
Iniguez, A. L., Dong, Y., & Triplett, E. W.
(2004). Nitrogen fixation in wheat provided by Klebsiella pneumoniae 342. Molecular
Plant-Microbe Interactions, 17(10), 1078–1085.
Kumar, S., Stecher, G., & Tamura, K. (2016).
MEGA7: Molecular Evolutionary Genetics Analysis version 7.0 for bigger datasets
Sudhir. Mol
Biol Evol.,
33(7), 1870‐1874.
Lara-Cortés,
E., Bautista- Baños, S., Barrera-Necha, L. L., Hernández-Zárate, G., &
León- Rodríguez, R. (2019). Detección e identificación molecular de Pantoea
vagans en flores de Dahlia sp. TIP Revista Especializada En Ciencias
Químico-Biológicas, 22, 1–8.
Li, J. H., Wang, E. T., Chen, W. F., & Chen,
W. X. (2008). Genetic
diversity and potential for promotion of plant growth detected in nodule
endophytic bacteria of soybean grown in Heilongjiang province of China. Soil
Biology and Biochemistry, 40(1), 238–246.
Luo, T., Ou-Yang, X. Q., Yang, L. T., Li, Y. R.,
Song, X. P., Zhang, G. M., Gao, Y. J., Duan, W. X., & An, Q. (2016).
Raoultella sp. strain L03 fixes N2 in association with micropropagated
sugarcane plants. Journal
of Basic Microbiology,
56, 1–7.
Montero,
M. M. (2012). Pseudomonas aeruginosa multiresistente: aspectos
epidemiológicos, clínicos y terapéuticos [Universitat Autónoma de
Barcelona].
Munif, A., Hallmann, J., & Sikora, R. (2012).
Isolation of Endophytic Bacteria from Tomato and Their Biocontrol Activities
against Fungal Diseases Bacteria from Tomato and Their Biocontrol Activities
against Fungal Diseases. Microbiology Indonesia, 6(4), 148–156.
Nair, D. N., & Padmavathy, S. (2014). Impact
of endophytic microorganisms on plants, environment and humans. The Scientific
World Journal, 2014(March).
Pérez, A., & Chamorro, L. (2012). Bacterias endófitas: una
alternativa biologica para el control de burkholderia glumae en el cultivo del
arroz en colombia. Revista Colombiana de Ciencia Animal -, 4(1),
172–184.
Rosenblueth, M., & Martínez-Romero, E. (2006).
Bacterial
endophytes and their interactions with hosts. Molecular Plant-Microbe
Interactions, 19(8), 827–837.
Saitou, N., & Nei, M. (1987). The
neighbor-joining method: a new method for reconstructing phylogenetic trees. Molecular
Biology and Evolution, 4(4), 406–425.
Sakiyama, C. C. H., Paula, E. M., Pereira, P. C.,
Borges, A. C., & Silva, D. O. (2001). Characterization of pectin lyase
produced by an endophytic strain isolated from coffee cherries. Letters in
Applied Microbiology, 33(2), 117–121.
Sambrook, J., & Russell, D. W. (2006).
Purification of Nucleic Acids by Extraction with Phenol:Chloroform. Cold
Spring Harbor Protocols, 2006(1), pdb.prot4455.
Schicklberger, M., Shapiro, N., Loqué, D., Woyke,
T., & Chakraborty, R. (2015). Draft genome sequence of Raoultella terrigena
R1Gly, a diazotrophic endophyte. Genome Announcements, 3(3), 1–2.
Sessitsch, A., Howieson, J. G., Perret, X.,
Antoun, H., & Martínez, E. (2002). Advances in Rhizobium Research. Critical
Reviews in Plant Sciences, 21(4), 323–378.
Tamura, K., Nei, M., & Kumar, S. (2004). Prospects
for inferring very large phylogenies by using the neighbor-joining method. Proceedings
of the National Academy of Sciences of the United States of America, 101(30),
11030–11035.
Vincent, J. M. (1970). A Manual for the Practical
Study of the Root-Nodule Bacteria. In IBP Handbook No. 15, Blackwell
Scientific Publishers, Oxford. Wiley.
Wang, E. T., Tan, Z. Y., Guo, X. W.,
Rodríguez-Duran, R., Boll, G., & Martínez-Romero, E. (2006). Diverse
endophytic bacteria isolated from a leguminous tree Conzattia multiflora grown
in Mexico. Archives of Microbiology, 186(4), 251–259.
![]() https://orcid.org/0000-0002-5708-7464 J.
Bernabé:
https://orcid.org/0000-0002-5708-7464 J.
Bernabé: ![]() https://orcid.org/0000-0003-0313-1033
https://orcid.org/0000-0003-0313-1033 ![]() https://orcid.org/0000-0003-0313-1033
https://orcid.org/0000-0003-0313-1033 

