Bacterias
productoras de hidrolasas aisladas de suelos de cultivos de arroz:
Caracterización fenotípica y molecular
Hydrolase-producing
bacteria isolated from rice crop soils: Phenotypic and molecular
characterization
Manuel Vilchez-Chávez; Juan C. Flores-Santos;
Susana Calderón-Toledo; Julio Palma-Villanueva; Amparo Iris Zavaleta
Laboratorio
de Biología Molecular, Facultad de Farmacia y Bioquímica, Universidad Nacional
Mayor de San Marcos, Jr. Puno 1002, Lima, Perú.
* Autor
corresponsal: susana.calderon2@unmsm.edu.pe (S. Calderón-Toledo).
ORCID
de los autores:
M.
Vilchez-Chávez: http://orcid.org/0009-0001-2779-634X J. C. Flores-Santos: http://orcid.org/0000-0001-5632-7203
S.
Calderón-Toledo: http://orcid.org/0000-0001-7401-3291 J. Palma-Villanueva: http://orcid.org/0009-0001-1684-2558
A.
I. Zavaleta: http://orcid.org/0000-0003-3844-7185
RESUMEN
El objetivo de la presente investigación fue
aislar y caracterizar bacterias provenientes de suelos de cultivos de arroz
para determinar su potencial biotecnológico prospectivo. Para ello, se
recolectaron suelos de cultivos de arrozales del distrito de Rioja, Región de
San Martín, aislaron siete bacterias morfológicamente diferentes y determinaron
sus características fisiológicas, sensibilidad antimicrobiana y capacidad de
degradar diferentes sustratos. Las bacterias crecieron óptimamente entre 20 y
37 °C, a pH entre 5,0 y 10,0. Respecto a los perfiles de sensibilidad
antimicrobiana; una, tres, una, una y una cepa presentaron resistencia a 11,
12, 8, 7 y 4 antimicrobianos, respectivamente. La identificación molecular se
realizó mediante la amplificación y secuenciación de los genes ribosómicos 16S,
obteniendo similitud con los géneros Acinetobacter, Bacillus, Staphylococcus,
Comamonas y Aeromonas. Las bacterias, según su actividad
hidrolítica, formaron cinco grupos: tres degradaron gelatina, leche descremada,
caseína, aceite de oliva, almidón, celulosa, pectina, y tributirina; una,
aceite de oliva y tributirina; otra, gelatina, leche descremada, aceite de
oliva y tributirina; una, gelatina, aceite de oliva, almidón, tributirina; y
una, gelatina, leche descremada, caseína, aceite de oliva y tributirina. Las
bacterias aisladas presentan gran potencial como inoculantes para el
enriquecimiento y la biorremediación de suelos agrícolas.
Palabras
clave:
cultivos
de arroz; suelos agrícolas; hidrolasas; caracterización bioquímica; identificación
molecular.
ABSTRACT
The aim of this research
was to isolate and characterize bacteria from rice crop soils to determine
their prospective biotechnological potential. Therefore, seven morphologically
different bacteria were isolated and their physiological characteristics,
antimicrobial sensitivity and ability to degrade different substrates were
determined. The bacteria grew optimally between 20 and 37 °C, at pH between 5.0
and 10.0. Regarding antimicrobial sensitivity profiles; one, three, one, one,
one and one strain showed resistance to 11, 12, 8, 7 and 4 antimicrobials,
respectively. Molecular identification was performed by amplification and
sequencing of the 16S ribosomal genes, obtaining similarity with the genera Acinetobacter,
Bacillus, Staphylococcus, Comamonas and Aeromonas.
The bacteria, according to their hydrolytic activity, formed five groups: three
degraded gelatin, skim milk, casein, olive oil, starch, cellulose, pectin, and
tributyrin; one, olive oil and tributyrin; one, gelatin, skim milk, olive oil
and tributyrin; one, gelatin, olive oil, starch, tributyrin; and one, gelatin,
skim milk, casein, olive oil and tributyrin. The isolated bacteria show great
potential as inoculants for the enrichment and bioremediation of agricultural
soils.
Keywords: rice crops;
agricultural soils; hydrolases; biochemical characterization; molecular
identification.
Recibido: 18-12-2023.
Aceptado: 15-04-2024.

En las últimas
décadas se ha estimado que la generación de residuos sólidos a nivel mundial
asciende a más de 30 millones de toneladas diarias y en Perú supera las 14 mil,
de las cuales cerca del 50% se eliminan en botaderos, ríos, playas, entre
otros, agravando los problemas ambientales (MINAM, 2021).
Del total de
residuos sólidos generados en Perú, el 50% son de tipo orgánico con gran
potencial de reúso pues su bajo costo como materia prima es una opción
estratégica para incorporarlo nueva-mente en los procesos productivos
(García-Bucio et al., 2014; MINAM & MEF, 2021). Sin embargo, las
características recalcitrantes de algunos de estos residuos, requiere
tratamientos previos, encare-ciendo los costos y limitando su aprovechamiento
como materia prima (Kurniawan et al., 2006). En este contexto, se precisa la
búsqueda de nuevas tecnologías que solucionen eficazmente esta problemática,
donde la producción de enzimas hidrolíticas es una alternativa factible y
económica para su tratamiento (Parmar et al., 2001).
Tal motivo ha
conllevado a la selección de microorganismos con capacidad de hidrolizar sustratos
agrestes, de los cuales se ha identificado cepas de Pseudomonas, Proteus,
Acinetobacter, Serratia, Bacillus y Staphylococcus
productoras de diversas enzimas como proteasas, lipasas, amilasas, entre otras,
que facilitan altos contenidos de nitrógeno y carbono para inducir la proli-feración
de comunidades microbianas (Butt et al., 2021; Ganbat et al., 2022; Koul et
al., 2021; Mushtaq et al., 2022).
Por
consiguiente, la gran diversidad de suelos de cultivo en Perú representa una
oportunidad para el aislamiento de diversos microrganismos produc-tores de
hidrolasas. En el Perú y el mundo, uno de los cultivos de mayor importancia
económica por su producción y consumo, es el arroz. Así, a nivel mundial el
2020, la superficie cultivada de este cereal fue de 164,2 millones de ha y en
Perú de 417 mil ha, siendo el departamento de San Martin el productor de arroz
más importante (MINAGRI, 2021).
Los terrenos
de cultivo son sometidos a plaguicidas químicos para mejorar su rendimiento, lo
cual conlleva a la adaptación de los microorganismos a la producción de
diversas hidrolasas como parte de su mecanismo evolutivo. En este contexto, los
estudios relacionados con la microbiota de estos suelos de cultivo son escasos,
es por ello que se seleccionaron los suelos de cultivo de arroz del distrito de
Rioja – Región de San Martín con el objetivo de aislar y caracterizar bacterias
productoras de hidrolasas para su uso prospectivo en el control de suelos.
Aislamiento de
las bacterias. Se recolectaron muestras de suelos de cultivo arroz, en el
distrito de Rioja, provincia de Rioja, departamento de San Martín en Perú
durante la temporada de postcosecha (enero 2019), en frascos estériles, los
cuales fueron transportados a 4 °C en una caja térmica al Laboratorio de
Biología Molecular de la Facultad de Farmacia y Bioquímica de la Universidad
Nacional Mayor de San Marcos. Después, las muestras fueron pre enriquecidas en
caldo tripticasa de soya (TSB) Oxoid ThermoFisher - Scientific a 30 °C por 48
h. Seguidamente, se sembraron por extensión en agar tripticasa de soya (TSA) Oxoid
ThermoFisher - Scientific e incubaron a 37 °C por 24 h. Todas las colonias con
caracte-rísticas diferentes de morfología, tamaño, consis-tencia y coloración
fueron seleccionadas.
Caracterización
fisiológica. El crecimiento celu-lar se evaluó en medio TSB a distintas
concen-traciones de NaCl Sigma-Aldrich (0,5; 5,0; 10,0; 15,0 y 20,0%), pH (4,
5, 6, 7, 8, 9 y 10) y temperatura (4, 20, 37 y 45 °C) durante 24 h. Los aislados
se incubaron a 37 °C para las pruebas de salinidad y pH (Canales et al., 2014).
Pruebas
bioquímicas y metabolismo de azúcares. La producción de catalasa
se determinó con la adición de peróxido de hidrógeno 3% v/v. Las pruebas de
motilidad, indol, H2S, metabolismo de citrato y azúcares (arabinosa,
manosa, galactosa, celobiosa, maltosa, sorbitol, fructuosa, manitol, glucosa,
xilosa, sacarosa y glicerol, Sigma-Aldrich) con o sin producción de gas se
realizaron en medios sólidos y líquidos (Canales et al., 2014).
Sensibilidad
antimicrobiana. La prueba de difusión en disco descrita por Bauer et al.
(1966) fue utilizada con algunas modificaciones. Se aplicaron antibióticos en
base a sus diferentes mecanismos de acción y especificidad. Así, se prepararon
suspensiones bacterianas a 0,5 en la escala de McFarland, que fueron inoculadas
en agar Mueller Hinton Sigma-Aldrich. Se utilizaron: clindamicina 2 µg,
vancomicina 30 µg, cefoxitina 30 µg, eritromicina 15 µg, tobramicina 10 µg,
cloran-fenicol 30 µg, gentamicina 10 µg, novobiocina 5 µg, penicilina G 10U,
estreptomicina 10 µg, kanamicina 10 µg y ampicilina 10 µg. Luego, se incubaron
a 37 °C por 24 h y midieron los halos de inhibición. Los resultados fueron
reportados como sensible (S) o resistente (R) (Clinical and Laboratory
Standards Institute, 2018).
Actividad
hidrolítica. Se determinó sobre medios sólidos enriquecidos con diferentes
sustratos, que fueron inoculados por punción e incubados a 37 °C por 48 h para
la actividad proteolítica y 72 h para la pectinolítica, celulolítica,
amilolítica y lipolítica. La actividad proteolítica fue evaluada sobre agar
gelatina, caseína y leche descremada Sigma-Aldrich 1%. La hidrólisis en medio
gelatina se reveló con ácido tricloroacético 10%, la formación de halos traslúcidos
indicó positividad. Para la hidrólisis de ácidos grasos de cadena corta,
almidón, pectina, celulosa y ADN se usó tributirina 1%, almidón 2%, pectina de
manzana 0,5%, carboximetilcelulosa (CMC) 0,3% y medio ADN Sigma-Aldrich,
respecti-vamente. Los halos de hidrólisis se determinaron con la adición de
lugol, rojo de Congo 0,1%, bromuro de hexadeciltrimetilamonio 2% y HCl 1 M para
los medios suplementados con almidón, carboximetilcelulosa, pectina y ADN
(Canales et al., 2014).
Para la
hidrólisis de ácidos grasos de cadena larga se utilizó aceite de oliva 2,5%
(marca Filippo Berio, extra virgen y de 0,5% acidez) y 0,1 mg/l rodamina B. La
fluorescencia se observó por luz UV a 302 nm (Becerra & Horna, 2016).
Identificación
molecular y análisis filogenético. La extracción de ADN se
realizó siguiendo la metodología descrita por Calderón-Toledo et al. (2021).
La integridad
del ADN fue determinada mediante electroforesis en gel de agarosa 1%. La
amplificación y la secuenciación de los genes ribosómicos 16S se realizó
mediante el uso de cebadores específicos 16SF y 16SR en un termociclador SimpliAmp-
ThermoFisher Scientific (Canales et al., 2014).
Los nucleotidogramas
fueron alineados por Bioedit 7.0.5.3 y Cap3, y las secuencias nucleotídicas
fueron identificadas usando el algoritmo BLAST. Se realizó el análisis
filogenético mediante el alineamiento múltiple de los genes ribosómicos 16S,
luego de cual se utilizó el algoritmo de Neighbor-joining a fin de graficar un
árbol filogenético utilizando el programa Molecular Evolutionary Genetics
Analysis (MEGA).
Aislamiento
y caracterización fisiológica
En Perú, la Región de
San Martín tiene más de 100 000 ha con cultivos de arroz, ubicadas en la selva
alta a 820 m de elevación (MINAGRI, 2017). La sostenibilidad de estos
ecosistemas se basa en la gran diversidad microbiana que promueve la
composición orgánica de los suelos, conductividad eléctrica, pH, parámetros
ligados a la respiración, mineralización de la materia orgánica y el ciclo de
nutrientes en los suelos (Sritongon et al., 2022). Así, en esta investigación
se aislaron siete bacterias morfológicamente diferentes (Tabla 1).
Las bacterias aisladas
fueron caracterizadas como mesófilas debido a su crecimiento óptimo entre 20 y
37 °C. A estas temperaturas, el crecimiento del consorcio microbiano presenta
ventajas para su actividad conjunta. En este aspecto, en San Martín predomina
un clima cálido y húmedo entre 19 y 31 °C, donde los suelos bajo 15 cm de
profundidad están entre 27 y 31 °C, debido a la capacidad de absorber la
radiación solar, propiedades térmicas del suelo, porosidad, contenido de agua y
nivel de actividad microbiana, lo cual conlleva una mayor temperatura comparada
con la del suelo superficial (Puertas, 2009; Moraga, 2021). Así, las cepas MA3,
MA9 y MA12 crecieron hasta 45 °C.
Todas las cepas
mostraron crecimiento en el rango de pH entre 5,0 y 10,0, cercano al exigido
para el cultivo de arroz (pH 5,5 a 7,3). Además, el pH del suelo se modifica
por las inundaciones, en consecuencia, se promueve la adaptabilidad bacteriana
(Sanjinez, 2019). De esta forma, la acidificación de los suelos conlleva al
incremento de Actinobacterias, Firmicutes y Saccharibacteria, que tienen mayor
abundancia a pH entre 4,0 y 5,0 (32,94%) comparado a los de pH 6,0 y 7,0
(27,82%), lo cual explicaría los mecanismos de adaptabilidad a suelos
acidificados (Wang et al., 2022). Por ejemplo, las cepas MA2, MA3, MA9 y MA12
crecieron a pH 4.0.
En cuanto a la
halotolerancia, todas las cepas presentaron buen crecimiento a NaCl 0,5% la
cual fue determinada mediante espectrofotometría UV-Visible. Sin embargo, las
cepas MA3, MA9 y MA12 crecieron hasta 10% NaCl y MA2 hasta 20% (Tabla 1). En
este aspecto, la producción de clorofila, prolina y fenol en Oryza sativa
sería un mecanismo para mitigar el estrés salino cuando se inoculan bacterias
tolerantes a NaCl 12%, lo cual mejoraría la produc-tividad agrícola en suelos
salinos (Prittesh et al., 2020).
Caracterización
bioquímica y metabolismo de azúcares
Las bacterias aisladas
tuvieron actividad catalasa variada, esta contribuye en la conversión de un-trientes
debido a su capacidad de óxido-reducción e incrementa la fertilidad del suelo.
Por el contrario, todas las bacterias fueron indol y H2S negativo.
Asimismo, las cepas MA3, MA4, MA6, MA9, MA10 y MA12 presentaron motilidad, lo
que les permite adaptarse a las condiciones ambientales mediante el movimiento
hacia los nutrientes y evitar los tóxicos (Fei et al., 2020). Con referencia al
metabolismo de azúcares, las cepas MA1 y MA4 no fermentaron ninguno, mientras
que MA6 produjo gas con algunos de éstos (Tabla 1).
Sensibilidad
antimicrobiana
En relación con la
susceptibilidad antimicrobiana, el uso y la persistencia de pesticidas en los
campos de cultivo puede contribuir al incremento de resistencia antimicrobiana,
esto se evidenció en cepas de Azotobacter con resistencia múltiple
(Shahid & Khan, 2022).
La presencia de antimicrobianos
en suelos altera la actividad enzimática y el metabolismo de fuentes de
carbono, lo cual tiene impacto en la diversidad microbiana (Cycoń et al.,
2019). Así, las cepas MA3, MA9 y MA12 evidenciaron sensibilidad a todos los
antimicrobianos, por lo cual se identificaron cinco perfiles (Tabla 2). Todas
las cepas tienen sensibilidad a cefoxitina, eritromicina y cloranfenicol, esto
indicaría que las bacterias ambientales no poseen los mecanismos de resistencia
que si presentan las cepas clínicas. Entre los géneros que degradan
ciprofloxacino, cloranfenicol y norfloxacino, se describen a Acinetobacter,
Klebsiella y Bacillus (Cycoń et al., 2019).
Tabla
1
Características
fenotípicas de bacterias aisladas de suelos de cultivo de arroz del distrito de
Rioja
|
Características
|
Aislados
bacterianos
|
|
MA1
|
MA2
|
MA3
|
MA4
|
MA6
|
MA9
|
MA12
|
|
Morfología
celular
|
Bacilo
|
Coco
|
Bacilo
|
Bacilo
|
Bacilo
|
Bacilo
|
Bacilo
|
|
Tinción
Gram
|
-
|
+
|
+
|
-
|
-
|
+
|
+
|
|
Forma
de colonia
|
Redonda
|
Redonda
|
Redonda
|
Redonda
|
Redonda
|
Redonda
|
Redonda
|
|
Aspecto
de la colonia
|
Mediana de
bordes regulares
|
Pequeña de
bordes regulares
|
Mediana,
seca de bordes regulares
|
Mediana de
bordes regulares
|
Mediana,
mucosa de bordes regulares
|
Grande de
bordes irregulares
|
Mediana,
traslúcida mucosa de bordes regulares
|
|
Catalasa
|
+/-
|
+
|
+
|
+/-
|
+
|
+
|
+
|
|
Citrato
|
-
|
+
|
+
|
+
|
-
|
-
|
+
|
|
Motilidad
|
-
|
-
|
+
|
+
|
+
|
+
|
+
|
|
Temperatura
(°C)
|
20 - 37
|
20 - 37
|
20 - 45
|
20 - 37
|
20 - 37
|
20 - 45
|
20 - 45
|
|
pH
|
5-10
|
4-10
|
4-10
|
5-10
|
5-10
|
4-10
|
4-10
|
|
NaCl
(% p/v)
|
0,5 - 5
|
0,5 - 20
|
0,5 - 10
|
0,5
|
0,5
|
0,5 - 10
|
0,5 - 10
|
|
Fermentación
de azúcares
|
Arabinosa
|
NF/NG
|
NF/NG
|
NF/NG
|
NF/NG
|
F/NG
|
F/NG
|
NF/NG
|
|
Manosa
|
NF/NG
|
NF/NG
|
F/NG
|
NF/NG
|
F/G
|
F/NG
|
F/NG
|
|
Galactosa
|
NF/NG
|
F/NG
|
NF/NG
|
NF/NG
|
F/NG
|
NF/NG
|
NF/NG
|
|
Celobiosa
|
NF/NG
|
NF/NG
|
F/NG
|
NF/NG
|
NF/NG
|
F/NG
|
F/NG
|
|
Maltosa
|
NF/NG
|
F/NG
|
NF/NG
|
NF/NG
|
F/G
|
F/NG
|
F/NG
|
|
Sorbitol
|
NF/NG
|
NF/NG
|
NF/NG
|
NF/NG
|
NF/NG
|
NF/NG
|
NF/NG
|
|
Fructuosa
|
NF/NG
|
F/NG
|
F/NG
|
NF/NG
|
F/G
|
F/NG
|
F/NG
|
|
Manitol
|
NF/NG
|
NF/NG
|
F/NG
|
NF/NG
|
F/G
|
NF/NG
|
F/NG
|
|
Glucosa
|
NF/NG
|
F/NG
|
F/NG
|
NF/NG
|
F/G
|
F/NG
|
F/NG
|
|
Xilosa
|
NF/NG
|
NF/NG
|
NF/NG
|
NF/NG
|
NF/NG
|
NF/NG
|
NF/NG
|
|
Sacarosa
|
NF/NG
|
F/NG
|
F/NG
|
NF/NG
|
F/G
|
F/NG
|
F/NG
|
|
Glicerol
|
NF/NG
|
NF/NG
|
F/NG
|
NF/NG
|
F/NG
|
F/NG
|
F/NG
|
+, positivo; +/-, intermedio; (-), negativo;
NF, negativo para la fermentación del azúcar; NG, negativo para la producción
de gas; F, positivo para la fermentación del azúcar; G, positivo para la
producción de gas.
Tabla 2
Perfiles
de sensibilidad antimicrobiana de bacterias aisladas de suelos de cultivo de
arroz del distrito de Rioja
|
Cepa
|
AMP
|
FOX
|
DA
|
C
|
E
|
S
|
CN
|
K
|
NV
|
P
|
TOB
|
VA
|
|
MA1
|
S
|
S
|
-
|
S
|
S
|
S
|
S
|
S
|
-
|
-
|
S
|
-
|
|
MA2
|
S
|
S
|
S
|
S
|
S
|
S
|
S
|
R
|
S
|
S
|
S
|
S
|
|
MA3
|
S
|
S
|
S
|
S
|
S
|
S
|
S
|
S
|
S
|
S
|
S
|
S
|
|
MA4
|
R
|
S
|
-
|
S
|
S
|
R
|
R
|
S
|
-
|
-
|
R
|
-
|
|
MA6
|
R
|
S
|
-
|
S
|
S
|
S
|
S
|
S
|
-
|
-
|
S
|
-
|
|
MA9
|
S
|
S
|
S
|
S
|
S
|
S
|
S
|
S
|
S
|
S
|
S
|
S
|
|
MA12
|
S
|
S
|
S
|
S
|
S
|
S
|
S
|
S
|
S
|
S
|
S
|
S
|
S,
sensible; R, resistente; -, no aplica; AMP, ampicilina; FOX, cefoxitina; DA,
clindamicina; C, cloranfenicol; E, eritromicina; S, estreptomicina; CN,
gentamicina; K, kanamicina; NV, novobiocina; P, penicilina; TOB, tobramicina;
VA, vancomicina.
Actividad hidrolítica
Las cepas MA3, MA9 y MA12 hidrolizaron las
macromoléculas evaluadas (Tabla 3). A la vez, todas las bacterias degradan
tributirina y aceite de oliva, lo cual demuestra la adaptabilidad metabólica de
estas bacterias a medios con presencia de ácidos grasos. En este contexto, se
han descrito bacterias Gram positivas que hidrolizan tributirina a partir de
arrozales de la India y en suelo del páramo colombiano (Palacios et al., 2017;
Mithilesh et al., 2017). La cepa MA2 presentó un mayor grado de hidrólisis de
los ácidos grasos de cadena corta. Por otro lado, el 85,7% de los aislados
presentaron hidrólisis de un sustrato proteico. La actividad proteolítica se ha
descrito en aislados de rizósferas de cultivo de arroz en Cuba, donde su
inoculación en estas plantas se correlacionó con el incremento de la longitud
de la raíz (Hernández-Forte et al., 2022). De igual forma, la presencia de paja
de arroz en los suelos de cultivo incrementaría la actividad celulasa, ya que
estos serían degradados a celulosa y hemicelulosa, para luego transformarse en
carbohidratos, almidones y polisacáridos, y finalmente en monosacáridos (Tang
et al., 2022). En el presente estudio el 42,8% de las cepas tuvieron actividad
celulolítica, mientras que Balasjin et al. (2022) describen que el 84% de
bacterias de las subespecies japónica e indica poseen esta actividad. La
actividad pectinolítica y celulotítica se ha asociado a la tolerancia hasta
NaCl 8% en cultivos de arroz de Corea del Sur, comparado a las cepas MA3, MA9 y
MA12, que mostraron tolerancia hasta 10% (Walitang et al., 2017). La presencia
de varias enzimas hidrolíticas como amilasas, lipasas y gelatinasas ha sido
descrita por Roy et al. (2020) en arrozales de Purba Bardhaman (India), lo que
coincide con los aislados del presente estudio.
Tabla 3
Grado de hidrólisis de macromoléculas de
bacterias aisladas de suelos de cultivo de arroz del distrito de Rioja
|
Cepa
|
Gelatina
|
Leche
descremada
|
Caseína
|
Aceite
de oliva
|
Tributirina
|
Celulosa
|
Almidón
|
Pectina
|
|
MA1
|
0,00
|
0,00
|
0,00
|
1,75±0,06
|
3,00±0,00
|
0,00
|
0,00
|
0,00
|
|
MA2
|
2,25±0,35
|
1,50±0,00
|
0,00
|
1,12±0,15
|
10,75±0,35
|
0,00
|
0,00
|
0,00
|
|
MA3
|
1,40±0,13
|
1,36±0,03
|
1,61±0,04
|
1,25±0,11
|
14,20±0,40
|
4,33±0,04
|
1,11±0,00
|
1,21±0,04
|
|
MA4
|
1,36±0,04
|
0,00
|
0,00
|
1,88±0,02
|
1,20±0,02
|
0,00
|
4,00±0,00
|
0,00
|
|
MA6
|
3,64±0,65
|
3,00±0,26
|
3,36±0,31
|
2,15±0,01
|
1,87±0,17
|
0,00
|
0,00
|
0,00
|
|
MA9
|
2,92±0,12
|
1,50±0,00
|
2,45±0,19
|
1,32±0,03
|
1,43±0,29
|
1,67±0,03
|
1,50±0,00
|
1,81±0,03
|
|
MA12
|
3,25±0,12
|
3,10±0,77
|
3,22±0,35
|
1,19±0,04
|
1,39±0,03
|
5,50±0,00
|
1,33±0,04
|
2,40±0,00
|
Grado de hidrólisis: Proporción
entre el tamaño del halo generado (mm) y el tamaño de la colonia (mm).
Identificación molecular
Los resultados evidenciaron que las cepas
bacterianas se encuentran distribuidas en los géneros: Acinetobacter
(MA1), Bacillus (MA3, MA9 y MA12), Staphylococcus (MA2), Comamonas
(MA4) y Aeromonas (MA6). Se evidenció la cercanía evolutiva entre las
cepas MA9 y MA12 y el distanciamiento con la cepa MA3 (Figura 1). Así mismo,
las cepas MA1 y MA2 también evidenciaron cercanía evolutiva. Se ha descrito la
abundancia del género Bacillus en la rizósfera, ya que aislados de
suelos de cultivo de arroz de este género participan como promotores del
crecimiento vegetal al contribuir a la fertilidad del suelo debido a su
actividad hidrolítica (Rekha et al., 2018; Roy et al., 2020). Especies del
género Staphylococcus halotolerantes evidenciaron solubilizar fosfatos,
estimular el crecimiento celular y reducir el daño oxidativo celular antes el
estrés salino en Zea mays L. (Shahid et al., 2019). Además, Staphylococcus
epidermidis AT2, aislada de suelos en Malasia, hidrolizó ácidos grasos de
cadena larga (C18:1), acorde a la cepa MA2 (Kamarudin et al., 2014). Aeromonas
es capaz de degradar el fosfato inorgánico, reducir nitratos a nitritos, y
producir lipasas y proteasas (Aarab et al., 2017; Gonçalves et al., 2019). En
este caso, MA6 metabolizó algunos azúcares con o sin producción de gas. Por
otro lado, Acinetobacter spp. tienen como hábitats suelos de cultivo,
lodos, aguas residuales, entre otros, debido a sus mecanismos de adaptación a
diferentes nichos ecológicos. Especies de Acinetobacter son incapaces de
asimilar azúcares, citrato e hidrolizar gelatina, acorde al aislado MA1 (Carr
et al., 2003). Comamonas jiangduensis, aislada de suelos agrícolas en
Jiangdu (China), fue capaz de asimilar glucosa, manosa, manitol, arabinosa y
maltosa, y producir esterasas, similar a MA4, con diferencia en la producción
de lipasas y gelatinasas (Sun et al., 2013).
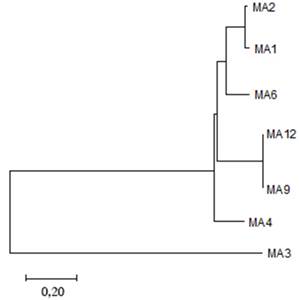
Figura
1.
Árbol filogenético de bacterias aisladas de suelos de cultivos de arroz del
distrito de Rioja.
El aislamiento y caracterización de nuevas
cepas bacterianas es muy importante debido que cada bacteria presenta diversas
características fisioló-gicas y metabólicas, y para su mejor aprove-chamiento
se requiere seguir investigando en la identificación de las especies mediante
pruebas confirmatorias como perfil lipídico de membrana, cálculo del porcentaje
de G+C e hibridación de ADN-ADN, así como uso de otros cronómetros moleculares.
Asimismo, es necesario determinar la capacidad promotora del crecimiento
vegetal de estas cepas con el propósito de utilizarlas como bio-estimulantes de
los cultivos de arroz. De igual modo, se debería evaluar la utilización de
pesti-cidas como fuente de carbono para una posterior aplicación en la
biorremediación de suelos.
De suelos de cultivo de
arroz del distrito de Rioja - Región de San Martín se aislaron bacterias con
crecimiento en un amplio rango de salinidad y pH, variada capacidad metabólica
de azúcares, y perfiles diferentes de sensibilidad.
Además, todas las bacterias
presentaron capacidad hidrolítica de ácidos grasos, algunas mostraron actividad
proteo-lítica, celulolítica, pectinolítica y amilolítica; asimismo se agrupan
en los géneros Bacillus, Acinetobacter, Aeromonas, Comamonas
y Staphylococcus. La capacidad metabólica adaptativa de las bacterias
permitirá utilizar los pesticidas de los suelos del cultivo de arroz como
fuente de carbono, lo cual sería de gran utilidad para la agricultura, el medio
ambiente y la salud pública.
En otros estudios se podría
evaluar la capacidad promotora del crecimiento vegetal de las cepas
bacterianas, para su uso como bioestimulantes de diversos cultivos.
AGRADECIMIENTOS
Esta
investigación fue financiada parcialmente por el contrato Nº 169-2017
FONDECYT-CONCYTEC, Perú.
REFERENCIAS
BIBLIOGRÁFICAS
Aarab,
S., Ollero, F. J., Megias, M., Laglaoui, A., Bakkali, M., & Arakrak, A.
(2017). Simultaneous p-solubilizing and biocontrol activity of
rhizobacteria isolated from rice rhizosphere soil. En: Kumar V., Kumar M.,
Sharma S. & Prasad R. (Eds) Probiotics in Agroecosystem, 207–215. Springer.
Singapore. https://doi.org/10.1007/978-981-10-4059-7_11
Balasjin,
N. M., Maki, J. S., Schläppi, M. R., & Marshall, C. W. (2022). Plant
growth-promoting activity of bacteria isolated from Asian rice (Oryza sativa
L.) depends on rice genotype. Microbiol Spectr., 10(4), e0278721.
https://doi.org/10.1128/spectrum.02787-21
Bauer,
A. W., Kirby, W. M., Sherris, J. C., & Turck, M. (1966). Antibiotic
Susceptibility Testing by a Standardized Single Disk Method. The American
Journal of Clinical Pathology, 45(4), 493–496. https://doi.org/10.1093/ajcp/45.4_ts.493
Becerra
Gutiérrez, L. K., & Horna Acevedo, M. V. (2016). Aislamiento de
microorganismos productores de biosurfactantes y lipasas a partir de efluentes
residuales de camales y suelos contaminados con hidrocarburos. Scientia
Agropecuaria, 7(1), 23–31. https://doi.org/10.17268/sci.agropecu.2016.01.03.
Butt,
M.Q., Zeeshan, N., Ashraf, N.M., Akhtar, M. A., Ashraf, H., Afroz, A., Shaheen,
A., & Naz, S. (2021). Environmental impact and
diversity of protease-producing bacteria in areas of leather tannery effluents
of Sialkot, Pakistan. Environ Sci Pollut Res, 28, 54842–54851.
https://doi.org/10.1007/s11356-021-14477-2.
Calderón-Toledo,
S., Horue, M., Alvarez, V. A., Castro, G. R., & Zavaleta, A. I. (2021).
Isolation and partial characterization of Komagataeibacter sp. SU12 and
optimization of bacterial cellulose production using Mangifera indica
extracts. Journal of Chemical Technology and Biotechnology, 97(6),
1482-1493. doi: https://doi.org/10.1002/jctb.6839
Canales,
P. E., Chávez-Hidalgo, E. L., & Zavaleta, A. I. (2014). Caracterización
de bacterias halófilas productoras de amilasas aisladas de las Salinas de San
Blas en Junín. Revista Colombiana de Biotecnología, 16(2),
150-157. https://doi.org/10.15446/rev.colomb.biote.v16n2.43763
Carr,
E. L., Kämpfer, P., Patel, B. K. C., Gürtler, V., & Seviour, R. J. (2003).
Seven novel species of Acinetobacter isolated from activated sludge. International
Journal of Systematic and Evolutionary Microbiology, 53(4), 953–963.
https://doi.org/10.1099/ijs.0.02486-0
Clinical
and Laboratory Standards Institute. (2018). Performance Standards for
Antimicrobial Susceptibility Testing. 28th ed. CLSI supplement M100. Wayne, PA:
Clinical and Laboratory Standards Institute.
Cycoń,
M., Mrozik, A., & Piotrowska-Seget, Z. (2019). Antibiotics in the soil
environment—degradation and their impact on microbial activity and diversity. Frontiers
in Microbiology, 10, 338. doi: https://doi.org/10.3389/fmicb.2019.00338
Fei,
Y., Huang, S., Zhang, H., Tong, Y., Wen, D., Xia, X., Wang, H., Luo, Y., &
Barceló, D. (2020). Response of soil enzyme activities and bacterial
communities to the accumulation of microplastics in an acid cropped soil. Science
of the Total Environment, 707, 135634. https://doi.org/10.1016/j.scitotenv.2019.135634.
Ganbat,
S., Ganbat, D., Jeong, G., Shin, K-S., Lee, Y-J., Park, M-H., Koh, J., &
Lee, S-J. (2022). A Study on the Diversity and Enzyme Productivity of
Mesophilic and Thermophilic Bacteria from the Arable Soil Fertilized with
Modified Peat Moss. Food Engineering Progress, 26, 84-90.
https://doi.org/10.13050/foodengprog.2022.26.2.84.
García-Bucio,
P., Sotelo-Navarro, P., Poggi-Varaldo, H., Cañizares-Villanueva, R. O., &
Escamilla-Alvarado, C. (2014). Indicadores de la bioeconomía circular
para el aprovechamiento de la fracción orgánica de los residuos sólidos
urbanos. Revista Internacional de Contaminación Ambiental, 38, 78-92. https://doi.org/10.20937/rica.54350.
Gonçalves
Pessoa, R. B., de Oliveira, W. F., Marques, D. S. C., Dos Santos Correia, M. T.,
& de Carvalho, E. V. M. M., Coelho, L. C. B. B. (2019). The genus Aeromonas: A general approach. Microbial
Pathogenesis, 130, 81–94. https://doi.org/10.1016/j.micpath.2019.02.036.
Hernández
Forte, I., Pérez-Pérez, R., Taulé-Gregorio, C. B., Fabiano-González, E.,
Battistoni-Urrutia, F., & Nápoles-García, M. G. (2022). New bacteria genera
associated to rice (Oryza sativa L.) in Cuba promote the crop growth. Agronomía Mesoamericana, 33(2), 47223. https://doi.org/10.15517/am.v33i2.47223.
Kamarudin,
N. H. A., Rahman, R. N. Z. R. A., Ali, M. S. M., Leow, T. C., Basri, M., &
Salleh, A. B. (2014). A new cold-adapted, organic solvent stable lipase from
mesophilic Staphylococcus epidermidis AT2. Protein J, 33, 296–307. https://doi.org/10.1007/s10930-014-9560-3.
Koul,
D., Chander, D., Manhas, R. S., & Chaubey, A. (2021). Isolation and Characterization of Serratiopeptidase Producing Bacteria
from Mulberry Phyllosphere. Curr Microbiol,
78, 351–357. https://doi.org/10.1007/s00284-020-02280-0.
Kurniawan,
T. A., Lo, W. H., & Chan, G. Y. (2006). Physico-chemical
treatments for removal of recalcitrant contaminants from landfill leachate. Journal
of Hazardous Materials, 129(1-3), 80-100.
https://doi.org/10.1016/j.jhazmat.2005.08.010.
Ministerio
de Agricultura, & Riego-MINAGRI. (2017). Informe del
arroz. Lima, Perú: Dirección de estudios económicos e información agraria.
Ministerio
de Agricultura, & Riego-MINAGRI. (2021).
Observatorio de las Siembras y Perspectivas de la Producción de Arroz. Boletín
Cuatrimestral Nº 1.3. Campaña agrícola 2020-2021. Lima, Perú: Dirección General
de Políticas Agrarias.
Ministerio
del Ambiente-MINAM. (2021). Diagnóstico de la situación de las brechas de
infraestructura o de acceso a bienes/servicios. Lima, Perú: Sector
Ambiente.
Ministerio
del Ambiente-MINAM, & Ministerio de Economía y Finanzas-MEF. (2021). Guía
para el cumplimiento de la Meta 3: Implementación de un sistema integrado de
manejo de residuos sólidos municipales. Programa de Incentivos a la Mejora
de la Gestión Municipal del año 2022. Lima, Perú: Dirección General de Gestión
de Residuos Sólidos - Ministerio del Ambiente.
Mithilesh,
K., Anshu, D., Mradul, T., & Venkatesh, K. (2017). Screening of soil
samples for isolation of lipase producing organisms. Research Journal of
Pharmacy and Technology, 10(6), 1730–1733. https://doi.org/10.5958/0974-360X.2017.00305.5.
Moraga
Méndez, D. (2021). Estimación de la temperatura del suelo mediante técnicas
de aprendizaje profundo. Tesis para la obtención del grado de Magíster en
Recursos Naturales. Pontificia Universidad Católica de Valparaíso.
Mushtaq,
H., Ganai, S. A., Jehangir, A., Ganai, B. A., & Dar, R. (2023) Molecular
and functional characterization of protease from psychrotrophic Bacillus
sp. HM49 in North-western Himalaya. PLOS
ONE, 18(3), e0283677. https://doi.org/10.1371/journal.pone.0283677
Palacios,
P. A., Borda-Molina, D., Salvador, J., & Baena, S. (2017). Isolation of lipolytic bacteria from Colombian Andean soils: A target
for bioprospecting. Journal of Microbiology, Biotechnology and Food Sciences,
6(6), 1250–1256. https://doi.org/10.15414/jmbfs.2017.6.6.1250-1256.
Parmar,
N., Singh, A., & Ward, O. P. (2001). Enzyme treatment to reduce solids and
improve settling of sewage sludge. Journal of Industrial Microbiol
Biotechnol, 26(6), 383-386. https://doi.org/10.1038/sj.jim.7000150.
Prittesh,
P., Avnika, P., Kinjal, P., Naik, H., Sakthivel, K., & Amaresan, N. (2020).
Amelioration effect of salt-tolerant plant growth-promoting bacteria on growth
and physiological properties of rice (Oryza sativa) under salt-stressed
conditions. Archives of Microbiology, 202(9), 2419–2428. https://doi.org/10.1007/s00203-020-01962-4.
Puertas
Ramos, F. V. (2009). Índices de calidad del suelo y parámetros de crecimiento
de cultivos de cobertura en una plantación de cacao (Theobroma cacao
L.). Tesis para la obtención del grado de Doctoris Philosophiae en Agricultura
Sustentable. Universidad Nacional Agraria La Molina.
Rekha,
K., Baskar, B., Srinath, S., & Usha, B. (2018). Plant-growth-promoting
rhizobacteria Bacillus subtilis RR4 isolated from rice rhizosphere induces
malic acid biosynthesis in rice roots. Canadian Journal of Microbiology,
64(1), 20–27. https://doi.org/10.1139/cjm-2017-0409.
Roy,
P., Chatterjee, S., Saha, N. C., & Gantait, V. V. (2020). Characterization
of a starch hydrolysing Bacillus flexus U8 isolated from rhizospheric
soil of the paddy plants. Proc. Natl. Acad. Sci., 90,
1075–1081. https://doi.org/10.1007/s40011-020-01176-0.
Sanjinez
Salazar, F. (2019). Sustentabilidad del agroecosistema del cultivo de arroz (Oryza
sativa L.) en Tumbes, Perú. Tesis para la obtención del grado de Doctoris
Philosophiae en Agricultura Sustentable. Universidad Nacional Agraria La
Molina.
Shahid,
M., Ahmed, T., Noman, M., Tariq Javed, M., Rizwan Javed, M., Tahir, M., &
Masood Shah, S. (2019). Non-pathogenic Staphylococcus strains augmented
the maize growth through oxidative stress management and nutrient supply under
induced salt stress. Annals of Microbiology, 69, 727–739. https://doi.org/10.1007/s13213-019-01464-9.
Shahid,
M., & Khan, M. S. (2022). Tolerance of pesticides and antibiotics among
beneficial soil microbes recovered from contaminated rhizosphere of edible
crops. Current Research in Microbial Sciences, 3, 100091. https://doi.org/10.1016/j.crmicr.2021.100091.
Sritongon,
N., Sarin, P., Theerakulpisut, P., & Riddech, N. (2022). The effect of
salinity on soil chemical characteristics, enzyme activity and bacterial
community composition in rice rhizospheres in Northeastern Thailand. Scientific
Reports, 12, 20360. https://doi.org/10.1038/s41598-022-24902-2.
Sun,
L.-N., Zhang, J., Chen, Q., He, J., Li, Q.-F., & Li, S.-P. (2013). Comamonas
jiangduensis sp. nov., a biosurfactant producing bacterium isolated from
agricultural soil. International Journal of Systematic and Evolutionary
Microbiology, 63, 2168–2173. https://doi.org/10.1099/ijs.0.045716-0.
Tang,
Z., Zhang, L., He, N., Liu, Z., Ma, Z., Fu, L., Wang, H., Wang, C., Sui, G.,
& Zheng, W. (2022). Influence of planting methods and organic amendments on
rice yield and bacterial communities in the rhizosphere soil. Frontiers in
Microbiology, 13, 918986. https://doi.org/10.3389/fmicb.2022.918986.
Walitang,
D.I., Kim K., Madhaiyan, M., Kim, Y.K., Kang, Y, & Sa, T. (2017).
Characterizing endophytic competence and plant growth promotion of bacterial
endophytes inhabiting the seed endosphere of rice. BMC Microbiol, 17,
209. https://doi.org/10.1186/s12866-017-1117-0.
Wang,
C., Liu, Z., Huang, Y., Zhang, Y., Wang, X., & Hu, Z. (2019).
Cadmium-resistant rhizobacterium Bacillus cereus M4 promotes the growth
and reduces cadmium accumulation in rice (Oryza sativa L.). Environmental
Toxicology and Pharmacology, 72, 103265. https://doi.org/10.1016/j.etap.2019.103265.

