 |
INTRODUCCIÓN
Patogenicidad de Pectobacterium chrysanthemi en Solanum tuberosum y PCR del espaciador transcrito interno (ITS) del ADNr 16S/23S en aislados de Musa sp. del norte del Perú
Pathogenicity of Pectobacterium chrysanthemi on Solanum tuberosum and PCR of the internal transcribed spacer (ITS) 16S/23S rDNA in isolates of Musa sp. from northern Peru
Edgar Abraham Maldonado Duque1; Angélica María Vigíl-Correa2; Lesly Gianela Zurita Chinguel1;
Maria Dorothy Torres de León2; Roberto Mendoza Rendón5; Cindy Yuriko Saavedra-Rios3, *;
Irwin David Villar Guevara4; Carlos Enrique Cubas Zúñiga5; Merli Clarita Peña Zarate3;
Karol Noemi Vilchez Estrada3; Archi Alejandro Ruiz Polo5
1 Laboratorio de fitopatología, Facultad de Agronomía, Universidad Nacional de Piura, Ciudad Universitaria, Urb. Miraflores S/N, Castilla, Piura, Perú.
2 Laboratorio de Microbiología, Universidad Nacional de Piura, Piura, Perú.
3 Inca Biotec S.A.C, Tumbes, Perú.
4 Departamento de Investigación, Universidad Sedes Sapientiae, Piura, Perú.
5 Departamento de Ciencias Biológicas, Universidad Nacional de Piura, Piura, Perú.
* Autor corresponsal: cyurikosaavedrar@gmail.com (C. Y. Saavedra-Rios).
ORCID de los autores:
E. A. Maldonado Duque: https://orcid.org/0000-0003-0030-3960 I. D. Villar Guevara: https://orcid.org/0000-0001-7958-5061
A. M. Vigíl-Correa: https://orcid.org/0000-0002-3485-6283 C. E. Cubas Zúñiga: https://orcid.org/0000-0001-8001-5357
L. G. Zurita Chinguel: https://orcid.org/0000-0002-4242-2709 M. C. Peña Zarate: https://orcid.org/0009-0004-1654-5651
M. D. Torres de León: https://orcid.org/0000-0002-7277-8474 K. N. Vilchez Estrada: https://orcid.org/0000-0002-3775-0759
R. Mendoza Rendón: https://orcid.org/0000-0002-7743-1082 A. A. Ruiz Polo: https://orcid.org/0009-0005-1273-2625
C. Y. Saavedra-Rios: https://orcid.org/0009-0000-0968-4080
RESUMEN
Pectobacterium chrysanthemi es el agente causal de la pudrición blanda que comúnmente afecta los cultivos de banano orgánico de todo el planeta. Las políticas actuales de control fitosanitario se basan en la sintomatología causada por la enfermedad. Sin embargo, al existir otras especies que causan síntomas similares, se recurre a pruebas de patogenicidad a las que se debe adicionar pruebas bioquímicas para llegar a identificar la bacteria, prescindiendo de ensayos moleculares que, en contraste, permitirían obtener resultados más específicos y en menor tiempo. Este artículo presenta una metodología alternativa en el diagnóstico rápido y específico de la pudrición blanda causada por Pectobacterium chrysanthemi en cultivos de banano orgánico del Perú. La detección mediante virulencia de la bacteria en tejido hospedante de Solanum tuberosum y la PCR del espaciador transcrito interno (ITS) del ADNr 16S/23S, permite el manejo oportuno de la enfermedad. Los resultados demuestran que en el complejo entre la prueba de patogenicidad y el ensayo PCR- ITS 16S/23S es posible detectar a Pectobacterium chrysanthemi en un periodo de 8 días, 3 horas y 15 minutos, demostrando que este método es más eficiente que las pruebas bioquímicas usualmente empleadas para el diagnóstico de la pudrición blanda en Musa sp.
Palabras clave: Pectobacterium chrysanthemi; Solanum tuberosum; prueba de patogenicidad; prueba bioquímica; PCR; ADNr.
ABSTRACT
Pectobacterium chrysanthemi is the causative agent of soft rot that commonly affects organic banana crops around the planet. Current phytosanitary control policies are based on the symptoms caused by the disease. However, since there are other species that cause similar symptoms, pathogenicity tests are used to which biochemical tests must be added to identify the bacteria, dispensing with molecular tests that, in contrast, would allow obtaining more specific results and in less time. time. This article presents an alternative methodology for the rapid and specific diagnosis of soft rot caused by Pectobacterium chrysanthemi in organic banana crops in Peru. Detection by virulence of the bacteria in host tissue of Solanum tuberosum and PCR of the internal transcribed spacer (ITS) of the 16S/23S rDNA allows timely management of the disease. The results demonstrate that in the complex between the pathogenicity test and the ITS-16S/23S PCR assay it is possible to detect Pectobacterium chrysanthemi in a period of 8 days, 3 hours and 15 minutes, demonstrating that this method is more efficient than conventional tests. biochemical tests usually used for the diagnosis of soft rot in Musa sp.
Keywords: Pectobacterium chrysanthemi; Solanum tuberosum; pathogenicity test; biochemical test; PCR; ADNr.
Recibido: 06-06-2024.
Aceptado: 26-08-2024.
El banano orgánico (con sus variedades musáceas) es uno de los frutos con mayor demanda internacional, por lo que su producción se mantiene durante todo el año (Mehendran et al., 2022). Esto genera que sea uno de los productos con mayor preocupación en cuanto a control fitosanitario se refiere.
Distintas bacterias fitopatógenas poseen una virulencia caracterizada por sintetizar enzimas pectinolíticas, que cuando infectan al vegetal causan la maceración de células y la pudrición del tejido parenquimatoso de los órganos afectados (Garibaldi & Bateman, 1971; Collmer & Keen, 1986). En Musáceas, esta virulencia se manifiesta en la enfermedad de la pudrición blanda, siendo las bacterias del género Pectobacterium (antes conocido como Erwinia) las principales responsables (Luna et al., 2009; Rodríguez et al., 2020), específicamente Pectobacterium chrysanthemi (Rahmanifar et al., 2012) y Pectobacterium carotovora subsp. carotovorum (Gardan et al., 2003).
Desde hace mucho se vienen realizando pruebas bioquímicas para la detección de Pectobacterium chrysanthemi en el diagnóstico de la pudrición blanda de musáceas (Snehalatharani & Khan, 2010). Sin embargo, demandan un mayor tiempo y un alto costo en la obtención de resultados. De hecho, al considerarse un método no muy confiable, resulta necesario que se realicen pruebas de patogenicidad previas a las pruebas bioquímicas (Llanos, 1967); optando por el uso de hospedantes como Solanum tuberosum (Cardoza et al., 2007), y otras especies de vegetales.
Por otro lado, se han desarrollado técnicas alternativas que emplean metodologías y equipos que permiten la identificación de Pectobacterium chrysanthemi empleando ácidos nucleicos, siendo la PCR de regiones espaciadoras de transcripción interna (ITS, por sus siglas en inglés) del ADNr 16S/23S una de ellas (Kwon et al., 2000). Esta técnica cuenta con un valor agregado adicional en el diagnóstico, ya que permite determinar si existe o no divergencia entre las especies (Sufys et al., 2001; Blaiotta et al., 2002).
La situación en Perú es desalentadora ya que existen pocos estudios dirigidos a la detección de Pectobacterium chrysanthemi en banano orgánico (Musa sp.), y en los que existen, solo se han utilizado métodos tradicionales, como pruebas de patoge-nicidad complementadas con pruebas bioquímicas. No obstante, actualmente el Instituto Nacional de Investigación Agraria (INIA) viene realizando la detección molecular de microorganismos fitopató-genos, como es el caso de Erwinia sp., aunque solo mediante la secuenciación del ADN genómico (Estrada et al., 2023).
En ese contexto, se ha desarrollado una métodología In vitro, con la cual se ha detectado a Pectobacterium chrysanthemi presente en especímenes del norte del Perú. El método es muy confiable, y consiste en una prueba de patoge-nicidad con Solanum tuberosum, seguido de una PCR del ITS-ADNr 16S/23S en aislados de Musa sp. Anteriormente, se han realizado estudios con técnicas moleculares en los que se han detectado especies del género Pectobacterium (Li et al., 2011). Sin embargo, escasamente se emplean en laboratorios de fitopatología del Perú; dando lugar a un desconocimiento de sus aplicaciones. Por ello, con la finalidad de promover métodos y/o procedimientos alternativos, que puedan ser provistos por laboratorios peruanos, en beneficio de empresas u organizaciones que se desempeñan en la producción de banano orgánico, se realizó este estudio.
Esta investigación tuvo como objetivo proponer un método alternativo y confiable para el diagnóstico de la pudrición blanda en Musa sp. mediante una prueba de patogenicidad de Pectobacterium chrysanthemi en Solanum tuberosum seguida de un ensayo PCR del espaciador transcrito interno (ITS) del ADNr 16S/23S.
MATERIAL Y MÉTODOS
Diseño de la investigación
La investigación fue de tipo observacional, des-criptiva e In vitro; se basa en el uso de técnicas de microbiología y técnicas moleculares con las que se propuso un complejo de detección rápido y específico de Pectobacterium chrysanthemi, para el diagnóstico confiable de la pudrición blanda en Musa sp.
Obtención de material biológico
Se analizó tejido vegetal de la capa externa del pseudotallo de banano orgánico (Musa sp.) proporcionado por el laboratorio de sanidad vegetal de la Universidad Nacional de Piura, cumpliendo criterios como: tejido con síntomas de necrosis y pudrición blanda.
Aislamiento bacteriano
Porciones de tejido vegetal con necrosis blanda fue colocado en tubos de ensayo con 5 mL de agua destilada estéril dejándose reposar por 10 min para la difusión de células bacterianas. Se observó un aspecto lechoso en la solución, utilizando un asa de Kolle flameada se realizó un sembrado por estría en placas Petri con agar nutritivo (AN) e incubaron en posición invertida a 28 ± 1 °C, durante 48 h en oscuridad (French, 1980).
Características morfológicas
Se evaluaron características morfológicas de cada aislado en medio AN (color, forma, tamaño, elevación y textura) (Skerman, 1960). Se tomaron como muestras presuntivas a los aislados de Musa sp. con morfología de Pectobacterium chrysanthemi y se masificaron en 1 mL de caldo nutritivo (CN).
Prueba de patogenicidad en S. tuberosum
Se usaron tubérculos de Solanum tuberosum (papa), en los cuales se desinfectó toda la superficie externa con alcohol al 70% por 30 segundos. Luego, fueron inmersos en hipoclorito de sodio al 1,5% por 4 minutos y se lavaron con agua destilada estéril. Los tubérculos se cortaron en rodajas con cortes céntricos, en cada una se inoculó 0,5 mL de caldo nutritivo (3 x 108 UFC/mL) y se incubaron en una cámara húmeda (Llanos, 1967). La manifestación de síntomas se evaluó cada 24 horas durante 5 días. Las rodajas con virulencia característica de Pectobacterium chrysanthemi se tomaron como presuntivas y se masificaron en 1 mL de CN incubado a 28 ± 1 °C por 24 horas.
Extracción de ADN
Los tubos con los aislados presuntivos fueron centrifugados a 14 mil rpm por 10 minutos, se eliminó el sobrenadante y en el pellet obtenido se añadió 300 uL de buffer lisis casero. Posteriormente, el tubo se incubó a 56 °C por 20 minutos, se centrifugó nuevamente y el sobrenadante se transfirió a un microtubo de 1,5 mL estéril. Se añadió 300 uL de etanol en 70% y se centrifugó nuevamente para luego eliminar el sobrenadante. El pellet se lavó con 300 uL de etanol en 70%, se centrifugó, se eliminó el sobrenadante y se dejó en reposo por 10 minutos a temperatura ambiente. Luego, se realizó un tratamiento con la enzima RNasa M4253 Promega™ de acuerdo con las instrucciones del fabricante. Por último, se añadió 100 uL de TE 1X (Tris 1 mM y EDTA 0,1 M pH8) y cada tubo se incubó a -20 °C.
PCR del espaciador transcrito interno del ADNr 16S-23S de Pectobacterium chrysanthemi
La PCR del ITS-ADNr 16S/23S se realizó con los cebadores diseñados por Li et al. (2013), quienes compararon ITS de 18 secuencias diferentes de Erwinia del GenBank y las analizaron en los softwares Primer3Plus de acuerdo con Li et al. (2013) y Net-Primer, para luego evaluar su especificidad con el algoritmo de alineación local básica (BLAST). Cebador LF (5-TTCGTCTAGAGGCCCAGGAC-3) y Cebador LR (5-TCAGCTTGTTCCGGATTGTT-3).
En cuanto a la amplificación del ITS-ADNr 16S/23S del presente estudio, los cebadores se mezclaron con componentes del kit GeneON™ DNA Polymerase en un volumen final de 25 µL, con 11,25 μL de agua ultra pura, 5 μL de buffer (1 X), 1,5 μL de MgCL (1,5 mM), 0,5 μL de dNTPs (200 μM), 1,25 μL de Forward (10 uM), 1,25 μL de Reverse (10 uM), 0,25 μL de enzima Gotaq Polimerasa (1 U/reacción) y 4 μL del ADN. Las condiciones térmicas y el ciclaje consistieron en una desnaturalización inicial de 98 °C por 4 minutos, seguido de 35 ciclos con 95 °C por 30 segundos para la desnaturalización, 60 °C por 45 s para la hibridación, 72 °C por 1 minuto para la extensión, una post extensión de 72 °C por 5 min y una temperatura de conservación de 4 °C por hasta 24 h.
Electroforesis en gel de agarosa
Los productos PCR se analizaron por electroforesis en gel de agarosa al 1,5 % (60 mL de Tris-Acetato-EDTA 1X, 0,9 gramos de agarosa, 3 uL de bromuro de etidio). En cada pocillo del gel se añadieron 4 uL de buffer (6X DNA loading dye) y 8 uL de muestra (productos PCR). Las condiciones eléctricas fueron de 80 voltios y 200 Amp por 30 min.
RESULTADOS Y DISCUSIÓN
Aislamiento bacteriano
Se aislaron 5 cepas bacterianas de diferentes localidades del norte del Perú (Tabla 1). Los aislados presentaron una morfología caracterizada por colonias pequeñas con forma circular y convexa de bordes enteros, con un color amarillo cremoso y de consistencia mucoide con brillo (Figura 1). Esta morfología concuerda con descripciones de Pectobacterium chrysanthemi aislada de cultivos Musa sp. de las mismas localidades (Querecotillo y Marcavelica) (Aguilar et al., 2021). Además, la especie comúnmente es descrita con colonias pequeñas, redondas, levantadas y ligeramente translúcidas de color amarillo (Dickey & Victoria, 1980; Kunstmann et al., 2006). Por tanto, la morfología observada corresponde a la bacteria de interés en este estudio, tomándose como presuntivas hasta las pruebas de patogenicidad y ensayos PCR.
Tabla 1
Aislados bacterianos de banano orgánico del norte del Perú
|
Localidad |
Coordenadas |
Código de aislados |
|
Chulucanas |
5°5′47.42″S/80°9′39.32″O |
AB1MV1 AB2MV1 |
|
Querecotillo |
4°50′16″S/80°38′44″O |
ABMV2 |
|
Marcavelica |
4°52′54″S/80°42′12″O |
ABMV3 |
|
Samán |
4°50'13"S/80°45'50″O |
ABMV4 |
AB: Aislado bacteriano; MV: Muestra Vegetal.
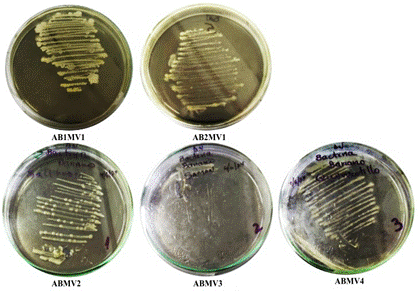
Figura 1. Aislados bacterianos del pseudotallo de Musa sp. del norte del Perú.
Prueba de patogenicidad en Solanum tuberosum
Indistintamente de las cepas y su origen, después de que los aislados se inocularon en Solanum tuberosum, en todas se observó resplandecimiento de pudrición en el día 3 y la percepción de olor fétido en el día 5 (Tabla 2). Estos síntomas y el tiempo de su manifestación son similares a los reportados por Aguilar et al. (2021) y Nabhan et al. (2012) en Solanum tuberosum (entre 2 y 7 días después de la inoculación). De acuerdo con Charkowski et al. (2020) y Mainello et al. (2024), Solanum tuberosum comúnmente es un vegetal huésped de especies Pectobacterium en todo el mundo. Así mismo, otros autores señalan que la infección de tubérculos de papa por especies del género Pectobacterium se manifiesta en la pudrición del tejido vegetal (Cardoza & Nalimova, 2008; Amaya et al., 2021). Por otro lado, como agente causal de necrosis y pudrición blanda en Musáceas, se ha reportado a Pectobacterium chrysanthemi y Pectobacterium carotovora subsp. carotovorum (Hernández, 2022). No obstante, existen especies de bacterias de la familia Enterobacter que en los últimos años se han reportado como agente causal de la pudrición blanda, tal es el caso de Klebsiella michiganensis y Klebsiella variicola (Nolasco Cárdenas et al., 2022). Sin embargo, la pudrición inducida con los inóculos sobre rodajas de Solanum tuberosum, puede variar en su aspecto, dependiendo de la especie involucrada, ya sea Pectobacterium, Pseudomonas, o Bacillus (Goto, 1990).
Tabla 2
Síntomas de la patogenicidad de Pectobacterium chrysanthemi en Solanum tuberosum
|
Síntoma |
Tiempo de manifestación |
Código de aislados |
|
Pudrición |
3 días |
AB1MV1 AB2MV1 |
|
Olor fétido |
5 días |
|
|
Pudrición |
3 días |
ABMV2 |
|
Olor fétido |
5 días |
|
|
Pudrición |
3 días |
ABMV3 |
|
Olor fétido |
5 días |
|
|
Pudrición |
3 días |
ABMV4 |
|
Olor fétido |
5 días |
AB: Aislado bacteriano; MV: Muestra Vegetal.
PCR del espaciador transcrito interno (ITS) del ADNr 16S-23S de Pectobacterium chrysanthemi
En los geles de agarosa se observó que todos los aislados amplificaron un fragmento ADN de 171 pb, el cual codifica una región ITS del ADNr 16S/23S de Pectobacterium chrysanthemi (Figura 2). Nuestros resultados coinciden con los de Li et al. (2013), de donde se obtuvieron las secuencias de cebadores empleados para este estudio. Los ITS del ADNr 16S/23S ya han sido probadas con éxito para la detección e identificación de Pectobacterium chrysanthemi (Toth et al., 2001). Estudios moleculares basados en la detección del material genético de este microorganismo han permitido su identificación específica en China (Liu et al., 2016), (Ragavil et al., 2019) y Turquía (Basim et al., 2019). Cabe resaltar que el presente estudio, es uno de los primeros en los que se ha detectado al agente causal de la pudrición blanda directamente de su material genético.
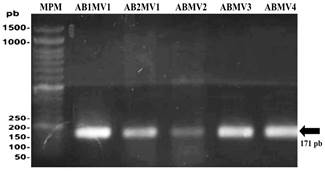
Figura 2. Electroforesis en gel de agarosa al 1.5% de productos PCR de la detección molecular de P. chrysanthemi en aislados bacterianos. MPM: Marcador de peso molecular de 100pb. AB1MV1: Aislamiento bacteriano 1 de Chulucanas. AB2MV1: Aislamiento bacteriano 2 de Chulucanas. ABMV2: Aislamiento bacteriano de Querecotillo. ABMV3: Aislamiento de Marcavelica. ABMV4: Aislamiento bacteriano de Saman.
En este estudio, realizando una prueba de patogenicidad en tejido vegetal hospedante de Solanum tuberosum y una PCR punto final, fue posible detectar a Pectobacterium chrysanthemi como agente causal de la pudrición blanda en Musa sp. del norte del Perú (Figura 3). Siendo un diagnóstico que toma 8 días, 3 horas y 15 minutos, tiempo menor al empleado en pruebas de patogenicidad complementadas con pruebas bioquímicas para la detección de las especies del género Pectobacterium (Cuppels & Kelman, 1974; Fiori et al., 2005; Vargas et al., 2022).
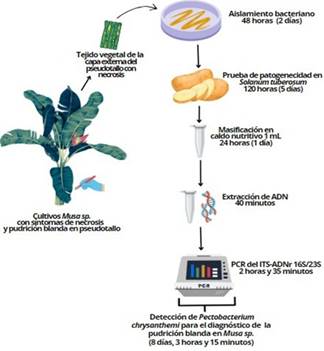
Figura 3. Flujo de trabajo en In vitro para el diagnóstico de la pudrición blanda de Musa sp. mediante patogenicidad de Pectobacterium chrysanthemi en Solanum tuberosum y PCR del espaciador transcrito interno (ITS) del ADNr 16S/23S en aislados.
La identificación de algunas especies del género Pectobacterium se divide en dos grupos: los clásicos y los moleculares. En el primer grupo, es necesario caracterizar los rasgos fenotípicos, fisiológicos, bioquímicos y patológicos (Dadaşoğlu & Kotan, 2017). En el segundo grupo, se analiza el ADN de los microorganismos a partir de marcadores genéticos (Zanoli & Spoto, 2013; Zhang & Tanner, 2017).
De especial interés son las técnicas moleculares, ya que son más rápidas, sensibles, específicas y confiables para la detección, identificación y cuantificación de microorganismos de interés en la producción de cultivos. Siendo la PCR y sus variantes, las que mayormente se utilizan por su rapidez y especificidad, ya sea desde una planta huésped o un cultivo bacteriano puro (Belete & Boyraz, 2019).
El presente estudio destaca por demostrar un complejo de detección alternativo y muy confiable para el diagnóstico de la pudrición blanda en Musa sp. La especificidad de este método permitirá la toma de decisiones oportunas en el manejo de la enfermedad dentro de potenciales escenarios epidemiológicos causados por el cambio climático actual del planeta. Además, que, al trabajar con material genético, se podrá estudiar la dispersión y/o circulación de cepas en el Perú y Sudamérica mediante análisis filogenéticos que contribuyan a la vigilancia de la enfermedad.
CONCLUSIONES
Pectobacterium chrysanthemi posee una virulencia caracterizada por sintetizar proteínas que degradan la pared celular vegetal. Dichas proteínas son conocidas como enzimas pectinolíticas, que una vez que infectan los órganos, maceran las células y causan la pudrición del parénquima. Esta patogenicidad en Solanum tuberosum se explicó en la degradación celular del tejido, observándose resplandecimiento de pudrición y olor fétido en el día 3 y 5 después de la inoculación, respecti-vamente. Estos resultados señalan el factible uso de Solanum tuberosum como tejido hospedante de la bacteria. En particular, por su fácil y económico acceso para pruebas de laboratorio en Perú.
El diagnóstico de la pudrición blanda en Musáceas basado en pruebas de patogenicidad en tejido hospedero no es suficiente, ya que otros micro-organismos poseen virulencia similar. Un adecuado diagnóstico es aquel en el que los resultados son específicos y se obtienen en un tiempo corto, sin la necesidad de recurrir a una variedad de sustratos y/o soluciones químicas (pruebas bioquímicas) que demandan mucho tiempo en las reacciones. Además, que, al trabajar una muestra, esta se pierde durante el proceso, algo no recomendable en la detección de nuevas etiologías, por lo que el diagnóstico de fitopatógenos a partir de material genético es la opción más atractiva debido a que adicionalmente se obtiene la información genética del microorganismo. A pesar de la importancia de estos métodos, poco o nada son utilizados en el Perú, lo cual se ve reflejado en la poca información genética disponible en los bancos de genes, justificando la necesidad de llevar a cabo el uso de la biotecnología molecular en el sector agrario. Finalmente, en este estudio se detectó a Pectobacterium chrysanthemi mediante PCR del espaciador transcrito interno del ADNr 16S/23S, evidenciando su actual circulación entre los culti-vos de banano orgánico (Musa sp.) de las localidades de Chulucanas, Querecotillo, Marcave-lica y Saman, ubicadas al norte del Perú.
AGRADECIMIENTOS
Al departamento de Sanidad Vegetal de la Univer-sidad Nacional de Piura, por proporcionarnos el material vegetal analizado en el presente estudio. Al MSc. Jaime Napoleón Fernandez Ponce, Jefe del Laboratorio de Microbiología de la Universidad Nacional de Piura, por facilitarnos el uso de áreas y equipos empleados en la investigación.
REFERENCIAS BIBLIOGRÁFICAS
Aguilar-Anccota, R., Ruiz, WR, Morales-Pizarro, A., Rafael-Rutte, R., Tirado-Lara, J., Saucedo-Bazalar, M., & Teodor, K. K. (2021). Pudrición blanda en el pseudotallo de banano orgánico (Musa sp): sintomatología, caracterización cultural y bioquímica, patogenicidad y alternativas de manejo. Scientia Agropecuaria, 12(4), 571-578. http://dx.doi.org/10.17268/sci.agropecu.2021.061
Amaya Guerrero, A. P., Beltrán Pineda, M. E., & Alfonso Vargas, N. C. (2021). Pectobacterium carotovorum: agente fitopatógeno causante de la pudrición blanda en la papa (Solanum tuberosum). Ciencia y Tecnología Agropecuaria, 22(2). https://doi.org/10.21930/rcta.vol22_num2_art:1710
Basim, H., Basim, E., Bakİ, D., & Turgut, A. (2019). Enfermedad de la pudrición húmeda del banano (Musa sp.) causada por Pectobacterium carotovorum subsp. carotovorum en Turquía. Revista canadiense de patología vegetal, 41(2), 174-187. https://doi.org/10.1080/07060661.2019.1577302
Blaiotta, G., Pepe, O., Mauriello, G., Villani, F., Andolf, R., & Moschetti, G. (2002). 16S–23S rDNA intergenic spacer region polymorphism of Lactococcus garvieae, Lactococcus rafnolactis and Lactococcus lactis as revealed by PCR and nucleotide sequence analysis. Syst Appl Microbiol, 25(4), 520–527. https://doi.org/10.1078/07232020260517652
Belete, T., & Boyraz, N. (2019). Biotechnological tools for detection, identification and management of plant diseases. African Journal of Biotechnology, 18(29), 797-807. https://doi.org/10.5897/AJB2018.16591
Cardoza, Y. F., Nalimova, M. S., & Izquierdo, M. F. C. (2007). Patogenicidad y virulencia de cepas de Pectobacterium carotovorum y Dickeya chrysanthemi en papa (Solanum tuberosum L.). Fitosanidad, 11(1), 15-18.
Cardoza, Y. F., & Nalimova, M. S. (2008). Determinación de actividades enzimáticas implicadas en la virulencia de cepas de Pectobacterium carotovorum subsp. carotovorum y Dickeya chrysanthemi aisladas de papa. Agro sur, 36(3), 130-136.
Charkowski, A., Sharma, K., Parker, M. L., Secor, G. A., & Elphinstone, J. (2020). Bacterial diseases of potato. The potato crop: its agricultural, nutritional and social contribution to humankind, 351-388. https://doi.org/10.1007/978-3-030-28683-5
Collmer, A. & Keen, N.T. (1986). El papel de las enzimas pécticas en la patogénesis de las plantas. Revista anual de fitopatología, 24(1), 383-409. https://doi.org/10.1146/annurev.py.24.090186.002123
Cuppels, D., & Kelman, A. (1974). Evaluation of selective media for isolation of soft-rot bacteria from soil and plant tissue. Phytopathology, 64(4), 468-475.
Dadaşoğlu, F., & Kotan, R. (2017). Identification and characterization of Pectobacterium carotovorum. Journal of Animal and Plant Sciences, 27(2), 647-654. http://www.thejaps.org.pk/docs/v-27-2/36.pdf
Dickey, R., & Victoria, J., (1980). Taxonomy and emended description of strains of Erwinia Isolated from Musa paradisiaca L. Int. J. Syst. Bacteriol, 30(3), 129–134.
Estrada, R., Saldaña, C. L., Pérez-Porras, W., Arteaga, L., Martínez, G., Injante, P., & Arbizu, CI (2023). Borrador de recurso de secuencia del genoma de Erwinia sp. Cepa INIA 01, fitopatógeno aislado de un tallo enfermo de maíz peruano. Anuncios de recursos de microbiología, 12(5), e00120-23. https://doi.org/10.1128/mra.00120-23
Fiori, M., Virdis, S., & Schiaffino. A. (2005). Phenotypic and genetic characterization of Erwinia carotovora ssp. Carotovora (Jones) Bergey et al., Isolates from grafted tomato in Sardinia, Italy. Phytopathol. Mediterr, 44, 50–57.
French, E., & Hebert, T. (1980). Métodos de Investigación Fitopatológica. San José, Costa Rica, Editorial IICA de Costa Rica. 236p.
Gardan, L., Gouy, C., Christen, R., & Samson, R. (2003). Elevation of three subspecies of Pectobacterium carotovorum to species level: Pectobacterium atrosepticum sp. nov., Pectobacterium betavasculorum sp. nov. and Pectobacterium wasabiae sp. nov. International journal of systematic and evolutionary microbiology, 53(2), 381-391. https://doi.org/10.1099/ijs.0.02423-0
Garibaldi, A., & Bateman, D. F. (1971). Enzimas pécticas producidas por Erwinia chrysanthemi y sus efectos sobre el tejido vegetal. Patología Fisiológica de las Plantas, 1(1), 25-40. https://doi.org/10.1016/0048-4059(71)90037-3
Goto, M. (1990). Fundamentals of bacterial plant pathology. San Diego, USA: Academic Press. 342 p.
Hernández, Y. (2022). Bacterias causantes de enfermedades en cultivos de interés agrícola en Venezuela. Revista de la Facultad de Agronomía, 1(76), 133.
Kunstmann, J. P., Ciampi, L., Böhm, L., Barrera, S., & Collado, L. (2006). Determinación de especies de Erwinia (grupo carotovora) como agentes causales de pudrición blanda en Cala (Zantedeschia spp.). Agricultura Técnica, 66(3), 247-255. http://dx.doi.org/10.4067/S0365-28072006000300003
Kwon, S.W., Myung, I.S., & Go, S.J. (2000). Detección de Pectobacterium chrysanthemi utilizando cebadores de PCR específicos diseñados a partir de la región espaciadora intergénica del ARNr 16S-23S. La Revista de Patología Vegetal, 16 (5), 252-256.
Llanos, C. (1967). Una nueva enfermedad del plátano en el Valle del Cauca: la bacteriosis. Agricultura Tropical, 23(12), 806-812.
Li, P., Lin, B., Shen, H., & Pu, X. (2013). Species-specific detection of Dickeya sp. (Pectobacterium chrysanthemi) in infected banana tissues, soil and water. African Journal of Biotechnology, 10(74), 16769-16773. http://doi.org/10.5897/AJB11.1248
Liu, Q., Xiao, W., Wu, Z., Li, S., Yuan, Y., & Li, H. (2016). Identificación de Dickeya dadantii como agente causal de la pudrición de la vaina bacteriana del banano en China. Revista de patología vegetal, 98(3), 503-510.
Luna-Rodríguez, M., Toledo-González, A., & Iglesias-Andreu, L. G. (2009). Identificación de Enterobacterias Causantes de Pudriciones Blandas en Cucurbita pepo L. en Veracruz, México. Revista mexicana de fitopatología, 27(1), 64-68.
Mainello-Land, A. M., Bibi, S., Gugino, B., & Bull, C. T. (2024). Secuencia multilocus y análisis fenotípico de cepas tipo Pectobacterium y Dickeya para la identificación de Pectobacteriaceae de pudrición blanda a partir de tallos y tubérculos de papa sintomáticos en Pensilvania. Microbiología Sistemática y Aplicada, 47(1), 126476. https://doi.org/10.1016/j.syapm.2023.126476
Mehendran, Y., Kartheeswaran, T., & Kodikara, N. (2022). Banana Freshness Identification Using Image Processing Techniques. 7th International Conference on Business and Industrial Research (ICBIR), 347–352. https://doi.org/10.1109/ICBIR54589.2022.9786519
Nabhan, S., De Boer, S. H., Maiss, E., & Wydra, K. (2012). Taxonomic relatedness between Pectobacterium carotovorum subsp. carotovorum, Pectobacterium carotovorum subsp. odoriferum and Pectobacterium carotovorum subsp. brasiliense subsp. nov. Journal of Applied Microbiology, 113(4), 904-913. https://doi.org/10.1111/j.1365-2672.2012.05383.x
Nolasco Cárdenas, O. P., Velarde Vílchez, M. M., Murrugarra Bringas, V. Y., Acuña Payano, R., & Chacón Aguilar, C. I. (2022). Ribotipificación de bacterias relacionadas a pudrición blanda en banano orgánico del distrito de Querecotillo. (Tesis de pregrado). Universidad Nacional Federico Villarreal, Lima.
Ragavil, G., Muthamilan, M., Nakkeeran, S., Kumaravadivel, N., Sivakumar, U., & Suganthi, A. (2019). Molecular detection of the causative agent of soft rot (Pectobacterium carotovorum subsp carotovorum) in banana (Musa sp.). International Journal of Current Microbiology and Applied Sciences, 8(11), 1854-1868. https://doi.org/10.20546/ijcmas.2019.811.218
Rahmanifar, B., Hasanzadeh, N., Razmi, J., & Ghasemi, A. (2012). Genetic diversity of Iranian potato soft rot bacteria based on polymerase chain reaction-restriction fragment length polymorphism (PCR-RFLP) analysis. African J Biotech. 11, 1314–1320. http://doi.org/10.5897/AJB11.2790
Rodríguez, D., Hernández, Y., Casassa-Padrón, A., & Arcia, M. A. (2020). La Sociedad Venezolana de Fitopatología y su aporte a la Sanidad Vegetal del país. Agronomía Tropical, 70, 1-15. https://doi.org/10.5281/zenodo.4323319
Skerman, V. B. (1960). A guide to the identification of the genera of bacteria. Academic Medicine, 35(1), 92.
Snehalatharani, A., & Khan, A. N. A. (2010). Biochemical and physiological characterisation of Erwinia species causing tip-over disease of banana. Archives Of Phytopathology And Plant Protection, 43(11), 1072-1080. https://doi.org/10.1080/03235400802285422
Sufys, P. N., da Silva, R. A., de Oliveira, M., Campos, C. E., Barreto, A. M., Portaels, F., Rigouts, L., Wouters, G., Jannes, G., van Reybroeck, G., Mijs, W., & Vanderborght, B (2001). Rapid identification of Mycobacteria to the species level using INNO-LiPA Mycobacteria, a reverse hybridization assay. J Clin Microbiol, 39(12), 4477–4482. https://doi.org/10.1128/JCM.39.12.4477-4482.2001
Toth, I. K., Avrova, A. O., & Hyman, L. J. (2001). Rapid identification and differentiation of the soft rot erwinias by 16S-23S intergenic transcribed spacer-PCR and restriction fragment length polymorphism analyses. Applied and Environmental Microbiology, 67(9), 4070-4076. https://doi.org/10.1128/AEM.67.9.4070-4076.2001
Vargas-Fernández, J. P., Wang-Wong, A., & Muñoz-Fonseca, M. (2022). Microorganismos asociados a la enfermedad conocida como pudrición suave del fruto de banano (Musa sp.) y alternativas de control microbiológicas y químicas a nivel in vitro. Agronomía Costarricense, 46(2), 61-76. http://dx.doi.org/10.15517/rac.v46i2.52046
Zhang, Y., & Tanner, N. A. (2017). Isothermal amplification of long, discrete DNA fragments facilitated by single-stranded binding protein. Scientific Reports, 7(1), 84-97. http://doi.org/10.1038/s41598-017-09063-x
Zanoli, L. M., & Spoto, G. (2013). Isothermal amplification methods for the detection of nucleic acids in microfluidic devices. Biosensors, 3(1), 18-43. http://doi.org/10.3390/bios3010018