Dispersión,
caracterización molecular y filogenética de Pectobacterium chrysanthemi
en el marco de fenómenos climáticos
Dispersion,
molecular and phylogenetic characterization of Pectobacterium chrysanthemi
within the framework of climatic phenomena
Cindy Yuriko Saavedra-Rios1;
Angélica María Vigil-Correa3; Irwin David Villar-Guevara4
Carlos Enrique Cubas-Zúñiga5;
Edgar Abraham Maldonado-Duque2; Lesly Gianela Zurita-Chinguel2
Maria Dorothy Torres de
León5; Roberto Mendoza-Rendón5; Alfredo Julian
Sandoval-Norabuena5
Archi Alejandro Ruiz-Polo5
1 Inca Biotec
S.A.C, Tumbes, Perú, Jr. Filipinas S/N, Tumbes, Tumbes, Perú
2 Laboratorio de
Fitopatología, Universidad Nacional de Piura, Piura, Perú.
3 Laboratorio de
Microbiología, Facultad de Ciencias, Universidad Nacional de Piura, Piura, Perú.
4 Departamento de
Investigación, Universidad Sedes Sapientiae, Piura, Perú.
5 Departamento de Ciencias
Biológicas, Universidad Nacional de Piura, Piura, Perú.
*
Autor corresponsal: cyurikosaavedrar@gmail.com
(C. Y.
Saavedra-Rios).
ORCID
de los autores:
C.
Y. Saavedra-Rios: https://orcid.org/0009-0000-0968-4080 L.
G. Zurita-Chinguel: https://orcid.org/0000-0002-4242-2709
A.
M. Vigil-Correa: https://orcid.org/0000-0002-3485-6283 M.
D. Torres de León: https://orcid.org/0000-0002-7277-8474
I.
D. Villar-Guevara: https://orcid.org/0000-0001-7958-5061 R.
Mendoza-Rendón: https://orcid.org/0000-0002-7743-1082
C.
E. Cubas-Zúñiga: https://orcid.org/0000-0001-8001-5357 A.
J. Sandoval-Norabuena: https://orcid.org/0000-0003-2947-419X
E.
A. Maldonado-Duque: https://orcid.org/0000-0003-0030-3960 A. A.
Ruiz-Polo: https://orcid.org/0009-0005-1273-2625
______________________________________________________________________________________________________________________________________________
RESUMEN
La investigación sobre la dispersión, así
como la caracterización molecular y filogenética de Pectobacterium
chrysanthemi, en el contexto de los fenómenos climáticos en Perú, no se ha
realizado hasta la fecha. El objetivo de este estudio fue determinar la
dispersión, y realizar la caracterización molecular y filogenética de Pectobacterium
chrysanthemi en el marco de fenómenos climáticos en Perú. Se realizaron
inspecciones sanitarias en el norte de Perú los días 4 y 30 de marzo de 2024.
Durante estas evaluaciones, se registró la temperatura ambiental, la presencia
de hojarasca en el suelo, la duración del riego y el número de plantas de
banano afectadas por la pudrición blanda. Se colectaron porciones de
pseudotallos afectados para el diagnóstico de la enfermedad, y se obtuvo el
marcador molecular ITS-ADNr 16S/23S de Pectobacterium chrysanthemi, que
fue secuenciado y analizado mediante herramientas bioinformáticas. En la
primera inspección, la temperatura ambiental fue de 27,9 °C, y en la segunda,
de 31,5 °C. En ambas inspecciones se observó la presencia de hojarasca y un
riego intensivo cada siete días. Se identificó la dispersión del patógeno desde
plantas enfermas a sanas en un periodo de 26 días y en un radio de 100 metros.
Las características moleculares y filogenéticas de las cepas estudiadas
revelaron dos genotipos circulantes (PN24.1. y PN24.2.). La pudrición blanda se
manifiesta y se dispersa de plantas enfermas a sanas en un lapso de 26 días y
en un radio de 100 metros. Los genotipos PN24.1. y PN24.2. son los
responsables.
Palabras
clave:
Pectobacterium
chrysanthemi;
ADN; PCR; banano; marchitez.
ABSTRACT
Research on the dispersal,
as well as the molecular and phylogenetic characterization of Pectobacterium
chrysanthemi, in the context of climatic phenomena in Peru, has not been
carried out to date. The objective of this study was to determine the dispersion
and carry out the molecular and phylogenetic characterization of Pectobacterium
chrysanthemi within the framework of climatic phenomena in Peru. Sanitary
inspections were carried out in northern Peru on March 4 and 30, 2024. During
these evaluations, the environmental temperature, the presence of leaf litter
in the soil, the duration of irrigation and the number of banana plants were
recorded. . Affected by soft rot. Portions of affected pseudostems were
collected for diagnosis of the disease, and the ITS-ADNr 16S/23S molecular
marker of Pectobacterium chrysanthemi was obtained, which was sequenced
and analyzed using bioinformatic tools. In the first inspection, the
environmental temperature was 27.9 °C, and in the second, 31.5 °C. In both
inspections, the presence of leaf litter and intensive irrigation every seven
days will be verified. The spread of the pathogen from diseased to healthy
plants was identified over a period of 26 days and within a radius of 100
meters. The molecular and phylogenetic characteristics of the strains studied
revealed two circulating genotypes (PN24.1. and PN24.2.). Soft rot manifests
and spreads from diseased to healthy plants within a period of 26 days and
within a radius of 100 meters. The PN24.1 genotypes. and PN24.2. They are
responsible.
Keywords: Pectobacterium
chrysanthemi; DNA; PCR; banana; wilt.
Recibido: 24-12-2024
Aceptado: 25-02-2025
INTRODUCCIÓN
Durante las últimas décadas, el banano se ha
consolidado como el cuarto cultivo más significativo a nivel global, superando
al maíz, el trigo y el arroz en más de 120 naciones (Jones, 2000; Heslop &
Schwarzacher, 2007).
Desde hace mucho se ha observado como el
Perú posee ventaja contra otros países europeos, ya que cuenta con muchos años
de experiencia de pro-ducción y comercialización de bananos orgánicos
certificados (Ccaccya & Huamán, 2020). Sin embargo, diversas enfermedades
provocadas por hongos, bacterias y virus afectan frecuentemente este cultivo
(Ancajima & Cortéz, 2022). Siendo la pudrición blanda una de las
enfermedades más importantes y comunes, especialmente en las regiones
tropicales (Martinez et al., 2020; Hugouvieux et al., 2014). La enfermedad es
causada por Pectobacterium chrysanthemi, una bacteria que ataca el
pseudotallo y el rizoma al secretar enzimas pectinolíticas que degradan los
pectatos de calcio de la lámina media de las paredes celulares produciendo la
maceración del tejido y en la liberación de un olor desagradable durante la
descomposición (Hugouvieux et al., 2014; Loganathan et al., 2019).
En ese contexto, para el futuro se prevén tempe-raturas
más elevadas y alteraciones en los patrones climáticos debido al cambio
climático, lo cual podría dar lugar a sequías e inundaciones que faciliten la
propagación de enfermedades y plagas que afectan los cultivos (Maletta, 2019),
ya que los vientos y las precipitaciones pueden desplazar a los patógenos y
restos de vegetación enferma a distancias significativas (Hernández, 2024), lo
que perjudica la producción agrícola a largo plazo.
Desde hace mucho se han desarrollado
diversas estrategias para controlar las enfermedades de las plantas, utilizando
disciplinas como la microbio-logía y la biotecnología molecular (Suárez &
Peñaranda, 2022; Cordova et al., 2021), que, de forma individual o combinada,
permiten identificar el agente causante y la enfermedad asociada, así como
determinar tratamientos adecuados a través de métodos de detección rápida y
específica. Asimismo, facilitan la identificación de los geno-tipos de las
cepas circulantes mediante técnicas de caracterización molecular y filogenética
(Salinas et al., 2020; Martínez et al., 2018).
Actualmente, entre el 2023 y 2024, Perú ha
experi-mentado varios fenómenos climáticos significa-tivos como el ciclón Yaku
(El Comercio, 2023), El Niño Costero (SENAMHI, 2024a), El Niño Global (El
Peruano, 2023), y recientemente un escenario de La Niña (SENAMHI, 2024b). Por
tanto, la comprensión de cómo Pectobacterium chrysan-themi se comporta
bajo condiciones climáticas variables y/o extremas antes, durante o después, es
crucial para anticipar y mitigar brotes, protegiendo así la producción
agrícola. En ese contexto, su caracterización molecular y filogenética permite
identificar las cepas circulantes y su diversidad genética, facilitando las
estrategias de manejo mediante la vigilancia epidemiológica de acuerdo con las
cepas nativas o introducidas.
En virtud de lo anteriormente descrito, el
objetivo del presente estudio fue determinar la dispersión, realizar la
caracterización molecular y filogenética de Pectobacterium chrysanthemi
en el marco de fenómenos climáticos en Perú, 2024.
Diseño y área de estudio
La investigación es de tipo
transversal, diseño no experimental, enfoque cualitativo y nivel descrip-tivo.
Se ha realizado en una parcela agrícola ubicada a los 92 m.s.n.m
(Lat.-5.067112º/Long.-80.126062º) en la costa norte de Perú, provincia de
Chulucanas, departamento de Piura.
Dispersión
El 4 y 30 de marzo de 2024,
se realizaron inspec-ciones sanitarias considerando la temperatura ambiental y
humedad relativa en el área de estudio. Durante cada inspección, se examinó el
estado fitosanitario de la parcela, notando la presencia de hojarasca en el
suelo, la duración de riego del cultivo y la presencia de plantas afectadas por
pudrición blanda. En la primera (4 de marzo) y segunda (30 de marzo)
inspección, los bananos se clasificaron con los siguientes códigos: “SE” (sin
enfermedad), “EI” (enfermedad en etapa inicial) representado por pseudotallos con
síntomas de necrosis y tejido blando, y “EA” (enfermedad en etapa avanzada)
representado por pseudotallos con síntomas de necrosis, pudrición acuosa y
hojas marchitas. Para estudiar la dispersión del fitopatógeno y/o la enfermedad
que causa, en la primera inspección se identificó bananos “SE” y “EI” en 100
metros a la redonda de un banano “EA”. Luego, en la segunda inspección se
evaluó si los bananos desarrollaron otros síntomas o no. Todas las plantas
fueron codificadas y georreferenciadas con estacas en el suelo para su
análisis. La temperatura y humedad relativa ambiental se midió con un
termohigrómetro modelo SH-110 de la marca Boeco Germany.
Obtención del material
vegetal
Se colectó material vegetal de una planta de
banano clasificado como EA. Se utilizaron hojas Nº 10 de bisturis estériles
para realizar incisiones de 20 a 30 cm con una disección longitudinal de 10 a
15 cm en la capa externa del pseudotallo. Luego, el material vegetal
diseccionado se colocó en bolsas ziploc estériles e inmediatamente después, se
trasladaron al laboratorio de Fitopatología del Departamento de Sanidad Vegetal
de la Universidad Nacional de Piura (UNP).
Aislamiento de bacterias
Trozos de material vegetal del pseudotallo
se colocaron en tubos de ensayo con 5 mL de agua destilada estéril y se dejó
reposar por 10 min para la difusión de bacterias.Transcurrido el tiempo, la
solución adquirió un aspecto lechoso en donde se insertó un asa de Kolle
flameada con la que se hizo siembras por estrías en placas Petri con agar
nutritivo (AN), que luego se incubaron invertidas a 28±1 °C por 48 h y en
oscuridad (French, 1980). En los aislados obtenidos, se evaluaron
características morfológicas como el color, forma, tamaño, elevación y textura
(Skerman, 1960), con las que se consideraron como presuntivos.
Diagnóstico
El protocolo de detección desarrollado por
Maldonado et al. (2024) se utilizó para llevar a cabo el diagnóstico de la
pudrición blanda. Dicho protocolo se basa en una prueba de patogenicidad con Solanum
tuberosum y un ensayo de reacción en cadena de la polimerasa (PCR) que
amplifica en 171 pb, el espaciador transcrito interno (ITS) del ADNr 16S/23S a
partir del ADN extraído de los aislados presuntivos para Pectobacterium chry-santhemi.
Se utilizaron los cebadores diseñados por Li et al. (2013): LF:
5-TTCGTCTAGAGGCCCAGGAC-3 y LR: 5-TCAGCTTGTTCCGGATTGTT-3. El ciclaje con sus
condiciones térmicas fueron una desna-turalización inicial de 98 °C por 4
minutos, seguido de 35 ciclos con 95 °C por
30 segundos para la desnaturalización, 60 °C por 45 segundos para hibridar, 72 °C
por 1 minuto para la extensión, una post-extensión de 72 °C por 5 minutos y una
temperatura de conservación de 4 °C por hasta 24 h.
Secuenciación de ADN
Los productos PCR (amplicones) obtenidos del
diagnóstico fueron secuenciados mediante la tec-nología de Sanger doble cadena
por la empresa Macrogen (https://dna.macrogen.com/). Poste-riormente,
las secuencias de las bases nitrogenadas del gen se analizaron mediante
herramientas bioinformáticas.
Caracterización molecular
El programa bioinformático de análisis de
genética evolutiva molecular MEGA versión 11 (https://www.megasoftware.net/) se utilizó para analizar
las secuencias del gen de interés de cada cepa. Se llevó a cabo el alineamiento
para determinar las secuencias consenso utilizando las lecturas directa e
inversa del gen. Para cada cepa realizó la asignación taxonómica de la especie
utilizando la herramienta de búsqueda de alineación local básica (BLAST, por
sus siglas en inglés https://blast.ncbi.nlm.nih.gov/Blast.cgi) mediante mapeo de
similitudes con secuencias de genes disponibles en el banco de genes del Centro
Nacional para la Información Biotecnológica (NCBI, por sus siglas en inglés).
Las secuencias consenso que presentaron similitud del 100% se ingresaron y
procesaron en el programa DNAsp v5. Finalmente, se obtuvo un archivo de texto
con los polimorfismos, su ubicación y los haplotipos que resultaron.
Filogenética
Utilizando las similitudes halladas en el
banco de genes, se realizó un análisis evolutivo constru-yendo un árbol
filogenético utilizando el método de máxima verosimilitud (Maximum Likelihood
tree, en inglés) y el modelo de 3 parámetros de Tamura con 1000 réplicas de
arranque disponibles en MEGA 11. Como grupo externo se consideró la secuencia
AF232678.1
Dispersión
Se aislaron dos cepas (cepa 2 y cepa 4)
obtenidas del tejido de banano enfermo que se recolectó. Así mismo, durante la
primera y segunda inspección sanitaria, se identificó la acumulación de
hojarasca en el suelo, así como un régimen de riego intensivo aplicado cada siete
días. Se observó la progresión de la
enfermedad, evidenciando que Pectobacterium chrysanthemi se dispersó
desde plantas de banano enfermas a plantas sanas en un período de 26
días, abarcando un radio de 100 m. Esto se infiere debido a que los bananos
clasificados como "EI" comenzaron a manifestar síntomas de
"EA", mientras que tres ejemplares catalogados como "SE" presentaron
síntomas de "EI" (Figura 1). Las temperaturas ambientales registradas
durante la primera y segunda inspección fueron de 27,9 °C y 31,5 °C,
respectivamente.
Pectobacterium chrysanthemi es una bacteria
fitopatógena perteneciente a la familia Entero-bacteriaceae, reconocida por su
capacidad para causar pudrición blanda en diversas plantas, especialmente en
cultivos de banano y papa. Esta bacteria se caracteriza por su habilidad para
secretar enzimas pectinolíticas, como pectinasas, que degradan los pectatos de
calcio de la lámina media presentes en las paredes celulares de las plantas, lo
que provoca la descomposición del tejido vegetal (Hugouvieux et al., 2014;
Loganathan et al., 2019; Martinez et al., 2020).
Conocer la interacción de los patógenos con
el medio ambiente es fundamental para desarrollar métodos más efectivos de
control y mitigación, asegurando así la sostenibilidad de la producción de un
cultivo (Vera et al., 2024). Según Hugouvieux et al. (2014), los cultivos de
banano infectados por cepas de Pectobacterium bajo condiciones natura-les
presentan síntomas de necrosis y pudrición blanda en el pseudotallo, que son
consecuencia de la degradación enzimática de la pectina en el tejido vascular.
Aguilar et al. (2021) demostraron que, en condiciones controladas, tras la
inoculación de pseudotallos de banano con Pectobacterium, los síntomas
comienzan a manifestarse a los 6 días. Sin embargo, a los 16 días se observa la
descompo-sición total del pseudotallo, acompañada de un olor característico de
descomposición. Ramírez et al. (2014) evidenciaron que al inocular plántulas de
banano con una solución de Pectobacterium sp. a una concentración
aproximada de 108 UFC/ml, la enfermedad de la pudrición blanda se
desarrolla en un máximo de 45 días. Sin embargo, estos resultados solo se
obtienen bajo la aplicación de inductores de resistencia, lo que sugiere que el
tiempo de desarrollo de la enfermedad difiere en condiciones naturales.
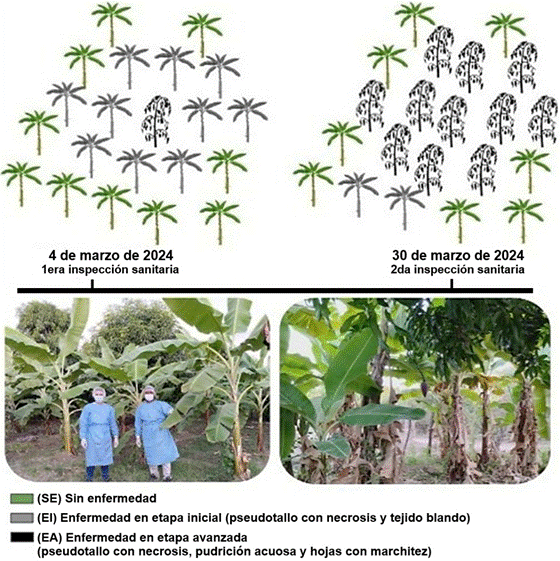
Figura 1. Infograma de dispersión y/o desarrollo de
la pudrición blanda, enfermedad causada.
No obstante, la dispersión de la pudrición
blanda y/o Pectobacterium chrysanthemi que hallamos en la parcela
analizada, puede atribuirse a factores abióticos, tales como la acumulación de
hojarasca en el suelo, la práctica de un riego intensivo, así como temperaturas
ambientales de 27,9 °C a 31,5 °C que observamos en este estudio. Estas condi-ciones
podrían considerarse propicias para la aparición y dispersión de la pudrición
blanda. Al respecto, Martínez & Müller (2023) señalan que la propagación de
enfermedades y plagas está pro-fundamente influenciada por condiciones clima-ticas
extremas, como sequías e inundaciones. Ormeño (2023) indica que algunas
especies de plantas son susceptibles a la adquisición de enfermedades debido a
cambios en la humedad del suelo y la temperatura. De acuerdo con Hernández
(2024), tanto los vientos como las precipitaciones, juegan un papel crucial en
la dispersión de restos de vegetación infectada. Medina et al. (2024), seña-lan
que normalmente, los agentes fitopatógenos ingresan a las plantas por heridas,
a través del agua, insectos y nematodos. En India, tras períodos de lluvias
intensas, se ha registrado la aparición de bananos con síntomas de necrosis
blanda, atribu-yéndose la causa a Pectobacterium chrysanthemi (Nagrale
et al., 2013). Este escenario es factible debido a que, en el contexto agrícola,
algunas bacterias se propagan a través del agua de riego, utilizándose como un
medio de transmisión (Obregón, 2016). Según Snehalatharani et al. (2010) la
infección por bacterias del género Pectobacterium en plantas de banano
provoca con frecuencia el colapso del pseudotallo, que se quiebra en la zona
más descompuesta del tallo, particularmente cerca del nivel del suelo. En este
contexto, se favorecen las infecciones y la dispersión de enfermedades a través
de factores abióticos, como la acumulación de hojarasca en el suelo y la
práctica de un riego intensivo, junto con una temperatura y humedad que brindan
condiciones ideales.
Caracterización molecular y
filogenética
Como resultado del secuenciamiento de la
región espaciadora intergénica ADNr 16S/23S (171 pb) de las cepas analizadas (cepa
2 y cepa 4) y su posterior alineamiento, se obtuvieron secuencias consen-suadas
de 129 pb, que, al ser comparadas con las disponibles en el banco de genes del
NCBI, presentaron similitud con un fragmento de AF232682.1. Este fragmento
corresponde a la secuencia completa de la región espaciadora intergénica
16S-23S y del gen ARNt-Glu de la cepa KACC 10163 de Pectobacterium
chrysanthemi, que posee un tamaño total de 354 pb (Figura 2). Entre los
fragmentos de 129 pb que se analizaron, se halló variabilidad entre las bases,
que, tras su caracterización molecular por bioinformática, se reflejó en 101
sitios monomórficos, 28 sitios polimórficos y 34 mutaciones, representadas en 3
haplotipos (Hap-1, Hap-2, Hap-3).
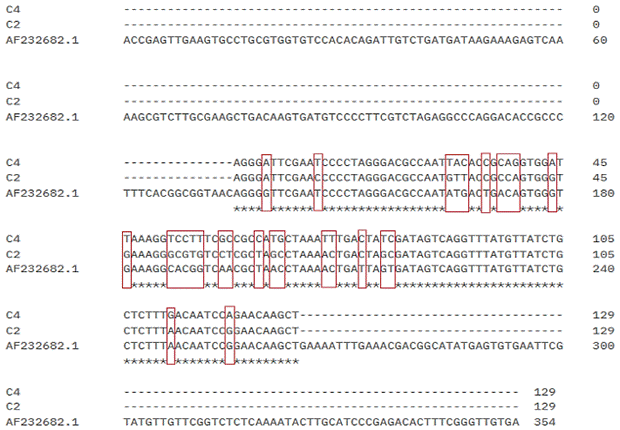
Figura 2. Alineamiento
de la región espaciadora intergénica ADNr 16S/23S de las cepas 2 y 4, y la
secuencia AF232682.1. Los guiones (-) representan espacios (gaps), los
asteriscos (*) indican posiciones idénticas, y los recuadros en rojo señalan
variaciones de bases nitrogenadas entre las posiciones correspondientes de cada
secuencia.
La secuencia de AF232682.1 (Hap-1) presenta
un solo haplotipo, al igual que las secuencias de cepa 2 (Hap-2) y cepa 4
(Hap-3). En color amarillo se señala una base nitrogenada distinta en una posi-ción,
mientras que en color verde se representan tres bases nitrogenadas diferentes
para una misma posición. Estos resultados sugieren la circulación de dos
genotipos de Pectobacterium chrysanthemi en el norte de Perú (Tabla 1).
Por otra parte, en lo que respecta a las
relaciones evolutivas entre los haplotipos identificados de las secuencias ITS-ADNr
16S/23S que se analizaron, se ilustra un árbol filogenético elaborado mediante
el método de máxima verosimilitud. Según la hipótesis evolutiva derivada, se
aprecian dos clados, clado I (secuencias de cepa 2 y cepa 4) y clado II
(secuencia de AF232682.1.). El clado I se sitúa en una rama que emerge de otra
compartida con el clado II. Según Ragavil et al. (2019), la región ITS-ADNr 16S/23S
es útil para la detección de Pectobacterium carotovorum subsp. carotovorum
y su análisis filogenético en comparación con otras secuencias disponibles.
Esto es respaldado por autores como Kwon et al. (2000), quienes trabajaron con
la región ITS-ADNr 16S/23S para la diferenciación de Pectobacterium
carotovorum subsp. carotovorum y Pectobacterium chrysanthemi. Sin
embargo, Basim et al. (2019) sugieren que, como alternativa para la
identificación molecular de bacterias del género Pectobacterium, los
genes pel y 16S pueden ser utilizados.
Tabla 1
Características moleculares del ITS-ADNr 16S/23S
de las secuencias analizadas
|
Posiciones
|
5
|
12
|
30
|
31
|
32
|
35
|
37
|
38
|
39
|
44
|
46
|
52
|
53
|
54
|
|
Hap-3
|
A
|
T
|
T
|
A
|
C
|
C
|
C
|
A
|
G
|
A
|
T
|
T
|
C
|
C
|
|
Hap-2
|
A
|
C
|
G
|
T
|
T
|
C
|
C
|
C
|
A
|
G
|
G
|
G
|
C
|
G
|
|
Hap-1
|
G
|
T
|
A
|
T
|
G
|
T
|
A
|
C
|
A
|
G
|
G
|
C
|
A
|
C
|
|
Posiciones
|
55
|
56
|
59
|
60
|
64
|
66
|
67
|
73
|
74
|
78
|
81
|
82
|
112
|
120
|
|
Hap-3
|
T
|
T
|
G
|
C
|
C
|
T
|
G
|
T
|
T
|
C
|
T
|
C
|
G
|
A
|
|
Hap-2
|
T
|
G
|
C
|
T
|
T
|
G
|
C
|
A
|
C
|
C
|
G
|
C
|
A
|
G
|
|
Hap-1
|
G
|
G
|
A
|
A
|
T
|
A
|
C
|
A
|
C
|
T
|
G
|
T
|
A
|
G
|

Figura 3. Árbol filogenético basado en el
método de máxima verosimilitud y el modelo 3 parámetros de Tamura con un valor
bootstrap de 1000 réplicas. Muestra la relación existente entre la secuencia de
los haplotipos del ITS-ADNr 16S/23S de Pectobacterium chrysanthemi aislado
de banano orgánico (Musa sp.) de Perú y la secuencia AF232682.1
disponible en el banco de genes del NCBI.
Rafael et al. (2022) han realizado una
investigación empleando el gen 16S en Perú, con el que han logrado la
identificación molecular de Pectobacterium carotovorum y Pectobacterium
chrysanthemi. Kwon et al. (2000) mencionan que la secuencia AF232682.1 se
ha obtenido mediante la amplificación de cebadores diseñados a partir de
fragmentos de ADN de 474 a 569 pb y de 354 a 459 pb, amplificados con dos
conjuntos de cebadores que permiten diferenciar diversas especies del género Pectobacterium
gracias a su elevada diversidad nucleotídica, lo cual difiere con el marcador
universal 16S.
En este marco, los hallazgos de nuestro
estudio no pueden ser completamente interpretados respecto a la diversidad genética
de Pectobacterium chrysanthemi en Perú, dado que se trata de la primera
investigación que emplea este marcador específico (ITS-ADNr 16S/23S). No
obstante, se ha logrado establecer una diferenciación genotípica ya que se
utilizó una región de ADN con alta tasa de mutación, pero con suficiente
conservación como para definir características moleculares distintivas de la
especie, como hemos demostrado en este estudio.
Además, el tamaño del fragmento amplificado
en nuestra investigación coincide con lo reportado por Li et al. (2013), cuyos
cebadores fueron utilizados para este estudio. Por tanto, se resalta la
limitación de publicaciones e investigaciones sobre este marcador. No obstante,
se han hallado dos genotipos (cepa 2 y cepa 4) nuevos, que se encuentran
circulando en el norte de Perú, los cuales se han designado como PN24.1. y
PN24.2.
En este estudio se encontró que Pectobacterium
chrysanthemi se dispersó de plantas de banano enfermas a sanas en 26 días,
abarcando un radio de 100 metros. Factores como la presencia de hoja-rasca en
el suelo, el riego intensivo y temperaturas ambientales de 27,9 °C a 31,5 °C,
podrían estar influyendo negativamente en la infección por pudrición blanda.
Estos factores son relevantes en el contexto agrícola, ya que suelen contribuir
a la infección de cultivos por bacterias fitopatógenas, y al desarrollo y
dispersión de las enfermedades asociadas. Asimismo, la pudrición blanda
analizada en este estudio se atribuyó a dos genotipos de Pectobacterium
chrysanthemi, PN24.1. y PN24.2, reportados por primera vez para Perú. Los
esfuerzos por comprender la dispersión y características moleculares de Pectobacterium
chrysanthemi en condiciones abióticas específicas permitieron identificar
dos genotipos, por lo cual sugerimos continuar con estudios adicionales, en los
que se analice múltiples parcelas en diversas zonas geográficas y bajo
diferentes escenarios climáticos, así como el uso de marcadores mole-culares
complementarios para ampliar el cono-cimiento sobre estos genotipos circulantes
y la identificación de otros que aún se desconocen. Además, es crucial realizar
estudios experimen-tales en condiciones de laboratorio en los que se determine
las temperaturas específicas que influ-yen en el desarrollo de síntomas de la
pudrición blanda. Estos datos permitirán estimar el tiempo en que se desarrolla
la enfermedad y su posterior dispersión según el genotipo y la agresividad que
posea, facilitando la implementación de medidas adecuadas para el control de la
pudrición blanda, ya sea a través de charlas informativas para agricultores o
estrategias sanitarias por parte de los organismos gubernamentales competentes.
Al Departamento de Sanidad
Vegetal de la Universidad Nacional de Piura por permitirnos realizar los aisla-mientos
bacterianos en sus laboratorios. Al departamento de Ciencias Biológicas de la
misma institución por facilitar el acceso a los equipos de biotecnología
molecular en el desarrollo de esta investigación. A Zinadine Zidanne
Barranzuela Carrasco y José Javier Benites Alamo, estudiantes de la Universidad
Sedes Sapientiae que brindaron apoyo en las inspecciones sanitarias, colecta y
traslado de material vegetal de banano.
REFERENCIAS
BIBLIOGRÁFICAS
Aguilar-Anccota, R., Ruiz, W., Morales-Pizarro, A.,
Rafael-Rutte, R., Tirado-Lara, J., Saucedo-Bazalar, M., & Teodor, K.
(2021). Pudrición blanda en el pseudotallo de banano orgánico (Musa sp):
sintomatología, caracterización cultural y bioquímica, patogenicidad y
alternativas de manejo. Scientia Agropecuaria, 12(4), 571-578. http://dx.doi.org/10.17268/sci.agropecu.2021.061
Ancajima, A., & Cortéz, H. (2022). Enfermedades que
afectan la agroexportación de banano orgánico (Musa paradisiaca) en el
Valle Del Chira, 2021. [Tesis de bachillerato, Universidad Nacional de
Frontera].
Basim, H., Basim, E., Bakİ, D., & Turgut, A.
(2019). Enfermedad de la pudrición húmeda del banano (Musa sp.) causada
por Pectobacterium carotovorum subsp. carotovorum en Turquía. Revista
canadiense de patología vegetal, 41(2), 174-187.
Ccaccya Díaz, K. I., & Huamán Tirado, M. A. (2020).
Efectos de la certificación de comercio justo en el departamento de Piura con
respecto a las exportaciones de banano orgánico con partida arancelaria
0803.90. 11.00 a los Países Bajos durante el periodo de 2013-2018. [Tesis de
licenciatura, Universidad Peruana de Ciencias Aplicadas].
Córdova, L., Zelaya, L., Ávila, N., Valenzuela, V.,
Cortés, N., Parra, F., & Santos, S. (2021). Potencial
de las ciencias ómicas en la bioprospección de agentes microbianos de control
biológico: el caso de la agro-biotecnología mexicana. Revista mexicana de
fitopatología, 39(1), 147-184. https://doi.org/10.18781/r.mex.fit.2009-3
El Comercio. (2023). Ciclón Yaku 2023 en el Perú y
Ecuador: cómo se origina y por qué afecta estos países. El Comercio.
El Peruano. (2023). Niño Global comenzó en el mundo,
pero aún no en Perú, ¿cuándo se sentirán sus efectos?. El Peruano.
French, E., & Hebert, T. (Ed.). (1980).
Métodos de Investigación Fitopatológica. IICA de Costa Rica. (pp. 236)
Hernández, R. C. (2024). Evaluación de la actividad
antifúngica in vitro de extractos totales de dos especies vegetales Marco y
Molle frente a uno de los hongos patógenos causantes de Phytophthora infestans
en Tomate Riñón. [Tesis de licenciatura, Pontificia Universidad Católica del
Perú.].
Heslop-Harrison, J., & Schwarzacher, T. (2007).
Domestication, genomics and the future for banana. Annals of Botany, 100(5),
1073-1084. https://doi.org/10.1093/aob/mcm191
Hugouvieux-Cotte-Pattat, N., Condemine, G., &
Shevchik, V. (2014). Bacterial pectate lyases, structural and functional
diversity. Environmental Microbiology Reports, 6(5), 427-440. https://doi.org/10.1111/1758-2229.12166
Ormeño, S. (Ed.). (2023). Introducción sintética a las
enferme-dades de las plantas. Ormeño, Madrid: Universidad Politécnica de
Madrid.
Jones, D. R. D. (Ed.). (2000). Enfermedades del banano,
abacá y enset. ABI, Wallingford (pp. 544).
Kwon, S., Myung, I., & Go, S. (2000). Detección de Pectobacterium
chrysanthemi utilizando cebadores de PCR específicos diseñados a partir de
la región espaciadora intergénica del ARNr 16S-23S. La Revista de Patología
Vegetal, 16(5), 252-256.
Kwon, S. W., Cheun, M. S., Kim, S. H., & Lim, C. K.
(2000). Phylogenetic analysis of Pectobacterium species
using the 16S-23S rRNA intergenic spacer regions. The Plant Pathology
Journal, 16(2), 98-104.
Li, P., Lin, B., Shen, H., & Pu, X. (2013).
Species-specific detection of Dickeya sp. (Pectobacterium
chrysanthemi) in infected banana tissues, soil and water. African Journal
of Biotechnology, 10(74), 16769-16773. https://doi.org/10.5897/AJB11.1248
Loganathan, M., Thangavelu, R., Padmanaban, B., & Uma, S. (2019).
Status of rhizome and pseudostem wet rot diseases of banana. International
Journal of Current Microbiology and Applied Sciences, 8, 764–771. https://doi.org/10.20546/ijcmas.2019.805.090
Maldonado, E. A., Vigil Correa, A., Zurita Chinguel, L.
G., Mendoza Rendón, R., Torres, M. D., Saavedra Rios, C. Y., Villar, D., Cubas
Zúñiga, C. E., Peña Zárate, M. C., Vilchez Estrada, K. N. & Ruiz Polo, A.
A. (2024). Patogenicidad de Pectobacterium chrysanthemi en Solanum
tuberosum y PCR del espaciador transcrito interno (ITS) del ADNr 16S/23S en
aislados de Musa sp. del norte del Perú. Manglar, 21(3), 299-304.
https://doi.org/10.57188/manglar.2024.032
Martínez Quijón, F. I., & Müller Mora, C. A. (2023).
El impacto del cambio climático en el uso de los suelos agrícolas de la comuna
de Parral en el periodo 2002-2022. [Tesis de licenciatura, Universidad del
Bío-Bío – Chile].
Martínez-Solórzano, G., Rey-Brina, J., Pargas-Pichardo,
R., & Manzanilla, E. (2020). Marchitez por Fusarium raza tropical 4:
Estado actual y presencia en el continente americano. Agronomía
Mesoamericana, 31(1), 259-276. http://dx.doi.org/10.15517/am.v31i1.37925.
Martínez, H. F., Chávez-Arteaga, K., Guato-Molina, J.,
Peñafiel-Jaramillo, M., & Mestanza-Uquillas, C. (2018). Bacterias
fluorescentes productoras de metabolitos antagónicos de cultivares nativos de Musa
sp. y su diversidad filogenética al gen ARNr 16S. Ciencia y Tecnología,
11(2), 17-29.
Medina, N. B., Sagnay-Ramírez, B., & Leon-Reyes, A.
(2024). Memorias del VI Simposio en Fitopatología, Control Biológico e
Interacciones Planta-Patógeno. Archivos Académicos
USFQ, (51). https://doi.org/10.18272/archivosacademicos.vi51.3308
Nagrale, D., Borkar, S., Gawande, S., Mandal, A., &
Raut, S. (2013). Characterization of a bacterial collar and rhizome rot of
banana (Musa paradisiaca) caused by strains of Erwinia chrysanthemi
pv. paradisiaca. Journal of Applied and Natural Science, 5(2),
435-441. https://doi.org/10.31018/jans.v5i2.349
Obregon, V., Ibanez, J., & Lattar, T. (Ed.). (2016).
Guía para la identificación de las enfermedades de pimiento en invernadero.
Bellavista, Argentina. INTA. (pp. 44).
Rafael-Rutte, R., Zavala, L., A. Maldonado, E.,
Aguilar-Anccota, R., et al. (2022). Characterization of
rhizome and pseudostem wet rot of organic banana (Musa sp.) in Piura,
Perú. Chilean Journal of Agricultural & Animal Sciences, 38(2),
176-188. https://doi.org/10.29393/CHJAA38-17CRRC70017
Ragavil, G., Muthamilan, M., Nakkeeran,
S., Kumaravadivel, N., Sivakumar, U., & Suganthi, A. (2019). Molecular
detection of the causative agent of soft rot (Pectobacterium carotovorum
subsp carotovorum) in banana (Musa sp.) International Journal
of Current Microbiology and Applied Sciences, 8(11), 1854-1868. https://doi.org/10.20546/ijcmas.2019.811.218
Ramírez, G., Jaraba, B., & Buriticá,
PE. (2014). Manejo de la pudrición acuosa del pseudo-tallo (Dickeya
sp.) en plátano (Musa sp.) bajo condiciones de invernadero. Agronomía
Costarricense, 38(2), 83-92. https://doi.org/10.15517/rac.v38i2.17277
Suárez-Contreras, L. Y., &
Peñaranda-Figueredo, F. A. (2022). Identifi-cación molecular de hongos
filamentosos y su potencial biotec-nológico. Biotecnología en el sector
agropecuario y agroindustrial, 20, 194-206. https://doi.org/10.18684/bsaa.v20.n1.2022.1914
Salinas, L. I., Torre Hernández, M. E. D.
L., Aguirre Garrido, J. F., & Ramírez Saad, H. C. (2020). Caracterización
molecular de bacterias rizosféricas asociadas a Echinocactus platyacanthus en
invernadero y silvestres. Revista mexicana de ciencias agrícolas, 11(3),
531-542. https://doi.org/10.29312/remexca.v11i3.2017
SENAMHI (2024a). El Niño Costero
2023-2024 fue el más intenso de los últimos 20 años en el Oeste de Sudamérica. Servicio
Nacional de Meteorología e Hidrología del Perú.
SENAMHI. (2024b). Condiciones actuales
de El Niño/La Niña: No activo. Servicio Nacional de Meteorología e
Hidrología del Perú.
Skerman, V. B. (1960). A
guide to the identification of the genera of bacteria. Academic Medicine, 35(1),
92.
Snehalatharani, A., & Khan, A. (2010). Biochemical
and physiological characterisation of Erwinia species causing tip-over
disease of banana. Archives Of Phytopathology and Plant Protection, 43(11),
1072-1080. https://doi.org/10.1080/03235400802285422
Vera-Morales, M., Castañeda-Ruiz, R. F., Sosa, D.,
Arias-Vega, C., Quevedo, A., & Ratti, M. F. (2024). Compuestos
bioactivos de bacterias y hongos en el control de nematodos fitopatógenos:
mecanismos de acción, interacciones y aplicaciones. Scientia Agropecuaria,
15(1), 143-157. https://doi.org/10.17268/sci.agropecu.2024.011