Variabilidad
genética en cultivares de Musa spp. mediante ADN polimórfico amplificado
aleatoriamente (RAPD)
Genetic
variability in Musa spp. cultivars using randomly amplified polymorphic
DNA (RAPD)
Segundo Melecio
Garcia-Garcia1, 2; Jalmer Fidel Campaña-Olaya1; César
Augusto Mogollón-Farias2
Joel Michel Riojas-Gonzales2;
John Henrry Rimaycuna-Ramirez1; Karla Lucia Seminario-Juárez1
Archi Alejandro Ruiz-Polo2;
José Stalyn Córdova Campos2; Erick Antonio Suarez-Peña1
1. Facultad de Ciencias
Agrarias, Universidad Nacional de Tumbes. Ciudad Universitaria, Av.
Universitaria S/N, Tumbes, Perú.
2. Estación Experimental
Agraria Los Cedros. Dirección de Recursos Genéticos y Biotecnología. Instituto
Nacional de Innovación Agraria (INIA), Panamericana Norte Kilómetro 12, Tumbes.
Perú.
*
Autor corresponsal: Melecio.leo2000@gmail.com (S. M.
Garcia-Garcia).
ORCID
de los autores:
S. M. Garcia-Garcia: https://orcid.org/0009-0003-6300-9221 K. L.
Seminario-Juárez: https://orcid.org/0009-0001-7059-8365
J. F. Campaña-Olaya: https://orcid.org/0000-0002-0804-1208 A. A.
Ruiz-Polo: https://orcid.org/0009-0005-1273-2625
C. A. Mogollón-Farias: https://orcid.org/0009-0000-5390-9208 J. S.
Córdova Campos: https://orcid.org/0000-0002-5891-4679
J. M. Riojas-Gonzáles: https://orcid.org/0009-0007-9966-9554 E. A.
Suarez-Peña: https://orcid.org/0000-0003-0137-8251
J. H. Rimaycuna-Ramirez: https://orcid.org/0000-0002-2767-9733
RESUMEN
El norte de Perú presenta condiciones
abióticas favorables para la producción de banano. Sin embargo, la selección
artificial ha generado cultivares que, en ocasiones, son difíciles de
distinguir fenotípicamente, lo que limita la selección de especímenes con resistencia
a plagas y alta calidad productiva. Por esta razón, se recurre al análisis del
material genético para identificar los genotipos presentes. Como objetivo se
determinó la variabilidad genética de 9 cultivares de Musa spp. mediante
ADN polimórfico amplificado aleatoriamente (RAPD). Se seleccionó la
tercera hoja de cada planta de banano perteneciente a un cultivar, y utilizando
tijeras estériles, se extrajo una porción que luego se disectó en pequeñas
secciones. Posteriormente, se procedió a la extracción de ADN genómico, seguida
de una amplificación mediante PCR-RAPD utilizando los marcadores OPA-1, OPA-2,
OPA-3, OPA-4, A-01, OPC-2 y OPC-15, con el fin de identificar la variabilidad
genética a través polimorfismos. Finalmente, el análisis del número de bandas
amplificadas, polimórficas y sus respectivos porcentajes se realizó mediante el
software R-Studio con el que se obtuvo un dendrograma como producto. En los
cultivares IC2, Valery, Montecristo, Cavendish Gigante, Red Dacca, Williams,
Gran enano, Gros Michel y M. paradisiaca cv. Zapatito, se amplificaron
un total de 76 bandas, de las cuales 41 fueron polimórficas. El dendrograma
reveló una relación genética estrecha entre cinco cultivares (Gran Enano,
Williams, Montecristo, IC2 y Valery) debido a la similitud en las bandas
polimórficas. En cambio, los cultivares Gros Michel, Red Dacca y Zapatito
presentaron una diferenciación genética significativa, sin agruparse, debido a
la ausencia o mayor cantidad de bandas polimórficas en cada uno. La evidencia
sugiere que en el norte del Perú existe una notable diversidad genética entre
los cultivares de Musa spp., lo que representa un recurso estratégico
para el mejoramiento genético. Por ello, se plantea la integración sistemática
de herramientas moleculares en los procesos de selección y certificación, con
el objetivo de potenciar la sostenibilidad y competitividad del cultivo en la
región.
Palabras clave: Cultivares;
Polimorfismo; Diversidad genética; PCR-RAPD; Marcador molecular.
ABSTRACT
The north of Peru presents favorable abiotic
conditions for banana production. However, artificial selection has generated
cultivars that are sometimes difficult to distinguish phenotypically, limiting
the selection of specimens with resistance to pests and high productive
quality. For this reason, genetic material analysis is used to identify the present
genotypes. The objective was to determine the
genetic variability of 9 Musa spp. cultivars using Random Amplified Polymorphic
DNA (RAPD). The third leaf from each banana plant belonging to a cultivar was
selected, and using sterile scissors, a portion was extracted and then
dissected into small sections. DNA extraction was then performed, followed by
amplification using PCR-RAPD with markers OPA-1, OPA-2, OPA-3, OPA-4, A-01,
OPC-2, and OPC-15, to identify genetic variability through polymorphism.
Finally, the analysis of the number of amplified polymorphic bands and their
respective percentages was performed using R-Studio software, which produced a
dendrogram. A total of 76 bands were amplified across the cultivars IC2,
Valery, Montecristo, Cavendish Gigante, Red Dacca, Williams, Gran Enano, Gros
Michel, and M. paradisiaca cv. Zapatito, of which 41 were polymorphic. The
dendrogram revealed a close genetic relationship between five cultivars (Gran
Enano, Williams, Montecristo, IC2, and Valery) due to the similarity in their
polymorphic bands. In contrast, the cultivars Gros Michel, Red Dacca, and
Zapatito showed significant genetic differentiation, not clustering together,
due to the absence or greater number of polymorphic bands in each. Evidence
suggests that there is significant genetic diversity among Musa spp.
cultivars in northern Peru, which represents a strategic resource for genetic
improvement. Therefore, the systematic integration of molecular tools into
selection and certification processes is proposed, with the goal of enhancing
the crop's sustainability and competitiveness in the region.
Keywords: Cultivars; Polymorphism;
Genetic diversity; PCR-RAPD; Molecular marker.
Recibido: 18-02-2025.
Aceptado: 08-06-2025
INTRODUCIÓN
El banano se destaca como la fruta de mayor
consumo a nivel global, siendo considerado un cultivo estratégico para la
seguridad alimentaria en diversas naciones y representando una de las
principales fuentes de exportación (Martínez & Rey, 2021; Mehendran et al.,
2022). Tal es el caso de que, en 2018, las exportaciones de banano (excluyendo
el plátano) alcanzaron un récord histórico de 19,2 millones de toneladas
(Organización de las Naciones Unidas para la Alimentación y la Agricultura
[FAO], 2022).
Sin embargo, diversas plagas y enfermedades afectan
tanto a la planta como a sus hojas en su conjunto. Entre las más relevantes se
encuentran distintas especies de nematodos que provocan afecciones en las
hojas; la Sigatoka negra, una enfermedad causada por un hongo que genera
manchas necróticas de color marrón oscuro o negro en la superficie foliar; el
picudo negro, un escarabajo que daña el pseudotallo al alimentarse de él,
debilitando la planta y Pectobacterium chrysantemi, una bacteria que
causa necrosis y pudrición acuosa en el pseudotallo Ávil et al., 2024, Martínez
et al., 2020, Vera, 2021; Paredes et al., 2024; Reyes & Ceballos, 2025;
Guamangate et al., 2024; Maldonado et al., 2024).
En Perú, este cultivo constituye una
actividad agrícola fundamental, con un impacto significativo en la economía del
país y en el sustento de numerosas familias rurales. Las principales áreas
productivas se localizan en la región norte, particularmente en los
departamentos de Piura, Tumbes, Lambayeque y Amazonas. Dichos territorios se
caracterizan por un clima cálido y la abundancia de agua, condiciones que
favorecen el desarrollo adecuado de la planta de banano, posicionándolos como
uno de los cultivos más relevantes para la exportación agrícola.
En el departamento de Tumbes, situado en la
región norte de Perú, existen localidades que son reconocidas por su alta
producción de variedades de banano debido a que poseen áreas con condiciones
edafoclimáticas favorables para la actividad agrícola de dicho fruto (Flores,
2010).
Una de las especies de bananos más representativas
en el departamento de Tumbes es Musa acuminata y Musa paradisiaca,
la cual se puede hallar con distintos cultivares. No obstante, su
diferenciación se realiza mediante métodos tradicionales basados en la
caracterización del fenotipo con la que muchas veces se presentan duplicidades
y enfoques incompletos.
Ante esta problemática, el estudio de la
diversidad genética es crucial, ya que facilita la diferenciación entre
genotipos y permite evaluar la variabilidad genética (Del médico et al., 2021),
lo cual contribuye al desarrollo económico de zonas productoras a través de la
selección de especies resistentes a plagas y con alta calidad productiva.
Además, de la identificación de genotipos de banano presentes en una ubicación
geográfica específica (Nadal et al., 2009). Por ello, es necesario realizar una
caracterización molecular complementaria a la morfológica en los cultivos de
banano, ya que permiten una identificación precisa y detallada de los
diferentes genotipos que existen (Buitrago et al., 2020).
Estudios previos de las últimas décadas
realizados en distintas partes del mundo, señalan que la aplicación de técnicas
moleculares como la RFLP (Fragmentos de restricción de longitud polimórfica) y
RAPD (ADN polimórfico amplificado aleatoriamente), son de gran apoyo en la
identificación de genotipos de banano, ya que son muy eficientes en la
detección de combinaciones genéticas artificiales (Pillay, Nwakanma, &
Tenkouano, 2000; Pillay et al., 2001; Pillay et al., 2004; Darmayani et al.,
2018).
En Perú, la técnica molecular RAPD ya se ha
aplicado en 42 muestras de 18 variedades de café, hallando un bajo grado de
diversidad genética y una agrupación discreta entre las variedades (Palomino et
al., 2014). No obstante, en bananos, la aplicación de la técnica es escasa
(Cruz et al., 2019).
Por tanto, la aplicación de técnicas
moleculares como RAPD, en cultivares de Musa spp. del departamento de
Tumbes, permitirá una identificación más precisa mediante la diversidad
genética presente en el genoma de las poblaciones de banano de esta región. Lo
cual da lugar a una diferenciación y selección de cultivares con
características agronómicas favorables, tales como resistencia a plagas y alta
productividad, contribuyendo a mejorar la competitividad en el mercado global.
Es por ello que esta investigación es clave para el desarrollo de estrategias
de conservación y mejoramiento genético del banano, promoviendo la
sostenibilidad y el crecimiento económico en el norte de Perú.
En virtud de lo anteriormente descrito, en
el presente estudio se planteó el objetivo de determinar la variabilidad
genética en cultivares de Musa spp. mediante ADN polimórfico amplificado
aleatoriamente (RAPD).
Diseño y área del estudio
Se realizó un
estudio basado en el análisis de la variabilidad genética de 9 cultivares de Musa
spp. mediante la técnica molecular de ADN polimórfico amplificado
aleatoriamente (RAPD). Los cultivares analizados corresponden a las especies de
Musa acuminata (8 cultivares) y Musa paradisiaca (1 cultivar)
situados en el departamento de Tumbes, ubicado al norte de Perú (Figura 1).
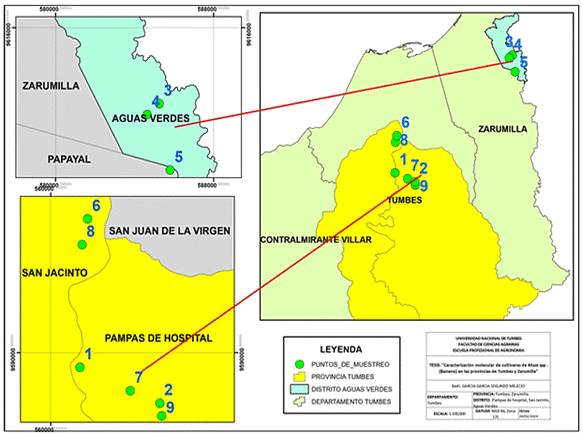
Figura 1. Ubicación geográfica de los
puntos de muestreo de los cultivares del departamento de Tumbes analizados.
Localización de
cultivares y muestreo
La localización de los cultivares se realizó
mediante recorridos por los campos del departamento de Tumbes. Las plantas madre
fueron identificadas utilizando un sistema de geoposicionamiento satelital
(Figura 1 y Tabla 1). Posteriormente, se recolectaron muestras de los
cultivares de banano, seleccionando la tercera hoja de cada planta. Se extrajo
una porción de la hoja utilizando tijeras estériles, la cual fue colocada en
bolsas Ziploc y almacenada en refrigeradores portátiles para su transporte al laboratorio
de biotecnología ambiental de la Universidad Nacional de Tumbes (UNTumbes). Las
muestras se conservaron a temperatura ambiente hasta su procesamiento.
Pretratamiento de las
muestras
Las hojas de banano
se cortaron en secciones pequeñas con el fin de facilitar su procesamiento. La
superficie foliar se desinfectó utilizando una solución de hipoclorito de sodio
al 10 % y Tween al 0,1 % durante dos minutos (Lallana & García, 2013).
Posteriormente, se eliminaron los residuos del desinfectante con agua destilada
estéril durante dos minutos adicionales. Luego, las muestras se dejaron secar
sobre papel estéril. Una vez secas, las muestras fueron congeladas con
nitrógeno líquido antes de ser liofilizadas, con el objetivo de preservar la
población de endófitos (Silva et al., 2009).
Extracción de ADN
La extracción de
ADN se realizó siguiendo las indicaciones del Mini Kit de purificación de ADN
genómico Thermo Scientific GeneJET (K0721). Al cual se añadió un paso de
maceración con nitró-geno líquido. Posteriormente, la concentración de la
molécula fue cuantificada en un espec-trofotómetro modelo ND-1000 Nanodrop.
Tabla 1
Colección de muestras de las variedades de Musa
spp. por zona en el departamento de Tumbes
|
Nº
|
Zona de muestreo
|
Coordenadas
|
Nombre común
|
Nombre científico
|
|
Este
|
Norte
|
|
1
|
Loma Saavedra
|
585248
|
9612834
|
Montecristo
|
Musa acuminata cv.
|
|
2
|
Uña de gato
|
585821
|
9609880
|
Red Dacca
|
Musa acuminata cv.
|
|
3
|
Positos
|
584624
|
9611750
|
Cavendish Gigante
|
Musa acuminata cv.
|
|
4
|
Pampas de Hospital
|
561473
|
9589296
|
IC2
|
Musa acuminata cv.
|
|
564027
|
9588146
|
Gran Enano
|
Musa acuminata cv.
|
|
5
|
Becerra
|
565523
|
9587542
|
Valery
|
Musa acuminata cv.
|
|
565620
|
9586922
|
Zapatito
|
Musa paradisiaca cv.
|
|
6
|
San Jacinto
|
561587
|
9595286
|
Gros Michel
|
Musa acuminata cv.
|
|
561866
|
9596553
|
Willians
|
Musa acuminata cv.
|
PCR-RAPD
La PCR-RAPD se realizó siguiendo las
indicaciones del proveedor del kit comercial GoTaq y empleando 5 unidades de
cebadores descritos por Chen et al. (2011) (OPA-1: 5’-CAGGCCCTTC-3, OPA-2:
5’-TGCCGAGCTG -3’, OPA-3: 5’-AGTCAGCCAC-3’, OPA-4: 5’-AATCGGGCTG-3’, A-01:
5’-CAGGCCCTTC-3’) y Padmalatha & Prasad (2007) (OPC-2: 5’-GTGAGGCGTC-3’,
OPC-15: 5’-GACGGATCAG-3’). El volumen final de reacción para el ADN de cada
cultivar fue de 24,4 uL, compuesto por 15,2 µL de agua ultra pura (AUP), 5 µL
de buffer 10X, 2 µL de MgCl2 (25 mM), 0,5 µL de dNTPs, 0,6 µL de cebadores R,
0,1 µl de Taq ADN polimerasa y 1 µl de ADN. Las condiciones térmicas y el
ciclaje consistieron en una desnaturalización inicial de 95 °C por 5 min,
seguido de 45 ciclos con 93 °C por 1 min para la desnaturalización, 36 °C por
30 s para la hibridación, 72 °C por 1 min para la extensión, y una post
extensión de 72 °C por 7 min.
Electroforesis de ADN
Los productos PCR (amplicones) obtenidos de
los 9 cultivares se visualizaron mediante electroforesis en gel de agarosa al
1,5% en solución TAE 1X (Tris-Acetato-EDTA) teñido bromuro de etidio en 0,5
ng/mL. Para la migración se usó 3 uL de buffer de carga (6X DNA loading dye), 8
μL de muestra de ADN y 2 uL de marcador de peso molecular de 1000 pb (1 kb).
Las condiciones eléctricas fueron 90 voltios durante 25 min (Maldonado et al.,
2024).
Análisis de datos
Los RAPD y sus longitudes fueron
clasificados según el cultivar en hojas de cálculo de Microsoft Excel v.2021.
El número de bandas amplificadas, polimórficas y sus respectivos porcentajes se
recolectaron en hojas de cálculo y posteriormente exportaron al software
R-Studio Desktop v. 2022.12.0+353 para la obtención de dendrogramas.
Variabilidad genética
mediante PCR-RAPD
Los patrones generados por los marcadores
RAPD mostraron diferentes resultados en cuanto a las bandas amplificadas,
bandas polimórficas y el porcentaje de polimorfismo en los 9 cultivares de Musa
spp. Analizados (Figura 2 y Tabla 2). Para OPA-1 se generaron 8 bandas
polimórficas a partir de los 9 cultivares de Musa spp., lo cual
representa un 88,8% de polimorfismo respecto al total de las bandas
amplificadas (Figura 2 1.A). Así mismo, con el marcador OPA-2 se generaron 8
bandas polimór-ficas, equivalente a un 47% de polimorfismo (Figura 2 2.A). No
obstante, con el marcador OPA-3 se generaron 9 bandas polimórficas, lo que
representa el 100% de las bandas amplificadas en los cultivares estudiados
(Figura 2 1.B). Por otro lado, con el marcador A-01 se observaron 7 bandas
polimórficas, lo que corresponde al 31,8% de polimorfismo respecto al total de
22 bandas amplificadas (Figura 2 1.C).
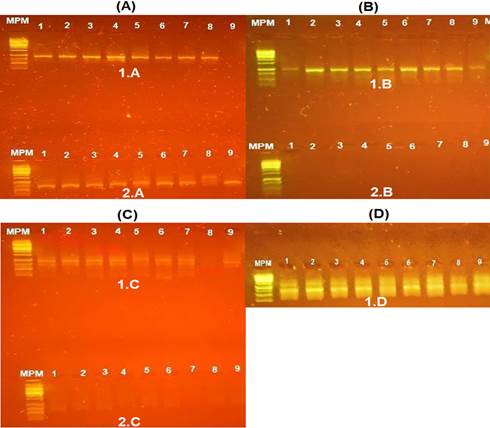
Figura
2. Gel
de agarosa al 1,5% de los amplicones RAPD obtenidos por PCR. MPM: Marcador de
peso molecular. 1: IC2. 2: Valery. 3: Montecristo. 4: Cavendish Gigante. 5: Red
Dacca. 6 Williams: 7: Gran Enano. 8: Gros Michel. 9: Zapatito.
En relación con el marcador OPC-2, se identificaron
un total de 9 bandas polimórficas, lo que repre-senta un 47,3% de polimorfismo
respecto al total de bandas amplificadas (Figura 2 1.D). Este hallazgo sugiere
una considerable variabilidad genética dentro de los cultivares evaluados. Sin
embargo, es relevante señalar que los marcadores OPA-4 y OPC-15 no mostraron
bandas polimórficas en ninguno de los 9 cultivares analizados (Figura 2 2B y
2C). Este comportamiento podría estar relacionado con la especificidad de los
cebadores utilizados y la naturaleza de los cultivares estudiados.
En ese contexto, los resultados obtenidos
con el marcador OPC-2 coinciden con los reportados por Singh et al. (2021),
quienes también observaron un número mayor de bandas polimórficas con el uso de
este marcador, encontrando 36 bandas polimórficas en 25 cultivares de banano (Musa
spp.). Este hallazgo refuerza la idea de que el marcador OPC-2 es efectivo para
detectar variabilidad genética dentro de especies de Musa spp. Según
Asniawati & Purwantoro (2019), el marcador OPC-2 permite identificar la
variabilidad genética en vegetales. Al respecto, Kanani & Shukla (2020)
destacaron que el marcador OPC-2 es particularmente útil para la diferenciación
genética de organismos, debido a los porcentajes de polimorfismo generados durante
la amplificación.
Por otro lado, a pesar de que no se
observaron bandas polimórficas con el marcador OPC-15 en este estudio, otros
trabajos, como el de Nadal et al. (2009), han demostrado que el marcador OPC-15
puede ser útil para la diferenciación de accesiones de Musa spp. a
través de los polimorfismos que se generan. Esto es respaldado por Abekova et
al. (2022), quienes señalan que los cebadores RAPD son herramientas efectivas
para diferenciar plantas, permitiendo la identificación de líneas parentales e
híbridos, lo que destaca el potencial de estos marcadores en estudios de
variabilidad genética.
En relación con los marcadores OPA-1, OPA-2,
OPA-3 y OPA-4, se observó una coincidencia en los primeros tres, pero no con el
último. De manera similar, en los marcadores A-01 y OPC-15, se encontró
concordancia solo con el primero, pero no con el segundo. Esta discrepancia en
la detección de bandas polimórficas puede atribuirse a la presencia de regiones
genómicas específicas en algunas variedades que no son amplificadas
eficientemente por los marcadores RAPD empleados. Este fenómeno ha sido
señalado previamente por Narváez et al. (2000), quienes sugieren que ciertos
loci genéticos pueden no ser susceptibles a la amplificación debido a
características particulares de las secuencias genómicas. Autores como Nadal et
al. (2009) reportaron la detección de 11 bandas polimórficas tanto en el
marcador A-01 como en OPC-15 en 22 accesiones de Musa spp., lo que
indica que la capacidad de los marcadores para generar polimorfismos puede
variar entre diferentes cultivares y/o accesiones. Esta variabilidad en los
resultados de los marcadores RAPD sugiere que no todos son igualmente efectivos
para todas las variedades y/o accesiones de una especie, lo que resalta la
necesidad de seleccionarlos cuidado-samente en función del objetivo del
estudio. Chen et al. (2012) destacan que los marcadores OPA-1, OPA-2, OPA-3 y
OPA-4 generan bandas polimórficas que permiten una adecuada diferenciación
genética de clones en Musa spp., lo cual es consistente con los
resultados obtenidos en este estudio para los tres primeros marcadores. Sin
embargo, la falta de amplificación con OPA-4 en las variedades analizadas
podría reflejar una limitación en la aplicabilidad de este marcador. En
Indonesia, Wahyudi, Adnin y Hapsari (2020) reportaron una variación
intraespecífica significativa en trece cultivares de Pisang Ambon (Musa
acuminata CV. AAA), utilizando veinte marcadores RAPD (OPA-1 a OPA-20),
encontrando un 86,82% de bandas polimórficas y un 13,18% de bandas
monomórficas. Este alto porcentaje de polimorfismo refleja una notable
variabilidad genética dentro de esta especie, lo que resalta la necesidad de
más de 10 marcadores RAPD para caracterizar la diversidad genética de plantas
de banano. Así mismo, es importante señalar que la variabilidad en la respuesta
de los diferentes marcadores en distintas variedades sigue siendo un tema
relevante por considerar para la interpretación de los resultados en estudios
de genética. Por tanto, los resultados de esta investigación pueden ser
dilucidados siendo consistentes con la bibliografía consultada.
Tabla 2
Número de bandas amplificadas, bandas
polimórficas y porcentaje de polimorfismo de los 9 cultivares de Musa spp
|
Oligonucleótido
|
Nº
de bandas amplificadas
|
Nº
de bandas polimórficas
|
Polimorfismo
(%)
|
|
OPA-1
|
9
|
8
|
89
|
|
OPA-2
|
17
|
8
|
47
|
|
OPA-3
|
9
|
9
|
100
|
|
OPA-4
|
0
|
0
|
0
|
|
A-01
|
22
|
7
|
32
|
|
OPC-15
|
0
|
0
|
0
|
|
OPC-2
|
19
|
9
|
47
|
|
Total
|
76
|
41
|
|
|
x̄
|
11
|
6
|
|
Filogenética
El análisis de polimorfismo basado en el
método RAPD reveló un dendrograma en el cual se agruparon los cultivares Enano
(Musa acuminata cv.), Williams (M. acuminata cv.), Montecristo
(M. acuminata cv.), IC2 (M. acuminata cv.) y Valery
(M. acuminata cv.). Estos cultivares mostraron una estrecha relación
genética, lo que sugiere una similitud en su composición genética y/o en los
marcadores amplificados. En contraste, los cultivares Gros Michel (M.
acuminata cv.), Red Dacca (M. acuminata cv.) y Zapatito
(M. paradisiaca cv.) no mostraron similitud con los grupos previamente
mencionados, lo que se atribuye a la ausencia o variabilidad de ciertos
marcadores en estos cultivares específicos. Este patrón de agrupamiento es
consistente con los resultados de Boonsrangsom et al. (2023), quienes también
encontraron una diferenciación genética entre grupos de Musa spp. al
analizar su dendograma, dividiendo las muestras en dos grupos principales con
subgrupos adicionales, lo que refleja la complejidad genética en este cultivo.
No obstante, dicha diferenciación podría estar relacionada con las
características genéticas específicas de cada cultivar. Lo cual es respaldado
por Loh et al. (2000) y Nadal et al. (2009), quienes señalan que, aunque los
cultivares de Musa como el Cavendish conforman un grupo taxonómico
amplio, no todos los cultivares dentro de este grupo presentan una agrupación
homogénea, lo que podría ser explicado por variaciones morfológicas y
organolépticas a nivel del cultivar. En este sentido, los hallazgos de Torres
et al. (2020) sobre la importancia de las características morfológicas como un
reflejo de la variabilidad genética, respaldan la idea de que las diferencias
fenotípicas observadas en los cultivares de banano pueden tener una base
genética subyacente. En estudios previos como el de Hinge et al. (2022), que
analizaron 20 cultivares de banano utilizando múltiples marcadores mole-culares,
se evidenció que la aplicación de técnicas como RAPD permite detectar loci
alélicos específicos, revelando diferencias significativas entre sus cultivares
evaluados. Otros autores como Lamare & Rao (2015) destacan la capacidad de
la técnica PCR-RAPD para determinar genotipos en M. acuminata. Esto es
apoyado por Hasanah et al. (2022), quien aplicó la técnica de PCR-RAPD en un
estudio dirigido a identificar la diversidad genética de las cebollas de varios
lugares del norte de Sumatra, hallando la existencia de un total de 11
variedades mediante el uso de los marcadores OPA-13, OPB-07, OPD-20 y OPM-01.
En relación a lo anteriormente escrito, la
precisión de esta técnica para identificar y diferenciar genotipos es evidente
en los resultados obtenidos en este estudio, que revelan una clara separación
entre los cultivares evaluados como se ha observado en el dendograma generado,
lo que respalda la utilidad del método RAPD en estudios de diversidad genética
en bananos, que también se han probado en otros cultivos banano como Musa
troglodytarum L (Karuwal, Kasiamdari & Daryono, 2024) e incluso para la
diferenciación de microorganismos de interés fitopatológico como Exobasidium
vexans, un hongo que afecta a los cultivos de té (Rachmad, Priyatmojo &
Widiastuti (2022). Por tanto, el análisis realizado con RAPD mostró una clara
diferenciación genética entre los cultivares estudiados según el dendrograma
que se generó y la bibliografía consultada, por lo que los resultados obtenidos
pueden ser dilucidados.
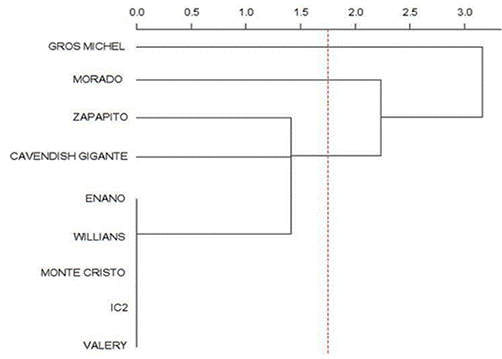
Figura 3. Dendrograma
generado con el software gratuito R-Studio, basado en el análisis de 7
marcadores RAPD de 9 cultivares (IC2, Valery, Montecristo, Cavendish Gigante,
Red Dacca, Williams, Gran Enano, Gros Michel, Zapatito. La escala de
disimilitud se presenta en el margen izquierdo, con un rango de 0.0 a 3.0.
El análisis realizado evidenció que el uso
de la técnica RAPD representa una herramienta altamente eficaz para evaluar el
polimorfismo de los genotipos de Musa spp. en la región de Tumbes. Así
mismo, el dendrograma obtenido reveló una estrecha relación genética entre
cinco cultivares, específicamente cv. Gran Enano, cv. Williams, cv.
Montecristo, cv.IC2 y cv. Valery, lo cual se atribuye a la similitud en el
número de bandas polimórficas detectadas. En contraste, los cultivares cv. Gros
Michel, cv. Red Dacca y cv. Zapatito mostraron una diferenciación genética
significativa, no agrupándolos debido a la ausencia de ciertas bandas
polimórficas o la presencia de un número superior de ellas dentro de cada
genotipo, respectivamente. Además, se observó un 100% de polimorfismo con el
marcador OPA-3, que reveló tanto bandas polimórficas como amplificadas, lo cual
infiere un mayor grado de polimorfismo.
En síntesis, el análisis de variabilidad
genética mediante la técnica RAPD ha permitido identificar la diversidad en los
nueve cultivares de Musa spp. analizados. Estos hallazgos son cruciales
para mejorar la selección de genotipos con características deseables, como
resistencia a plagas y alta productividad, contribuyendo a la optimización de
los cultivos en la región.
Se sugiere que los organismos estatales
responsables de la regulación y supervisión de la producción agrícola en el
Perú incorporen herramientas moleculares, como la técnica RAPD, en los procesos
de caracterización de cultivos. Esta integración permitiría superar las
limitaciones asociadas al uso exclusivo de descriptores fenotípicos en la
agrocaracterización de especies como el banano y/o plátano, favoreciendo una
identificación más precisa y eficiente de los cultivares.
A la Universidad
Nacional de Tumbes por brindarme los conocimientos necesarios para mi
desarrollo profesional en el campo de la investigación.
REFERENCIAS
BIBLIOGRÁFICAS
Abekova,
A. M., Yerzhebayeva, R. S., Bastaubayeva, S. O., Konusbekov, K., Bazylova, T.
A., Babissekova, D. I., & Amangeldiyeva, A. A. (2022). Assessment of sugar beet
genetic diversity in the Republic of Kazakhstan by using RAPD markers and
agromorphological traits. SABRAO Journal of Breeding and Genetics, 54(1),
67-78. http://doi.org/10.54910/sabrao2022.54.1.7
Asniawati,
M. D., & Purwantoro, A. (2019). Genetic diversity of croton (Codiaeum
variegatum (L.) Rumph. ex A. Juss) and its offspring based on RAPD markers.
Ilmu
Pertanian (Agricultural Science), 4(2), 52–58.
https://doi.org/10.22146/ipas.40899
Ávila,
J. A. C., Cárdenas, C. C. C., Moreira, A. V. C., Benites, L. F. V., &
Villanueva, M. S. C. (2024). Actividad antagonista de PGPR en nematodo
fitoparásito Pratylenchus spp. en Musa paradisiaca (Musa acuminata×
M. balbisiana) vc cavendish. Revista Alfa, 8(24), 717-728.
https://orcid.org/0000-0002-7127-2818
Boonsrangsom,
T., Fuenghoi, C., Premjet, D., Suvittawat, K., Ratanasut, K., & Sujipuli,
K. (2023). Genetic
relationships and genome verification of Thai banana cultivars using Random
Amplification of Polymorphic DNA (RAPD) markers. Biodiversitas Journal of
Biological Diversity, 24(7), 3758-3765 https://doi.org/10.13057/biodiv/d240713
Buitrago-Bitar,
M. A., Enríquez-Valencia, A. L., Londoño-Caicedo, J. M., Muñoz-Flórez, J. E.,
Villegas-Estrada, B., & Santana-Fonseca, G. E. (2020). Molecular and morphological
characterization of Musa spp. (Zingiberales: Musaceae) cultivars. Boletín Científico
Del Centro de Museos, 24(1), 33–47.
https://doi.org/10.17151/bccm.2020.24.1.2
Chen,
P., Salazar, E., Fernández, H., Castro, L., Russo, A., & Vásquez, S.
(2011). Detection
of RAPD polymorphisms in Musa spp. with differential response to the attack of Xanthomonas
campestris pv. musacearum. Trop Agronomy, 61(2),
125–132.
https://ve.scielo.org/scielo.php?script=sci_arttext&pid=S0002-192X2011000200003
Cruz
Arévalo, S. D., Romero Meza, R. F., Cedeño Moreira, Á. V., Verdosoto Valencia,
Á. V., Peñafiel Jaramillo, M. F., & Canchignia Martínez, H. F. (2019). Densidad
estomática, contenido de clorofila y relación filogenética en 17 cultivares de
Musa spp. Scientia Agropecuaria, 10(1), 47-54.
http://dx.doi.org/10.17268/sci.agropecu.2019.01.05
Darmayani,
S., Shofi, M., & Raharjeng, A. R. P. (2018). RAPD Analysis of the genetic diversity among
accessions of micropropagation bananas from Indonesia. Journal of Physics:
Conference Series, 1114(1),012137). http://dx.doi.org/10.1088/1742-6596/1114/1/012137
Flores, K.
(2010). Asociatividad
de los productores y cadena productiva de bananos orgánicos en el valle del río
Tumbes-Perú. Revista de Ciencias Empresariales de la Universidad de San
Martín de Porres, 1(1), 37–55.
Guamangate-Casillas,
E. F., León-Reyes, H. A., Granja-Guerra, E., & Barriga-Medina, N. N.
(2024). Evaluación de hongos entomopatógenos en el control de picudo (Metamasius
hemipterus L) en el cultivo de baby banano en el cantón La Maná. Revista
de Ciencias Agropecuarias, 7(14), 92-100.
https://doi.org/10.56124/allpa.v7i14.0080
Hasanah,
Y., Mawarni, L., Hanum, H., & Lestami, A. (2022). Genetic diversity of
shallots (Allium ascalonicum L.) from several locations in North
Sumatra, Indonesia based on RAPD markers. Biodiversitas Journal of
Biological Diversity, 23(5), 2405-2410. https://doi.org/10.13057/biodiv/d230518
Hinge, V.
R., Shaikh, I. M., Chavhan, R. L., Deshmukh, A. S., Shelake, R. M., Ghuge, S.
A., & Kadam, U. S. (2022). Assessment of genetic diversity and volatile
content of commercially grown banana (Musa spp.) cultivars. Scientific
Reports, 12(1), 7979. https://doi.org/10.1038/s41598-022-11992-1
Kanani, P.,
& Shukla, Y. M. (2020). Genetic variability: physiological characteristics,
pathogenicity and molecular diversity of Fusarium oxysporum f. sp. cumini
infecting Cuminum cyminum L. in India. Vegetos, 33(2),
265–276. https://doi.org/10.1007/s42535-020-00104-x
Karuwal,
R. L., Kasiamdari, R., & Daryono, B. S. (2024). Genetic variation of Fei
banana (Musa troglodytarum L.) in Maluku Islands using RAPD markers. SABRAO
J. Breed. Genet, 56(1), 101-111. http://doi.org/10.54910/sabrao2024.56.1.9
Kessel, A.
(2008). Aplicación
de técnicas biotecnológicas en frutales, una vía valiosa para el rescate y la
conservación de estas especies. Cultivos Tropicales, 29(3), 27–37.
Lallana, V.
H., & García, L. F. (2013). Effect of pretreatments on the seed viability
test of Trichocentrum jonesianum (Orchidaceae). Agricultural Research,
15(2), 129–132.
Lamare, A.,
& Rao, S. R. (2015). Efficacy of RAPD, ISSR, and DAMD markers in assessment
of genetic variability and population structure of wild Musa acuminata
Colla. Physiologia Plantarum, 21(3), 349–358. https://doi.org/10.1007/s12298-015-0295-1
Loh, J. P.,
Kiew, R., Set, O., Gan, L. H., & Gan, Y. (2000). Amplified Fragment Length
Polymorphism fingerprinting of 16 banana cultivars (Musa cvs.). Molecular Plant
Pathology,
17(3), 360–366.
https://doi.org/10.1006/mpev.2000.0848
Maldonado
Duque, E. A., Vigíl-Correa, A. M., Zurita Chinguel, L. G., Torres de León, M.
D., Mendoza Rendón, R., Saavedra-Rios, C. Y., Villar Guevara, I. D., Cubas
Zúñiga, C. E., Peña Zarate, M. C., Vilchez Estrada, K. N., & Ruiz Polo, A.
A. (2024). Patogenicidad de Pectobacterium chrysanthemi en Solanum
tuberosum y PCR del espaciador transcrito interno (ITS) del ADNr 16S/23S en
aislados de Musa sp. del norte del Perú. Manglar, 21(3), 299-304.
https://doi.org/10.57188/manglar.2024.032
Martínez,
G., Rey, J. C., Pichardo, R. E. P., & Manzanilla, E. E. (2020). Marchitez
por Fusarium raza tropical 4: Estado actual y presencia en el continente
americano. Agronomía Mesoamericana, 31(1), 259-276.
Martínez-Solórzano,
G. E., & Rey-Brina, J. C. (2021). Bananas (Musa AAA): Importance, production and
trade in Covid-19 times. Agronomía Mesoamericana, 32(3), 1034–1046. https://doi.org/10.15517/am.v32i3.43610
Mehendran,
Y., Kartheeswaran, T., & Kodikara, N. (2022). Banana freshness
identification using image processing techniques. In 7th International
Conference on Business and Industrial Research (ICBIR) (pp. 347–352). https://doi.org/10.1109/ICBIR54589.2022.9786519
Nadal-Medina,
R., Manzo-Sánchez, G., Orozco-Romero, J., Orozco-Santos, M., &
Guzmán-González, S. (2009). Genetic diversity of bananas and plantains (Musa spp.)
determined using RAPD markers. Revista Fitotecnia Mexicana, 32(1),
1–7.
Narváez,
R. C., Valenzuela, B. J., Muñoz Sch, C., & Hinrichsen, R. P. (2000). Comparison of RAPD and AFLP
as methods for genetic identification of Vitis based on the analysis of
anonymous genomic sequences. Biotechnology Journal, 12(6), 405–412.
Organización
de las Naciones Unidas para la Alimentación y la Agricultura [FAO]. (2022). Banano
- Análisis del mercado 2020. Roma.
Padmalatha,
K., & Prasad, M. N. V. (2007). Morphological and molecular diversity in
Pterocarpus santalinus Lf-an endemic and endangered medicinal plant. Medicinal
and Aromatic Plant Science and Biotechnology, 1(2), 263-273.
Palomino,
C., López, C., Espejo, R., Mansilla, R., & Quispe, J. (2014). Evaluación de la
diversidad genética del café (Coffea arabica L.) en Villa Rica (Perú). Ecología
aplicada, 13(2), 129-134.
Paredes,
Y. R. M., Cuatzo, K. M. C., Blas, M. D. J. D., & Resendiz, J. Á. M. (2024).
Usos de la hoja de plátano (Musa paradisiaca L.) en la gastronomía. Revista
de Gastronomía y Cocina, 3(1), 312-312. https://doi.org/10.70221/rgc.10619494
Pillay,
M., Nwakanma, D. C., & Tenkouano, A. (2000). Identification of RAPD markers linked to A
and B genome sequences in Musa. Genome, 43(5), 763–767. https://doi.org/10.1139/g00-038
Pillay,
M., Ogundiwin, E., Nwakanma, D. C., Ude, G., & Tenkouano, A. (2001). Analysis of genetic
diversity and relationships in East African banana germplasm. Theoretical
and Applied Genetics, 102(6–7), 965–970.
https://doi.org/10.1007/s001220000500
Pillay, M., Tenkouano,
A., Ude, G., & Ortiz, R. (2004). Molecular characterization of genomes in
Musa. In M. Jain & R. Swennen (Eds.), Banana improvement: Cellular,
molecular biology, and induced mutations (pp. 271–287). Science Publishers.
Rachmad, F.
D., Priyatmojo, A., & Widiastuti, A. (2022). Genetic diversity analysis of Exobasidium
vexans causing tea blister blight in Wonosobo, Central Java, Indonesia
using RAPD markers. Archives of Phytopathology and Plant Protection, 55(10),
1234-1249. https://doi.org/10.1080/03235408.2022.2086026
Reyes, B. P.
A., & Cevallos, S. (2025). Hongos asociados al cultivo de banano (Musa
spp.) con potencial biotecnológico para el desarrollo de inoculantes. Siembra,
12(1). https://doi.org/10.29166/siembra.v12i1.7053
Silva,
A., Trujillo, I., & Vidal, M. (2009). Evaluación de la inducción de
variabilidad genética en cambur 'Manzano' (Musa AAB) a través de marcadores
RAPD. Agronomía Tropical, 1–9.
Singh,
W. A., Singh, N. S., Devi, E. J., Handique, P. J., & Devi, H. S. (2021). Collection and
characterization of banana gene pools (Musa spp.) in Manipur (NE India)
using PCR–RFLP, RAPD, and ISSR markers. Brazilian Journal of Botany, 44(3),
671–684.
https://doi.org/10.1007/s40415-021-00722-y
Torres,
C. D., García, Á. L., Bermúdez, C., Sarría, Z., Hurtado, R. O., Delgado, E., Pérez,
A., & Fernández, M. O. (2020). Morpho-agronomic and organoleptic response of five
banana cultivars (Musa spp.) under field conditions. Revista
Biotecnología Vegetal, 20(1), 43–50.
Vera
Aviléz, M. J. (2021). Uso de Enmiendas y Activadores Biológicos para el manejo
de nematodos en el cultivo de banano (Musa AAA) zona Caracol, cantón Babahoyo.
Tesis de Maestría, Universidad Técnica de Babahoyo.
Wahyudi, D., Adnin, N. I., & Hapsari, L. (2020). Intraspecific
varia-tion of thirteen pisang Ambon cultivars (Musa acuminata cv. AAA)
from East Java and Central Java (Indonesia) based on random amplified
polymorphic DNA (RAPD) marker. International Journal of Conservation Science,
11(4).