Caracterización
molecular y filogenética del gen 18S ARNr del nemátodo entomopatógeno Heterorhabditis
indica aislado en el norte de Perú
Molecular and phylogenetic
characterization of the 18S rRNA gene of the entomopathogenic nematode Heterorhabditis
indica isolated in northern Peru
José Stalyn Córdova Campos2;
Joel Michel Riojas-González1,2; Pedro S. Castillo-Carrillo1
Rosa Emelda Cornejo-Hidalgo1;
César Augusto Mogollón-Farias2; Segundo Melecio Garcia-Garcia2
Erwin Saúl Rosillo-Urbina2;
Yair Eusebio Ruiz-Turpo2; Archi Alejandro
Ruiz-Polo2
1. Facultad de Ciencias
Agrarias, Universidad Nacional de Tumbes. Ciudad Universitaria, Av.
Universitaria S/N, Tumbes, Perú.
2. Estación Experimental
Agraria Los Cedros. Dirección de Recursos Genéticos y Biotecnología. Instituto
Nacional de Innovación Agraria (INIA), Panamericana Norte Kilómetro 12, Tumbes.
Perú.
* Autor corresponsal: jcordova@inia.gob.pe (J. S. Córdova
Campos).
ORCID
de los autores:
J. S. Córdova-Campos: https://orcid.org/0000-0002-5891-4679 J. M.
Riojas-Gonzáles: https://orcid.org/0009-0007-9966-9554
P. S. Castillo-Carrillo: https://orcid.org/0000-0002-0255-1047 R.
E. Cornejo-Hidalgo: https://orcid.org/0000-0002-2010-0442
C. A. Mogollón-Farias: https://orcid.org/0009-0000-5390-9208 S. M.
Garcia-Garcia: https://orcid.org/0009-0003-6300-9221
E. S. Rosillo-Urbina: https://orcid.org/0009-0008-5011-7588 E.
Ruiz Turpo: https://orcid.org/0009-0003-6477-8583
A. A. Ruiz-Polo: https://orcid.org/0009-0005-1273-2625
_____________________________________________________________________________________________________________________________________________
RESUMEN
La caracterización molecular y filogenética
de especies de nematodos entomopatógenos (NEPs) permiten la diferenciación de
genotipos circulantes así como el mapeo de su distribución geográfica. Además,
de los genotipos más agresivos en la infección de insectos. El objetivo de este
estudio fue realizar la caracterización molecular y filogenética del gen 18S
del ARNr del nemátodo entomopatógeno Heterorhabditis indica aislado en
el norte de Perú. Se analizaron 851 bases nitrogenadas de una secuencia
consenso del gen 18S del ARNr de una cepa de H. indica, aislada de
bosques de algarrobo ubicados en el norte del Perú, la cual ha demostrado
actividad entomopatógena contra Galleria mellonella (polilla de la
cera). La secuencia consenso fue sometida a un análisis de similitud mediante
la herramienta BLAST del GenBank del NCBI, empleando como criterios de búsqueda
un 90% de identidad y cobertura. Las secuencias con dichos criterios fueron
sometidas a un alineamiento múltiple con la secuencia consenso utilizando el
software MEGA v.11. Posteriormente, el alineamiento múltiple se insertó en el
software DnaSP v.5 para su caracterización a nivel de variabilidad monomórfica
y polimórfica, y así identificar y diferenciar haplotipos. En el alineamiento
múltiple, se eliminaron 74 bases no alineadas en sus extremos, de las que
quedaron 777 en las que se identificaron y excluyeron 10 huecos (gaps),
obteniéndose una matriz final de 767 bases para la caracterización molecular y
filogenética. En el análisis de similitud se identificaron 6 secuencias con
identidad de 84,89% a 85,11% y coberturas de 92% a 99%. En la caracterización,
se encontró 58 sitios conservados (monomórficos) y 109 mostraron variabilidad
(polimórficos), lo cual representa posibles eventos mutacionales. Asimismo, se
identificaron seis haplotipos distintos (Hap-1 a Hap-6), con un índice de
diversidad haplotípica (Hd) de 0,9524, evidenciando que la secuencia consenso
evaluada presentó un solo haplotipo (Hap-2) por lo que se le asignó un código
de diferenciación genotípica (C05HI). En la filogenética, la secuencia se
emparentó con una secuencia registrada en el GenBank (OK493747.1), formando un
grupo monofilético. Se sugiere la ampliación de estudios de H. indica
C05HI el cual puede ser encontrado en suelos de bosques de algarrobo en el
norte de Perú. Así mismo, se recomienda que organismos estatales implementen en
sus programas de capacitación, asistencia y/o control de plagas de la especie
de nematodo analizada.
Palabras clave: ARNr;
entomopatógeno; monomorfismos; polimorfismos; mutaciones; nematodo.
ABSTRACT
The molecular and phylogenetic characterization of
entomopathogenic nematode (EPN) species allows for the differentiation of
circulating genotypes as well as the mapping of their geographic distribution,
including the identification of the most aggressive genotypes in insect
infection. The aim of this study was to perform the molecular and phylogenetic
characterization of in 18S rRNA gene of the
entomopathogenic nematode Heterorhabditis indica isolated from northern
Peru. A total of 851 nucleotides from a consensus sequence of the 18S
rRNA gene of a H. indica strain, collected from algarrobo (carob tree)
forests in northern Peru and previously shown to exhibit entomopathogenic
activity against Galleria mellonella (wax moth), were analyzed. The
consensus sequence was subjected to a similarity search using the BLAST tool
from the NCBI GenBank database, applying a threshold of 90% for both identity
and coverage. Sequences meeting these criteria were aligned with the consensus
sequence through multiple sequence alignment using MEGA v.11 software. The
resulting alignment was then analyzed using DnaSP v.5 software to assess
monomorphic and polymorphic variability, allowing the identification and
differentiation of haplotypes. During the alignment process, 74 unaligned bases
at the sequence ends were removed, and 10 gap positions were excluded, yielding
a final matrix of 767 aligned bases for molecular and phylogenetic analysis.
The similarity analysis identified six sequences with identity ranging from
84.89% to 85.11% and coverage values between 92% and 99%. In the molecular
characterization, 658 sites were found to be conserved (monomorphic), while 109
were variable (polymorphic), representing potential mutational events.
Additionally, six distinct haplotypes (Hap-1 to Hap-6) were identified, with a
haplotype diversity index (Hd) of 0.9524, indicating that the analyzed
consensus sequence belonged to a single haplotype (Hap-2), to which a genotypic
differentiation code (C05HI) was assigned. In the phylogenetic analysis, the
sequence was closely related to a registered sequence in GenBank (accession
number OK493747.1), forming a monophyletic group. It is recommended that
further studies be conducted on H. indica C05HI, which may be present in
algarrobo forest soils in northern Peru. Likewise, it is suggested that
governmental institutions incorporate this nematode species into their
training, technical assistance, and/or pest control programs.
Keywords: ARNr; entomopathogen; monomorphisms; polymorphisms;
mutations; nematode.
Recibido: 17-02-2025.
Aceptado: 09-06-2025
INTRODUCCIÓN
El crecimiento demográfico y las crecientes
exigencias del mercado alimentario han impulsado un notable incremento en la
producción agrícola a nivel global, proyectando que la población mundial
alcanzará entre 9,4 y 10 mil millones de personas para el año 2050, lo que
supone una presión significativa sobre los sistemas de producción de alimentos
(UN Human Rights Council, 2017).
En ese contexto, las personas se ven
expuestas a un número creciente de productos agroquímicos a través de diversas
fuentes, como los alimentos y el entorno. Esta exposición puede ser continua y
acumulativa, incluso a niveles bajos, lo que plantea riesgos potenciales para
la salud a largo plazo. (Reffstrup, Larsen & Meyer, 2009; Bautista, Mesa
& Gómez, 2018), siendo los plaguicidas una de las principales sustancias
responsables de esta exposición (Correa-Núñez & Rojas-Jaimes, 2022).
Desde hace varias décadas, se ha
intensificado la preocupación social y científica respecto al uso extensivo e
inadecuado de plaguicidas en la agricultura. Esta inquietud se fundamenta en
numerosos estudios que han documentado los efectos negativos derivados de su
aplicación, tales como la disminución de la calidad del agua (Rice, Horgan
& Rittenhouse, 2010), la contaminación progresiva de los suelos agrícolas,
la selección de poblaciones de insectos resistentes y el incremento de riesgos
para la salud humana, incluyendo efectos neurotóxicos y endocrinos (Torres
& Capote, 2004; Elbaz et al., 2009; Delgado et al., 2020). Estos impactos
no solo comprometen la sostenibilidad de los agroecosistemas, sino que también
afectan la seguridad alimentaria y la biodiversidad (Solís, Ramírez &
Rodríguez, 2024). En respuesta a esta problemática, la investigación científica
ha orientado sus esfuerzos hacia el desarrollo de alternativas ecológicas de
control de plagas, entre las cuales destacan los nematodos entomopatógenos
(NEPs) por su alta especificidad y bajo impacto ambiental.
Se ha comprobado que algunas especies de
hongos y nematodos pueden actuar como agentes biológicos eficaces en el control
de plagas en entornos agrícolas (Půža & Tarasco, 2023), tal es el caso
de Steinernema riobrave y Rhabditis blumi que son nematodos
entomopatógenos (NEPs) de las larvas de Heliothis virescens (Gutiérrez
et al., 2025) o nematodos de los géneros Steinernema y Heterorhabditis
que son entomopatógenos de Eurhizococcus colombianus (Restrepo et al.,
2022).
Estudios realizados en condiciones
controladas de invernadero con Heterorhabditis spp. para el manejo de
larvas de Phyllophaga elenans en los estadios L2 y L3 mostraron una tasa
de mortalidad del 50% al aplicar una dosis de 481 nematodos por larva
(Rodríguez et al., 2009). De manera similar, este mismo género de nematodo ha
sido evaluado para el control de Cosmopolites sordidus (el picudo del
banano), en condiciones in vitro, demostrando que es capaz de infectar y causar
la muerte de los estadios larvales más avanzados (L5) en esta especie de
insecto (Amador et al., 2015).
Sin embargo, estudios dirigidos a la
identificación molecular, caracterización y/o análisis filogené-ticos de
nemátodos entomopatógenos (NEPs), realizados en Perú son escasos, como se ha demostrado
en la Libertad, donde se evaluaron tres cepas nativas que se multiplicaron en
larvas de Galleria mellonella y se enfrentaron a ninfas de Proarna
bergie para su control en espárragos, donde se evidenció su efecto letal,
siendo Heterorhabditis indica la especie nematoda identificada mediante
PCR dirigida al gen 18S ARNr (Cedano, 2019).
Debido a la aplicación de los NEPs en
insectos predadores de cultivos agrícolas y la limitada información disponible
sobre su caracterización molecular y filogenética en especies nativas del norte
de Perú como Heterorhabditis indica, se diseñó este estudio con el fin
de contribuir y/o ampliar el conocimiento sobre genotipos circulantes de dicha
especie de nematodo, utilizando el gen 18S ARNr como región diana para el
análisis. Así mismo, la información obtenida en el presente estudio podrá ser
empleada y consultada como base teórica para el desarrollo de estudios
posteriores dirigidos al registro de genotipos de NEPs para su integración en
los programas y/o estrategias del manejo de plagas. El objetivo del estudio fue
realizar caracterización molecular y filogenética del gen 18S ARNr del nemátodo
entomopatógeno Heterorhabditis indica aislado en el norte de Perú.
Diseño del estudio
Se realizó un estudio in
silico con un diseño no experimental, de nivel descriptivo y enfoque
cualitativo, basado en el análisis bioinformático de una secuencia consenso del
gen 18S ARNr, con una longitud de 851 bases nitrogenadas. La secuencia
corresponde al material genético de una cepa del nematodo H. indica,
aislada de suelos de bosques de algarrobo en el distrito de Corrales
(coordenadas: E-555240, N-9602910), en el departamento de Tumbes. Dicha cepa
posee la característica de que se ha identificado previamente mediante
taxonomía clásica (Grifaldo et al., 2020) y se ha evaluado frente a larvas de Gallera
mellonella (gusano de la cera), demostrando un efecto letal.
Obtención de las secuencias
del gen 18S ADNr y su consenso
El material genético
analizado se encontró en formato de lectura de texto (FASTA), el cual pertenece
a investigaciones realizadas por la Universidad Nacional de Tumbes en conjunto
con la subdirección de recursos genéticos de la Estación Experimental Agraria
(EEA) Los Cedros del Instituto Nacional de Innovación Agraria (INIA) del
Ministerio de Agricultura. Ambas instituciones se ubican en Tumbes, norte de
Perú.

Figura 1. Secuencia consenso del gen 18S ARNr de Heterorhabditis
indica del norte de Perú.
Para la obtención de las secuencias de ADN del gen
18S ARNr en ambos sentidos (directo y reverso), inicialmente se realizó una
extracción de ADN total con el Kit PowerSoil DNeasy® (QIAGEN, Alemania). Luego,
se amplificó el gen 18S ARNr mediante una reacción en cadena de la polimerasa
(PCR, por sus siglas en inglés), empleando los cebadores SSU18A:
AAAGATTAAGCCATGCATG y SSU26R: CATTCTTGGCAAATGCTTTCG (Blaxter et al., 1998).
Después, el producto PCR (amplicon) fue secuenciado con la tecnología Sanger de
doble cadena por la empresa Biotecnológica Macrogen (https://dna.macrogen.com/). Finalmente,
empleando el software MEGA v.11 (disponible en https://mega.io/es) se obtuvo la
secuencia consenso de 851 bases que se ha analizado en el presente estudio.
Selección de secuencias con
similitud
La secuencia consenso analizada en el
presente estudio se insertó en la herramienta básica de búsqueda de alineación
local (BLAST, por sus siglas en inglés https://blast.ncbi.nlm.nih.gov/Blast.cgi), donde se realizó
la búsqueda de similitudes con secuencias almacenadas en la base de datos del
Centro Nacional de Investigación Biotecnológica (NCBI, por sus siglas en ingles
http://www.ncbi.nlm.nih.gov/).
Posteriormente, las secuencias con las que
se encontró similitud se registraron mediante su código de acceso para la
caracterización molecular y filogenética.
Caracterización molecular
Para la caracterización molecular, la
secuencia consenso y las secuencias con las que se halló similitud, fueron
sometidas a un alineamiento múltiple software MEGA v.11 para la búsqueda de
regiones consensuadas finales. Posteriormente, fueron importadas y procesadas
en el software DNAsp v.5 (http://www.ub.edu/dnasp/). Dado que las
secuencias corresponden a un organismo diploide, se optó por la opción
"Open Unphase/Genotype Data file" para su importación. Los
polimorfismos, su localización y los haplotipos generados se obtuvieron de
manera inmediata.
Filogenética
La filogenética se llevó a cabo mediante un
análisis evolutivo detallado, en el cual se construyó un árbol filogenético
utilizando la herramienta MEGA v.11, aplicando el método de máxima
verosimilitud. La estimación de las distancias evolutivas se realizó con el
modelo de 3 parámetros de Tamura (1992) con 800 réplicas, utilizando como grupo
externo la secuencia del gen 18S ARNr del nemátodo Heterorhabditis
noenieputensis JN620538.1 para la calibración de las relaciones evolutivas.
Análisis de datos
Las secuencias consenso fueron organizadas
en documentos de Microsoft Word v.2021. De manera similar, los códigos de
acceso de las secuencias que mostraron similitud con la secuencia consenso
fueron clasificados en tablas utilizando Microsoft Excel v.2021.
Selección de secuencias con
similitud
En base a la identificación taxonómica
clásica del nemátodo Heterorhabditis indica, se seleccionaron las
similitudes encontradas en las secuencias del gen 18S ARNr disponibles en el
banco de datos genéticos del NCBI. Se identificaron seis coincidencias, cuyos
códigos de acceso son: PQ608564.1, OL470132.1, OK562793.1, OL470127.1, OK493747.1
y OL469960.1. Los porcentajes de identidad se hallaron entre 84,89% y 85,11%, y
los de cobertura entre 92% y 99%, infiriendo similitud entre la secuencia
consulta y las disponibles en el banco de genes. No obstante, se descartaron
similitudes que corresponden a otras especies. Estos hallazgos reflejan la
limitada información genética disponible para el gen 18S ARNr de Heterorhabditis
indica, siendo este uno de los pocos estudios que documentan el uso de
dicho marcador genético (Tabla 1).
El empleo de secuencias de códigos de
barras, como el gen 18S, para la identificación y caracterización molecular de
nematodos ha sido ampliamente documentado en la literatura científica de las
últimas décadas. Córdova-Campos et al. (2023), emplearon el gen 18S ARNr para
la identificación y caracterización molecular del nematodo agallador de la raíz
Meloidogyne javanica. Cedano (2019), identificó mediante el gen 18S ARNr
a Heterorhabditis iindica como una especie con efecto letal sobre Galleria
mellonella empleada como hospedados y ninfas de P. bergie a las que
se enfrentaron como plagas.
Tabla 1
Secuencias del banco de genes con
similitudes y/o coincidencias con la secuencia consenso del gen 18S ARNr de Heterorhabditis
indica analizada
|
Descripción
|
Nombre
científico
|
%
cobertura
|
%
identidad
|
Accesion
|
|
Heterorhabditis indica strain
IRQ ZAh17 small subunit ribosomal RNA gene_partial_sequnce
|
Heterorhabditis
indica
|
99
|
84,89
|
PQ608564.1
|
|
Heterorhabditis
indica isolate O1 small subunit ribosomal RNA
gene_partial_sequence
|
Heterorhabditis
indica
|
96
|
85,25
|
OL470132.1
|
|
Heterorhabditis
indica isolate HA1 small subunit ribosomal RNA
gene_partial_sequence
|
Heterorhabditis
indica
|
96
|
84,80
|
OK562793.1
|
|
Heterorhabditis
indica isolate O1 small subunit ribosomal RNA
gene_partial_sequence
|
Heterorhabditis
indica
|
92
|
85,39
|
OL470127.1
|
|
Heterorhabditis
indica isolate HR1 small subunit ribosomal RNA
gene_partial_sequence
|
Heterorhabditis
indica
|
93
|
84,86
|
OK493747.1
|
|
Heterorhabditis
indica isolate H1 small subunit ribosomal RNA
gene_partial_sequence
|
Heterorhabditis
indica
|
92
|
85,11
|
OL469960.1
|
Vrain et al. (1992)
llevaron a cabo un análisis de los polimorfismos de longitud de fragmentos de
restricción del ADNr en Xiphinema americanum, demostrando la
variabilidad genética intraespecífica en este nematodo. De manera similar,
Floyd et al. (2002) utilizaron el gen 18S ARNr como marcador molecular para la
identificación de nematodos obtenidos de muestras de suelo. Este gen fue
amplificado mediante cebadores desarrollados por Blaxter et al. (1998), quienes
destacaron su utilidad para estudios evolutivos dentro del filo Nematoda, dada
su capacidad para proporcionar información filogenética a nivel de especies y
géneros. No obstante, según Jaramillo et al. (2015), la secuenciación de
regiones ITS del ADNr de estadios juveniles de nematodos como los del género Meloidogyne.
Otros autores como Fernández et al. (2000), señalan el uso del gen ND3
mitocondrial para estudios moleculares dirigidos a la identificación de
nematodos hospedadores. Por su parte, Okamoto et al. (1995), han estudiado
especies de cestodos mediante secuencias del gen mitocondrial citocromo c
oxidasa subunidad I (COI). Por tanto, los resultados obtenidos en el presente
estudio pueden ser dilucidados.
Caracterización molecular
Del alineamiento múltiple entre la secuencia
consenso de 851 bases nitrogenadas del gen 18S ARNr Heterorhabditis indica
aislado del norte de Perú y las secuencias con las que se obtuvieron
similitudes, se eliminaron 74 bases no alineadas en sus extremos, de las que
quedaron 777 en las que se identificaron y excluyeron 10 sitios gaps, quedando
un total de 767 bases para la caracterización molecular, donde se halló que 658
corresponden a sitios invariables (monomórficos) y 109 a sitios variables
(polimórficos) (Figura 2). Estos sitios variables se distribuyeron en 108
mutaciones ubicadas en las posiciones 8; 10; 13; 15; 28; 35; 41; 43; 46; 56;
66; 75; 83; 85; 90; 92; 93; 96; 99; 100; 101; 105; 115; 121; 122; 123; 130;
133; 150; 151; 152; 166; 177; 178; 181; 182; 184; 186; 194; 199; 203; 205; 219;
220; 224; 225; 232; 240; 243; 256; 259; 267; 274; 275; 280; 284; 297; 305; 315;
316; 318; 320; 323; 332; 333; 340; 346; 348; 350; 351; 356; 360; 363; 369; 397;
403; 404; 422; 460; 463; 472; 473; 475; 476; 488; 490; 501; 507; 512; 513; 522;
528; 531; 533; 546; 550; 552; 558; 567; 570; 579; 582; 612; 618; 700; 724; 751;
765 y 1 sitio informativo de parsimonia en la posición 89. Así mismo, se
identificaron 6 haplotipos con una diversidad genética (Hd) de 0.9524: el Hap-1
corresponde a la secuencia OL469960.1, el Hap-2 a la secuencia consenso
analizada, el Hap-3 a PQ608564.1 y OL470132.1, el Hap-4 a OK562793.1, el Hap-5
a OL470127.1 y el Hap-6 a OK493747.1. Los resultados mostraron una diversidad
genética significativa entre las secuencias analizadas, lo que se asocia al
número de mutaciones. En este contexto, la secuencia consenso (Hap-2) estudiada
corresponde a un nuevo genotipo del gen 18S ARNr, por lo que se le asignó el
código C05HI.
Como se muestra en la Figura 2, las
principales características moleculares de diferenciación halladas en las
secuencias analizadas, correspon-den a mutaciones que han dado lugar a la
formación de polimorfismos en el gen 18S ARNr. Antes de este estudio, la literatura
disponible sobre Heterorhabditis en Perú se centraba principal-mente en
investigaciones vinculadas a su ecología y su potencial entomopatógeno, como lo
señalan Adrianzén et al. (2024) y Vergaray (2014). Lo cual también se ha
investigado en otros países (Valdés, Rodríguez & Velásquez, 2014; Kour et
al., 2022). No obstante, Córdova et al. (2023), han realizado la
caracterización molecular de Meloidogyne javanica mediante el gen 18S
ARNr en Perú, siendo uno de los pocos estudios en el que se ha empleado dicho
marcador. Así mismo, en India, se han llevado a cabo estudios que incluyen la
caracterización molecular de esta especie de nematodo, mediante el análisis del
ADNr, como se describe en el trabajo de Bhat et al. (2021).
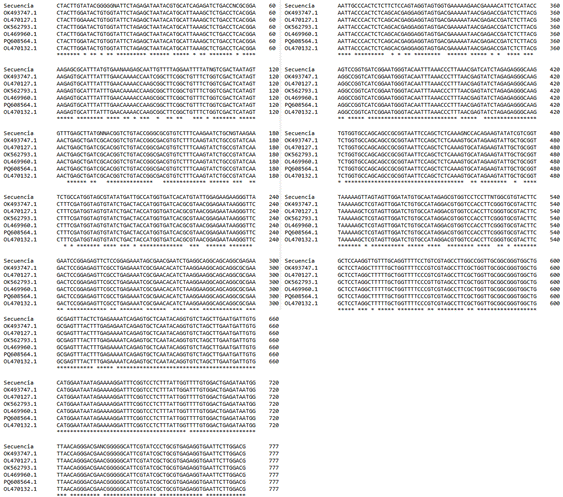
Figura 2. Alineamiento
múltiple entre la secuencia consenso del gen 18S ARNr de Heterorhabditis
indica del norte de Perú y las secuencias con las que se obtuvo similitud.
Los asteriscos (*) indican coincidencias entre las bases nitrogenadas, y su
ausencia variaciones en la secuencia.
Filogenética
El árbol filogenético construido mediante el
método de máxima verosimilitud reveló relaciones evolutivas entre las
secuencias analizadas. Como se aprecia en la figura 3, las secuencias se
agruparon formando cinco clados donde la secuencia consenso de la cepa analizada
se agrupa con OK493747.1 formando un grupo monofilético (clado IV). Así mismo,
las secuencias OL470127.1 y OL469960.1 se agrupan formando otro grupo
monofilético (clado I), OL470132.1 el clado (II), PQ608564.1 el clado (III) y
OK562793.1 el clado (V). Por otra parte, la hipótesis evolutiva muestra que el
clado (V) es el más antiguo, y de él desciende el clado (IV) donde se encuentra
la secuencia consenso de Heterorhabditis indica del norte de Perú con
OK493747.1. Así mismo, se aprecia que los clados (I) y (II) comparten un nodo
común que surge del clado (III), siendo descendientes que se emparentan con el
clado (IV).
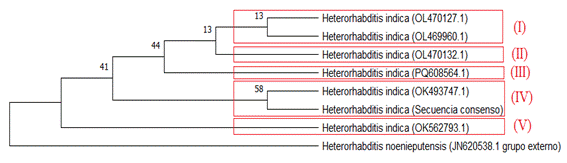
Figura
3.
Árbol
filogenético basado en el método de máxima verosimilitud y el modelo 3
parámetros de Tamura (1992) con un valor bootstrap de 800 réplicas, que
muestran relación entre las secuencias del gen 18S ARNr del nematodo
entomopatógeno Heterorhabditis indica.
Los resultados obtenidos en la filogenia
muestran consistencia con observaciones previas de Malan, Knoetze & Tiedt
(2014), quienes también reportaron la aparición de un grupo monofilético
conformado por secuencias de Heterorhabditis indica. Sin embargo, en su
caso, las secuencias utilizadas corresponden al espaciador interno transcrito
(ITS) y a la región D2D3 del gen 28S del ADNr. Así mismo, Bhat et al. (2021),
indican que el uso del gen ITS del ADNr en especies del género Heterorhabditis
permite la identificación de clados monofiléticos, lo que facilita la
inferencia de su clasificación taxonómica con mayor resolución. Según Rodríguez
(2002), el gen 18S del ADNr ha sido ampliamente usado desde el punto de vista
sistemático. Sin embargo, también se han utilizado otros genes para estudiar
relaciones filogenéticas. De acuerdo con Littlewood et al. (1998), la
combinación de las secuencias completas del gen 18S con las parciales del 28S
permiten un análisis filogenético de nematodos con la mayor la resolución
dado que integra información proveniente de diferentes regiones del ADNr. Por
tanto, en base a la bibliografía consultada los resultados de la filogenia de Heterorhabditis
indica aislado del norte de Perú pueden ser dilucidados. No obstante,
aunque el gen 18S ARNr ha demostrado ser útil para la identificación taxonómica
en este estudio, la comparación con otros marcadores como el ITS y el 28S
sugiere que una combinación de genes ribosomales que puede ser más eficaz y
precisa en la determinación de relaciones filogenéticas en especies de
nematodos.
La secuencia consenso de 851 bases del gen
18S ARNr del nematodo entomopatógeno Heterorhabditis indica aislado del
norte de Perú exhibe características moleculares distintivas en comparación con
las secuencias disponibles en el banco de genes del NCBI. Este hallazgo
evidencia la existencia de un genotipo (C05HI) propio de Heterorhabditis
indica para el norte de Perú, ya que no se han encontrado registros previos
en la bibliografía nacional y en las bases de datos genéticas internacionales.
Estos resultados, destacan la relevancia de aplicar técnicas moleculares
avanzadas para la asignación precisa de la identidad taxonómica y/o la
identificación de genotipos circulantes según el marcador molecular empleado,
facilitando el mapeo de la distribución geográfica de cepas de nematodos con
propiedades entomopatógenas. Así mismo, esta información es relevante para la
implementación de controladores biológicos y/o el desarrollo de estrategias
innovadoras en el manejo y control integrado de plagas, promoviendo así la
sostenibilidad de los sistemas agrícolas en Perú.
A la subdirección de recursos genéticos del
Instituto Nacional de Innovación Agraria (INIA) por el apoyo brindado en el
desarrollo del presente estudio. A la Universidad Nacional de Piura, por
proporcionar la secuencia de ADN analizada.
REFERENCIAS
BIBLIOGRÁFICAS
Adrianzén, J. E. R., Dávila, R. S. C., Tesén, E. D. P.,
Arias, C. P. C., & Cabrera, A. C. (2024). Ecological aspects of three
strains of entomopathogenic nematodes from the department of Lambayeque-Peru. Egyptian
Journal of Biological Pest Control, 34(1), 53.
Amador, M., Molina, D., Guillen, C., Parajeles, E., Jiménez,
K., & Uribe, L. (2015). Utilización del nematodo entomopatógenos Heterorhabditis
atacamensis CIA-NE07 en el control del picudo del banano Cosmopolites
sordidus en condiciones in vitro. Agronomía Costarricense, 39(3),
47-60. https://doi.org/10.15517/rac.v39i3.21791
Bautista, E. J., Mesa, L., & Gómez Alvarez, M. I. (2018).
Alternativas de producción de bioplaguicidas microbianos a base de hongos: el
caso de América Latina y El Caribe. Scientia Agropecuaria, 9(4), 585-604.
http://dx.doi.org/10.17268/sci.agropecu.2018.04.15
Bhat, A. H., Chaubey, A. K., Shokoohi, E., & Machado, R.
A. (2021). Molecular and phenotypic characterization of Heterorhabditis
indica (Nematoda: Rhabditida) nematodes isolated during a survey of
agricultural soils in Western Uttar Pradesh, India. Acta
Parasitologica, 66, 236-252. https://doi.org/10.1007/s11686-020-00279-y
Blaxter, M. L., De Ley, P., Garey, J., et al. (1998). A
molecular evolutionary framework for the phylum Nematoda. Nature, 392, 71–75. https://doi.org/10.1038/32160
Cedano
Saavedra, C. E. (2019). Nematodos entomopatógenos nativos para el control de
ninfas de (Proarna bergie) en espárrago, Paiján, La Libertad [Tesis de
Doctorado, Universidad Nacional Agraria La Molina]. Repositorio digital UNAM. http://45.231.83.156/handle/20.500.12996/4247
Córdova-Campos, J., Calle-Ulfe, P. G., Suarez-Peña, E.,
Mendez-Farroñan, S., Lindo-Seminario, D. E., Calle, S. G., ... &
Condemarín-Montealegre, C. (2023). Identification of effecting proteins by
MALDI TOF/TOF mass spectrometry of the root-knot nematode Meloidogyne
javanica. Tropical and Subtropical Agroecosystems, 26(1). http://doi.org/10.56369/tsaes.4518
Correa-Núñez, G., & Rojas-Jaimes, J. (2022). Uso de
plaguicidas no autorizados en alimentos agrícolas primarios, Perú (2011-2018). Manglar,
19(1), 61-65. https://doi.org/10.17268/manglar.2022.008
Delgado-Oramas, B. P., Marquetti, I. G., Hernández, M. G. R.,
& Pérez, O. P. (2020). La resistencia inducida por productos derivados de
plantas: alternativa para el manejo de plagas agrícolas. Revista de
Protección Vegetal, 35(3).
Elbaz, A., Clavel, J., Rathouz, P. J., Moisan, F., Galanaud,
J. P., Delemotte, B., Alpérovitch, A., et al. (2009). Professional
exposure to pesticides and Parkinson disease. Annals of Neurology, 66(4),
494-504. https://doi.org/10.1002/ana.21717
Fernandez, M., Aznar, F. J., Raga, J. A., & Latorre, A.
(2000). The origin of Lecithodesmus (Digenea: Campulidae)
based on ND3 gene comparison. Journal of Parasitology, 86(4), 850-852. https://doi.org/10.1645/0022-3395(2000)086[0850:TOOLDC]2.0.CO;2
Floyd, R., Abebe, E., Papert, A., & Blaxter, M. (2002). Molecular
barcodes for soil nematode identification. Molecular Ecology, 11(4),
839-850. https://doi.org/10.1046/j.1365-294X.2002.01485.x
Grifaldo-Alcantara, P. F., Alatorre-Rosas, R., Silva-Rojas,
H. V., Stock, S. P., Hernández-Rosas, F., Vargas-Madriz, H., &
Durán-Trujillo, Y. (2020). Caracterización molecular y
morfométrica de Heterorhabditis indica (cepa CP13JA) aislado en el
cultivo de caña de azúcar. Tropical and Subtropical
Agroecosystems, 23(2), 64.
Gutiérrez, C. G., Villezcas, H. M. M., Pérez-Álvarez, S.,
& Mondaca, E. C. (2025). Formulación de hongos y nematodos entomopatógenos
para el control de Heliothis virescens (Fabricius) en tomate. Revista
Internacional de Contaminación Ambiental, 41, 17-25. https://doi.org/10.20937/RICA.55086
Jaramillo-Pineda, J., Guerrero-Olazarán, M., Fuentes-Garibay,
J. A., Viader-Salvadó, J. M., Meza-García, J. L., & Morales-Ramos, L. H.
(2015). Identificación de especies de Meloidogyne utilizando la
secuenciación de regiones espaciadoras transcritas internas de ADN ribosomal de
estadios juveniles. Revista Mexicana de Fitopatología, 33(1), 1-11.
Kour, S., Khurma, U., Brodie, G., & Singh, S. (2022). Modeling
the potential global distribution of suitable habitat for the biological
control agent Heterorhabditis indica. Ecology and Evolution, 12(6),
e8997. https://doi.org/10.1002/ece3.8997
Littlewood, D. T. J., Rohde, K., & Clough, K. A. (1998). The
phylogenetic position of Udonella (Platyhelminthes). International
Journal for Parasitology, 28(8), 1241–1250.
https://doi.org/10.1016/S0020-7519(98)00108-8
Malan, A., Knoetze, R., & Tiedt, L. R. (2014). Heterorhabditis
noenieputensis n. sp. (Rhabditida: Heterorhabditidae), a new
entomopathogenic nematode from South Africa. Journal of Helminthology, 88,
139–151. https://doi.org/10.1017/S0022149X12000806
Okamoto, M., Bessho, Y., Kamiya, M., Kurosawa, T., & Horii, T.
(1995). Phylogenetic relationships within Taenia taeniaeformis variants
and other taeniid cestodes inferred from the nucleotide sequence of the
cytochrome c oxidase subunit I gene. Parasitology Research, 81(6),
451–458. https://doi.org/10.1007/bf00931785
Půža, V., & Tarasco, E. (2023). Interactions
between entomopathogenic fungi and entomopathogenic nematodes. Microorganisms,
11(1), 163. https://doi.org/10.3390/microorganisms11010163
Reffstrup, T. K., Larsen, J. C., & Meyer, O. (2009). Risk assessment
of mixtures of pesticides: Current approaches and future strategies. Regulatory
Toxicology and Pharmacology, 56, 174–192. https://doi.org/10.1016/j.yrtph.2009.09.013
Restrepo-García, A. M., Bernal-Arias, L., & Soto-Giraldo, A. (2022).
Eurhizococcus colombianus Jakubski
(1965) (Hemiptera: Margarodidae): Evaluación de su control biológico utilizando
una mezcla de los nematodos entomopatógenos Steinernema (Rhabditida:
Steinernematidae) y Heterorhabditis (Rhabditida: Heterorhabditidae). Boletín
Científico. Centro de Museos. Museo de Historia Natural, 26(1), 155–168. https://doi.org/10.17151/bccm.2022.26.1.11
Rice, P. J., Horgan, B. P., & Rittenhouse, J. L. (2010). Evaluation
of core cultivation practices to reduce ecological risk of pesticides in runoff
from Agrostis palustris. Environmental Toxicology and Chemistry, 29(6),
1215–1223. https://doi.org/10.1002/etc.179
Rodríguez, D., Torres, M., Uribe, L., & Flores, L. (2009). Susceptibilidad
de los estadios L2 y L3 de Phyllophaga elenans a una cepa nativa de Heterorhabditis
sp. en condiciones de invernadero. Agronomía Costarricense, 33(2),
171–182.
Rodríguez, P. F. (2002). Estudio faunístico y sistemático
de helmintos de aves canarias (Doctoral dissertation, Universidad de La
Laguna, Canary Islands, Spain).
Saitou, N., & Nei, M. (1987). The neighbor-joining method: A new
method for reconstructing phylogenetic trees. Molecular Biology and
Evolution, 4, 406–425. https://doi.org/10.1093/oxfordjournals.molbev.a040454
Solis, V. M.
T., Ramirez, Y. D. R. M., & Rodríguez, M. R. (2024). Agricultura de subsistencia
en las zonas de influencia de las áreas naturales protegidas en Tamaulipas:
Agricultura de subsistencia. Revista Mexicana de Agroecosistemas, 11(2).
https://doi.org/10.60158/rma.v11i2.436
Tamura,
K. (1992). Estimation
of the number of nucleotide substitutions when there are strong
transition-transversion and G + C-content biases. Molecular Biology and
Evolution, 9, 678–687. https://doi.org/10.1093/oxfordjournals.molbev.a040752
Torres, D.,
& Capote, T. (2004). Agroquímicos un problema ambiental global: uso del
análisis químico como herramienta para el monitoreo ambiental. Ecosistemas, 13(3).
UN Human
Rights Council. (2017). Report of the Special Rapporteur on the right to
food, Olivier De Schutter. United Nation. https://digitallibrary.un.org/record/766914?v=pdf
Valdés, M.
D. C. C., Rodríguez, J. G., & Velásquez, E. P. (2014). Susceptibilidad de
larvas y pupas de Typophorus nigritus F. al nematodo entomopatógeno Heterorhabditis
indica Poinar. Centro Agrícola, 41(3), 23–28.
Vergaray
Ramírez, M. A. (2014). Patogenicidad de nemátodos entomopatógenos del género
Heterorhabditis (Nematoda: Heterorhabditidae) sobre larvas de Aedes
aegypti L. (Diptera: Culicidae) en condiciones de laboratorio.
Vrain, T.
C., Wakarchuk, D. A., Levesque, A. C., & Hamilton, R. I. (1992).
Intraspecific rDNA restriction fragment length polymorphisms in the Xiphinema
americanum group. Fundamental and Applied Nematology, 15, 563–574.