Diversidad
bacteriana en el humedal costero artificial Poza
La Arenilla mediante el análisis de ADN ambiental
Bacterial
diversity in the artificial coastal wetland Poza La Arenilla through
environmental DNA analysis
Mayra Silva1;
Angel I. Ramirez1; Abraham Espinoza-Culupú1; Mario Cueva1;
Rina Ramírez1;
Pedro E. Romero1*
1 Facultad
de Ciencias Biológicas, Universidad Nacional Mayor de San Marcos, Av. Germán Amezaga N° 375,
Lima, Perú.
* Autor
corresponsal: promeroc@unmsm.edu.pe (P. E. Romero).
ORCID
de los autores
RESUMEN
La Poza La Arenilla es un humedal costero
artificial ubicado en La Punta, Callao, Perú. En nuestra investigación, se
caracterizó la comunidad bacteriana de esta localidad en junio y septiembre de
2024 utilizando metabarcoding de ADN ambiental. La secuenciación
mediante la tecnología Oxford Nanopore identificó cinco filos principales:
Proteobacteria (dominante), Actinobacteriota, Bacteroidota, Campylobacterota y
Desulfobacterota, concordante con patrones de humedales costeros globales. La
comunidad reveló una notable diversidad taxonómica, presentando a los géneros Amylibacter,
Planktomarina y Glaciecola, el género candidato Candidatus
Actinomarina, y grupos marinos como HIMB11, NS11-12 marine group,
OM43 clade, SAR86 clade y Clade Ia. Se observaron
variaciones temporales significativas en los índices de diversidad entre ambos
meses correlacionados con cambios en el oxígeno disuelto, pH, salinidad y
temperatura del agua. La presencia significativa de las categorías
"Otros" y uncultured (no asignados) sugiere la necesidad de
investigaciones futuras para identificar su taxonomía y sus roles en los procesos
biogeoquímicos y como bioindicadores de calidad ambiental, complementando los indicadores
convencionales de la normativa peruana. Este estudio establece la primera línea
base molecular de la diversidad bacteriana en esta localidad, contribuyendo al
desarrollo de estrategias de conservación y gestión ambiental efectivas.
Palabras
clave: microbiota
acuática; ecología microbiana; ecosistema artificial; metabarcoding;
bioindicadores.
ABSTRACT
The Poza La Arenilla is an artificial coastal
wetland located in La Punta, Callao, Peru. Our research characterized
the bacterial community of this ecosystem in June and September 2024 using DNA
metabarcoding. Sequencing through the Oxford Nanopore technology identified
five main phyla: Proteobacteria (dominant), Actinobacteriota, Bacteroidota, Campylobacterota,
and Desulfobacterota, which is consistent with patterns observed in global
coastal wetlands. The bacterial community revealed notable taxonomic diversity,
presenting formally described genera such as Amylibacter, Planktomarina,
and Glaciecola, the candidate genus Candidatus Actinomarina,
and various understudied marine groups including HIMB11, NS11-12 marine group,
OM43 clade, SAR86 clade, and Clade Ia. Significant temporal variations were
observed in diversity indices between months, which is correlated with changes
in physicochemical parameters such as dissolved oxygen, pH, salinity, and water
temperature. The significant presence of categories such as "Others"
and uncultured suggests the necessity of future research to identify their
taxonomy and their ecological roles in biogeochemical processes and as
potential bioindicators of environmental quality, complementing conventional
indicators established in Peruvian regulations. This study establishes the
first molecular baseline of bacterial diversity at this locality, contributing
to effective conservation strategies and environmental management plans.
Keywords:
aquatic
microbiota; microbial ecology; artificial ecosystem; metabarcoding;
bioindicators.
Recibido: 25-07-2025.
Aceptado: 02-12-2025.
La
contaminación representa una creciente amenaza global para los ecosistemas
naturales y la salud humana, siendo los humedales costeros particularmente
vulnerables a estos impactos (Zegarra Tello et al., 2025). La Poza La Arenilla
es un humedal artificial costero de 18,2 Ha limitado por dos rompeolas (Sánchez
et al., 2014), ubicado en el distrito de La Punta, Callao, y construido en
1967. Este ecosistema se caracteriza por sus aguas litorales semiestancadas
debido a la limitada circulación de agua (Cotillo et al., 2018; Velazco &
Solís, 2013).
A pesar de su
cercanía a la zona residencial de La Punta, el conocimiento sobre su
biodiversidad, especialmente respecto a su comunidad bacteriana, sigue siendo
escaso. Esta brecha de conocimiento resulta particularmente relevante
considerando que la interacción entre este humedal artificial y el entorno
urbano circundante podría generar im-pactos significativos en el ecosistema y
la pobla-ción, afectando la calidad del agua, permitiendo la proliferación de
patógenos o alterando los ciclos biogeoquímicos fundamentales.
Estudios microbiológicos
previos en humedales costeros peruanos han empleado predominan-temente métodos
de cultivo tradicionales. Estos métodos permiten identificar entre el 0,1% y el
10% de las bacterias presentes en el ambiente (De la Cruz-Leyva et al., 2015).
Por ello, aún existe un vacío significativo en el conocimiento de la riqueza
microbiana de estos ecosistemas.
La
implementación de técnicas moleculares actuales como el metabarcoding de
ADN ambiental emerge como una solución necesaria para superar estas limitaciones.
Esta metodología genera un perfil representativo de la comunidades bacteria-nas,
detectando tanto grupos cultivables como no cultivables (Padilla-García et al.,
2021).
Recientemente,
la tecnología Oxford Nanopore se ha utilizado para metabar-coding generando
una secuenciación de ampli-cones completos del gen 16S rRNA (Szoboszlay et al.,
2023; Yeo et al., 2024; Cuscó et al., 2019).
Diversos
estudios han demostrado la eficacia del metabarcoding en ecosistemas
acuáticos. Por ejemplo, Cakin et al. (2025) encontraron correla-ciones
significativas entre taxones específicos y eficiencia en la eliminación de
contaminantes en humedales artificiales. Por otro lado, Robertson (2024)
estableció que las comunidades bacterianas sedimentarias constituyen excelentes
bioindicado-res del estado ecosistémico, con variaciones estacionales más
pronunciadas en el agua que en sedimentos.
En el presente
estudio se tuvo como objetivo caracterizar la diversidad bacteriana de la Poza
La Arenilla mediante metabarcoding para establecer la primera línea base
microbiana en este humedal artificial costero. Asimismo, se buscó identificar
la estructura comunitaria bacteriana, sus patrones de variación estacional y su
relación con parámetros fisicoquímicos, trascendiendo los indicadores microbiológicos
convencionales establecidos en la normativa peruana actual (MINAM, 2017), que
solo considera coliformes termotolerantes. Los resulta-dos obtenidos buscan
contribuir al conocimiento de la biodiversidad microbiana en ecosistemas costeros
peruanos, proporcionando información para el desarrollo de estrategias
efectivas de conservación y planes de manejo ambiental.
Área de estudio
Se establecieron cinco puntos de muestreo distribuidos
a lo largo de la Poza La Arenilla (Figura 1), siguiendo la metodología descrita
por Sánchez et al. (2014). La recolección de muestras se efectuó en dos meses,
junio y septiembre de 2024, para evaluar posibles variaciones estacionales en
la composición de la comunidad bacteriana.
Los cinco puntos de muestreo (M1 a M5), con sus
respectivas réplicas biológicas (M1D a M5D), están distribuidos espacialmente
para obtener una representación adecuada del ecosistema.
Los muestreos se realizaron en dos meses del año, junio
y septiembre de 2024. Para garantizar la calidad y reproducibilidad de los
resultados, también se realizaron réplicas técnicas (dos eventos de
secuenciación) para cada periodo:
- Junio: Secuenciaciones J1 (11/7/2024) y J2 (09/11/2024).
- Septiembre: Secuenciaciones S1 (28/10/2024) y S2 (09/11/2024).
Cada muestra está identificada con un código único, un
número de barcode específico y su correspondiente fecha de
secuenciación. Los códigos de muestra siguen una nomenclatura que indica el mes
de colecta, mientras que las fechas de secuenciación fueron: 11/07/2024,
28/10/2024 y 09/11/2024, Tabla 1.
Recolección y procesamiento de muestras
En cada punto de muestreo se recolectó un litro de agua
en recipientes estériles de PVC, los que fueron transportados a 4 °C hasta el
Laboratorio de Biología Integrativa de la Facultad de Ciencias Biológicas
(UNMSM), en un tiempo no mayor a 12 h post-recolección para su procesamiento. Se
contó con la autorización de la Municipalidad de La Punta, Callao (Memorando Nº
280-2024-MDLP-GSC: Autorización para la realización de trabajos para estudio de
investigación en Poza La Arenilla).
Las muestras fueron procesadas mediante un sistema de
filtración por bomba de vacío (Millipore, WP6122050, Alemania) utilizando
filtros SterivexTM Filter Unit (0,22 μm) (Merck Millipore,
SVGP01050, EE. UU.). Los filtros Sterivex fueron sellados en sus extremos con una
película parafilm y almacenados a -20 °C en un congelador (Sole,
3120SOLCONGV135, Perú) hasta la realización del protocolo de extracción de ADN.
Cada filtro fue etiquetado con un código único que incluía la identificación
del punto de muestreo, la fecha de colecta, el número de réplica y la fecha de
filtrado.
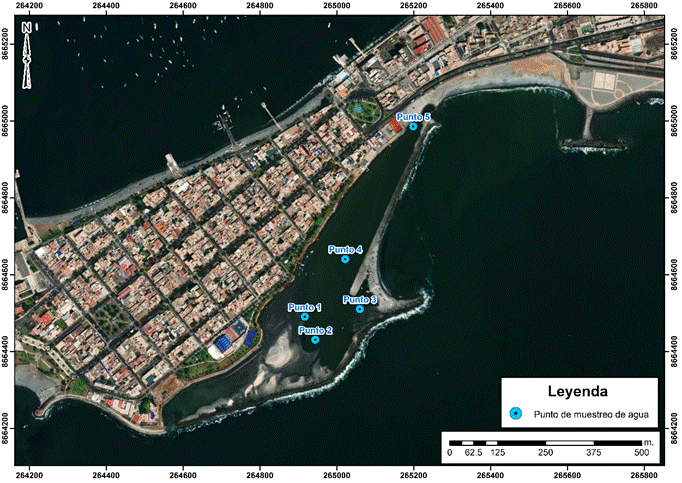
Figura 1. Distribución espacial de
los puntos de muestreo en la Poza La Arenilla, La Punta, Callao. Los cinco
puntos de muestreo fueron seleccionados para cubrir diferentes zonas del
humedal artificial. Las coordenadas están expresadas en sistema UTM WGS 84.
Tabla 1
Metadatos de las
muestras recolectadas
|
N.º
|
Código de Muestra
|
Barcode
|
Mes de Muestreo
|
Fecha de secuenciación
|
|
1
|
M5_J1
|
7
|
Junio 2024
|
11/07/2024
|
|
2
|
M4D_J1
|
12
|
Junio 2024
|
11/07/2024
|
|
3
|
M5_J2
|
7
|
Junio 2024
|
09/11/2024
|
|
4
|
M1D_J2
|
1
|
Junio 2024
|
09/11/2024
|
|
5
|
M2D_J2
|
2
|
Junio 2024
|
09/11/2024
|
|
6
|
M3D_J2
|
3
|
Junio 2024
|
09/11/2024
|
|
7
|
M4D_J2
|
4
|
Junio 2024
|
09/11/2024
|
|
8
|
M5D_J2
|
5
|
Junio 2024
|
09/11/2024
|
|
9
|
M1_S1
|
7
|
Septiembre 2024
|
28/10/2024
|
|
10
|
M2_S1
|
8
|
Septiembre 2024
|
28/10/2024
|
|
11
|
M3_S2
|
11
|
Septiembre 2024
|
09/11/2024
|
|
12
|
M4_S2
|
12
|
Septiembre 2024
|
09/11/2024
|
|
13
|
M5_S2
|
13
|
Septiembre 2024
|
09/11/2024
|
|
14
|
M1D_S2
|
16
|
Septiembre 2024
|
09/11/2024
|
|
15
|
M2D_S2
|
17
|
Septiembre 2024
|
09/11/2024
|
|
16
|
M3D_S2
|
18
|
Septiembre 2024
|
09/11/2024
|
|
17
|
M4D_S2
|
19
|
Septiembre 2024
|
09/11/2024
|
|
18
|
M5D_S2
|
20
|
Septiembre 2024
|
09/11/2024
|
Análisis molecular
Extracción de ADN
La extracción de ADN se realizó con el kit comercial
NucleoMagR DNA/RNA water (Macherey-Nagel, 744220.4, Alemania), con un protocolo
optimizado en cuatro fases secuenciales. Inicialmente, se procedió con la etapa
de lisis celular mediante la adición de 900 μL de Buffer Lisis C1 a cada
filtro Sterivex, seguido de un procesamiento mecánico-térmico que incluyó
agitación vigorosa en vórtex (Gilson, 3611750, EE. UU.) durante cinco minutos,
incubación a 70 °C por cinco minutos adicionales y una agitación final para
maximizar la liberación del material genético. El lisado resultante fue
recuperado mediante un sistema de jeringa estéril y transferido a microtubos
estériles de 1,5 mL. Posteriormente, se realizó la purificación del ADN
transfiriendo 450 μL del lisado obtenido a un nuevo tubo, al cual se
incorporaron 25 μL de partículas magnéticas NucleoMag B-Beads y 475
μL de Buffer MWA2, sometiendo la mezcla a homogeneización por pipeteo
durante cinco minutos antes de efectuar la separación magnética (Cenmed
Enterprises, Genesig EASY-MR, EE. UU.).
La purificación del ADN captado se ejecutó mediante
lavados secuenciales empleando Buffer MWA3 en dos ciclos consecutivos y Buffer
MWA4 en un ciclo final, realizando en cada etapa la correspondiente
homogeneización y separación magnética; el exceso de solvente fue eliminado
exponiendo el tubo abierto al separador magnético durante 15 minutos.
Finalmente, la elución del ADN se completó añadiendo 100 μL de agua libre
de RNasas, seguido de un proceso de resuspensión durante cinco minutos y una
separación magnética final de dos minutos, tras lo cual el ADN purificado en el
sobrenadante fue transferido a un nuevo microtubo estéril y almacenado en
condiciones de congelación para análisis posteriores.
Amplificación del gen 16S
rRNA y preparación de biblioteca genómica
Las reacciones de PCR (reacción en cadena de la
polimerasa) se prepararon en un volumen final de 50 μL, conteniendo: 5
μL de agua libre de RNasa, 10 μL de ADN genómico (10ng), 25 μL
de Long Hot Start Taq polimerasa 2X (New England Biolabs, M0533, EE.UU.) y 10
μL de cada cebador. La amplificación se realizó en un termociclador
Bio-Rad T100™ (Bio-Rad, 1861096, EE.UU.) bajo las siguientes condiciones: 30
ciclos de desnatu-ralización (95 °C, 20 s), hibridación (55 °C, 30 s) y
extensión (65 °C, 2 min), seguidos de un ciclo de extensión final (65°C, 5
min). Las muestras fueron etiquetadas siguiendo un esquema de distribución de
barcodes utilizando el kit de barcoding 16S (Oxford Nanopore
Technologies, SQK-16S024, Reino Unido), permitiendo el procesamiento simultáneo
de múltiples muestras.
Secuenciación usando la
tecnología Oxford Nanopore
La secuenciación se realizó empleando celdas de flujo
Nanopore (Oxford Nanopore Technologies, FLO-MIN106, Reino Unido) en la
plataforma Oxford Nanopore (MinION, Oxford Nanopore Techno-logies, Reino Unido).
El protocolo se desarrolló en cuatro fases secuenciales. Inicialmente, se
formuló una solución de acondicionamiento combinando 30 μL del agente de
anclaje (FLT) con 970 μL de la solución tampón de lavado (FB), generando
un medio óptimo para la activación de los nanoporos. Luego, se procedió al
acondicionamiento de la celda de flujo mediante la introducción de la solución
preparada: La fase de preparación de la celda de flujo consistió en la carga
secuencial del Priming Mix, iniciando con 800 μL seguido de un
período de estabilización de 5 minu y culminando con la adición de los 200
μL restantes para asegurar la distribución uniforme de la solución a
través de la membrana de nanoporos. La tercera fase com-prendió la preparación
de la mezcla de carga: 37,5 μL de SQB (Sequencing Buffer), 25,5
μL de LB (Loading Beads) y 12 μL de la biblioteca genómica
previamente procesada. La etapa de activación del sistema involucró la transferencia
de la mezcla completa (75 μL) a la celda previamente acondi-cionada.
Análisis bioinformático y estadístico
El análisis de los datos producidos por la secuen-ciación
se realizó siguiendo un proceso ordenado. La adquisición inicial de datos fue
mediante el software MinKNOW (Oxford Nanopore Techno-logies, versión 24.06,
Reino Unido), que permitió la supervisión en tiempo real de los parámetros
críticos durante la secuenciación, asegurando la calidad y continuidad del
proceso.
El trabajo bioinformático se desarrolló en el entorno conda
(Anaconda Inc., 2025), facilitando la gestión de paquetes y dependencias para
los análisis computacionales.
La primera fase se centró en la limpieza y control de
calidad de las secuencias crudas, empleando una combinación de herramientas
específicas. Se utilizó Porechop (Wick, 2017) para la identificación y
eliminación precisa de secuencias adaptadoras, seguido por NanoFilt (De Coster
et al., 2018) para realizar un filtrado que preservó únicamente las lecturas
con longitudes entre 250 a 1600 pares de bases (pb) lo que resultó en un
conjunto final de dieciocho (18) muestras (Tabla 1). Este proceso culminó con
la implementación de NanoStat (De Coster et al., 2018) para la evaluación de
los parámetros cualitativos de las secuencias resultantes.
La segunda fase abordó el análisis específico de las
secuencias filtradas. Inicialmente, se aplicaron minimap2 (Li, 2018) y yacrd
(Marijon et al., 2020) para la detección y eliminación de estructuras
quiméricas que pudieran comprometer la interpretación taxonómica.
A continuación, las secuencias depuradas fueron
sometidas a un proceso de alineamiento contra la base de datos Silva_138, un
repositorio especializado en secuencias ribosomales (Quast et al., 2013; Yilmaz
et al., 2014).
Finalmente, se generaron tablas taxonómicas mediante la
aplicación secuencial de los algoritmos filterPAF.py y merfePAF.py (Lapeña,
2023), transformando las coincidencias de alineamiento en clasificaciones
taxonómicas estructuradas.
La fase final consistió en un análisis estadístico
utilizando las plataformas RStudio (Posit, 2023) y Minitab (Minitab, LLC,
2023). Este análisis integró múltiples dimensiones de la diversidad micro-biana,
incluyendo la cuantificación de abundancias relativas de los distintos taxones
identificados, el cálculo de índices de diversidad alfa (Shannon y Simpson)
para caracterizar la riqueza y equita-tividad de las comunidades, y la
evaluación de la diversidad beta obteniendo valores de similitud de Bray-Curtis
que son usados para un análisis de coordenadas principales. Adicionalmente, se
caracterizó la composición taxonómica a nivel de filo y género, y se realizaron
análisis de varianza, pruebas de significancia y evaluaciones de correlación
para identificar patrones significativos en la estructura comunitaria y sus
relaciones con variables ambientales.
Parámetros fisicoquímicos
Las mediciones de parámetros fisicoquímicos se
realizaron in situ utilizando un equipo multípara-métrico Multi 3630 IDS SET G
(WTW, Serie 2FD57G, Alemania), determinando simultánea-mente: oxígeno disuelto,
pH, salinidad y tempe-ratura. Estos datos se utilizaron para correlacionar las
condiciones ambientales y la diversidad bacteriana identificada.
Variaciones estacionales en parámetros fisicoquímicos y
diversidad bacteriana en la Poza La Arenilla
Se observó variación entre los meses de
junio y septiembre de 2024. En particular, las muestras de septiembre
presentaron un ligero incremento en los valores de pH (de 7,9 - 8,0 en junio a
8,0 - 8,1 en septiembre) y oxígeno disuelto (de 6,7 - 10,3 mg/L en junio a 9,1 -
10,3 mg/L en septiembre), mientras que la temperatura mostró una leve
disminución (de 18,0 - 18,6 °C en junio a 17,4 - 18,1 °C en septiembre). Estos
cambios se reflejan también en los índices ecológicos, con valores de
diversidad de Shannon generalmente más altos en septiembre (3,3 - 3,8) en
comparación con junio (2,7 - 3,2). Los valores específicos por punto de
muestreo, junto con los parámetros fisicoquímicos (oxígeno disuelto, pH, salinidad
y temperatura) y los índices ecológicos se encuentran registrados en la Poza La
Arenilla, se detallan en la Tabla 2.
Distribución
del número de secuencias por sitio de muestreo durante dos periodos
estacionales
Los resultados revelan que, en junio, se
observó alta variabilidad en la distribución del número de secuencias entre puntos
de muestreo. En septiembre, se presentó una distribución más uniforme (Figura 2).
Riqueza
taxonómica a nivel de géneros
Se observa una
tendencia estacional en la riqueza taxonómica: las muestras de septiembre
presentan más géneros bacterianos en comparación con las muestras de junio (Figura
3).
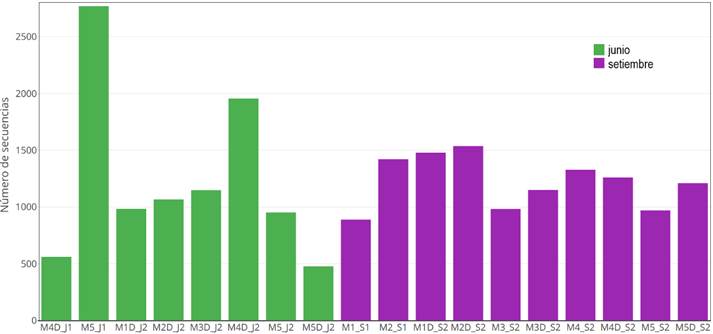
Figura 2. Distribución del número de
secuencias por muestra obtenidas mediante secuenciación de Oxford Nanopore en
junio (barras verdes) y septiembre (barras moradas). Los códigos en el eje X
indican el código de muestra.
Tabla 2
Parámetros fisicoquímicos
e índices ecológicos
|
N.º
|
Código de Muestra
|
pH
|
Salinidad
|
Oxígeno Disuelto
|
Temperatura
|
Índice Shannon
|
Índice Simpson
|
|
1
|
M5_J1
|
8,0
|
35,3
|
7,2
|
18,6
|
3,2
|
0,9
|
|
2
|
M4D_J1
|
8,0
|
35,1
|
10,3
|
18,2
|
3,1
|
0,9
|
|
3
|
M5_J2
|
8,0
|
35,3
|
7,2
|
18,6
|
3,1
|
0,9
|
|
4
|
M1D_J2
|
8,0
|
35,0
|
7,8
|
18,6
|
3,0
|
0,9
|
|
5
|
M2D_J2
|
8,0
|
34,8
|
6,7
|
18,5
|
3,0
|
0,9
|
|
6
|
M3D_J2
|
7,9
|
34,5
|
7,1
|
18,0
|
3,0
|
0,9
|
|
7
|
M4D_J2
|
8,0
|
35,1
|
10,3
|
18,2
|
2,7
|
0,9
|
|
8
|
M5D_J2
|
8,0
|
35,3
|
7,2
|
18,6
|
3,1
|
0,9
|
|
9
|
M1_S1
|
8,1
|
35,4
|
10,3
|
18,0
|
3,1
|
0,9
|
|
10
|
M2_S1
|
8,0
|
35,3
|
9,1
|
17,4
|
3,7
|
1,0
|
|
11
|
M3_S2
|
8,1
|
35,4
|
9,6
|
17,6
|
3,8
|
1,0
|
|
12
|
M4_S2
|
8,1
|
35,4
|
9,1
|
18,1
|
3,5
|
0,9
|
|
13
|
M5_S2
|
8,1
|
35,4
|
9,3
|
18,0
|
3,8
|
1,0
|
|
14
|
M1D_S2
|
8,1
|
35,4
|
10,3
|
18,0
|
3,3
|
0,9
|
|
15
|
M2D_S2
|
8,0
|
35,3
|
9,1
|
17,4
|
3,5
|
0,9
|
|
16
|
M3D_S2
|
8,1
|
35,4
|
9,6
|
17,6
|
3,6
|
0,9
|
|
17
|
M4D_S2
|
8,1
|
35,4
|
9,1
|
18,1
|
3,5
|
1,0
|
|
18
|
M5D_S2
|
8,1
|
35,4
|
9,3
|
18,0
|
3,6
|
1,0
|
Caracterización de la diversidad alfa de la comunidad bacteriana
en Poza La Arenilla
El análisis de la biodiversidad bacteriana
mediante índices ecológicos permitió cuantificar la diversi-dad bacteriana en
la Poza La Arenilla durante junio y septiembre de 2024. Se evidenció variación
en el índice de Shannon, con valores mayores en septiembre (3,1-3,8) en
comparación con junio (2,7 - 3,2) a nivel de género (p < 0,05, en el test de
Mann-Whitney). De manera similar, encontramos que el índice de Simpson también
presenta diferencias significativas entre junio y septiembre (0,9 - 1,0) (p <
0,05, en el test de Mann-Whitney).
Distribución
de principales filos
El análisis reveló la presencia de cinco
filos bacterianos: Proteobacteria, Bacteroidota, Actino-bacteriota, Campylobacterota
y Desulfobacterota. Proteobacteria se mantuvo como el filo dominante, mientras
Bacteroidota aumentó su representación de junio a septiembre. Los filos
Actinobacteriota, Campylobacterota y Desulfobacterota, aunque menos abundantes,
persistieron en ambos periodos de muestreo (Figura 4).
Distribución
de los principales géneros
La comunidad bacteriana de la Poza La
Arenilla reveló una notable diversidad taxonómica, presen-tando géneros
formalmente descritos como Amylibacter, Planktomarina y Glaciecola.
También se identificaron Candidatus_Actinomarina (un género candidato) y diversos
grupos filogenéticos marinos incluyendo HIMB11, NS11-12_marine_group,
OM43_clade, SAR86_clade y Clade_Ia. Cabe destacar que las categorías
"Otros" y
uncultured (no asignados) constituyeron
una proporción significativa del total, evidenciando la presencia de una
considerable diversidad microbiana que aún requiere caracterización completa (Figura
5).
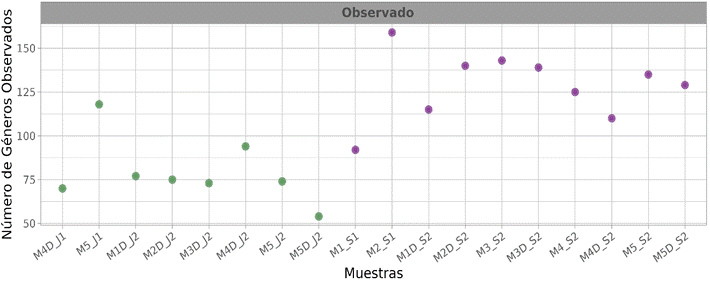
Figura 3. Número de géneros
bacterianos observados en junio (verde) y septiembre (morado).
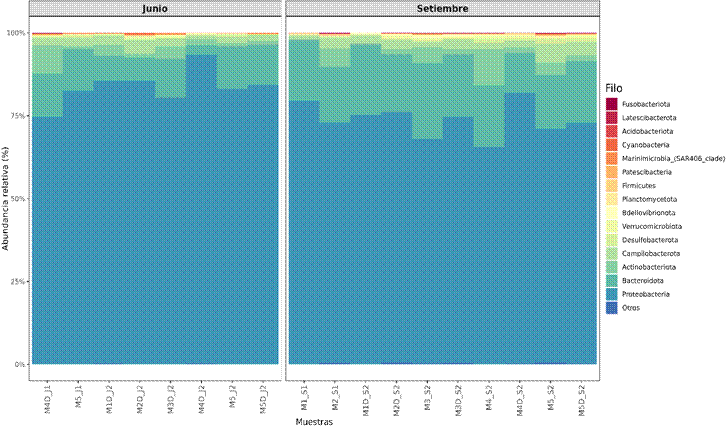
Figura 4. Distribución taxonómica a
nivel de filo de la comunidad bacteriana de Poza La Arenilla. El gráfico
muestra la abundancia relativa de filos bacterianos. Las Proteobacteria
constituyeron el filo dominante en ambos períodos, representando aproximadamente
60-80% de las secuencias totales.
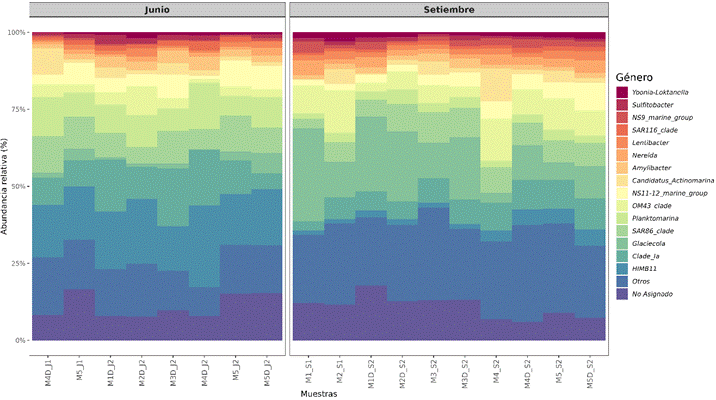
Figura
5. Distribución de los principales
géneros bacterianos en junio y septiembre en la Poza La Arenilla. El gráfico
muestra la abundancia relativa de géneros bacterianos.
Análisis de
coordenadas principales (PCoA) con disimilitud de Bray-Curtis
El análisis de coordenadas principales
(PCoA) a nivel de género revela una clara separación estacional de las
comunidades bacterianas en la Poza La Arenilla entre las estaciones de junio y
septiembre (Figura 6). Esta
marcada
diferenciación está principalmente influenciada por los niveles de oxígeno
disuelto, que muestran el contraste más significativo entre estaciones (6,7 - 10,3
mg/L en junio frente a valores más estables y elevados de 9,1 - 10,3 mg/L en
septiembre). Los cambios en la concentración de oxígeno disuelto parecen ser el
factor ambiental determinante en la estructuración de las comunidades
bacterianas, explicando gran parte de la variación observada en la composición
taxonómica entre ambos meses.
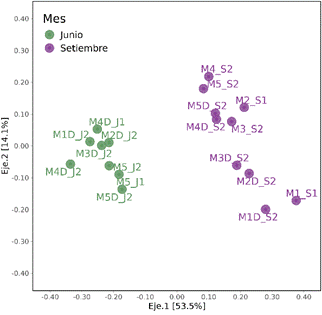
Figura 6. Análisis de Coordenadas
Principales (PCoA) a nivel de género bacteriano en Poza La Arenilla. Las
muestras fueron colectadas en junio (verde) y septiembre (morado).
Esta correlación sugiere que las
fluctuaciones estacionales en la oxigenación del agua constituyen un factor
crítico para la dinámica de la comunidad bacteriana en este humedal costero
artificial.
Análisis estadístico de las variables ambientales
El análisis estadístico reveló correlaciones
signifi-cativas entre los índices ecológicos y los pará-metros fisicoquímicos.
Los datos no cumplieron con los supuestos de normalidad (prueba Ryan-Joiner, p <
0,05), por lo que se emplearon pruebas no paramétricas. Las correlaciones de
Spearman evidenciaron asociaciones positivas significativas del índice de Shannon
con el pH (r = 0,473, p < 0,05) y la salinidad (r = 0,581, p < 0,05),
mientras que se detectó una correlación negativa significativa con la
temperatura (r = -0,650, p < 0,05). Estos resultados indican que condiciones
de mayor pH y salinidad, junto con temperaturas más bajas, favorecen una mayor
diversidad bacteriana en este ecosistema.
Nuestros resultados
proporcionan un aporte significativo al conocimiento de la diversidad
bacteriana en la Poza La Arenilla. La comprensión microbiológica de este ecosistema
ha evolucionado considerablemente a lo largo del tiempo, comen-zando con el
estudio pionero de Yamashiro et al. (1997), quienes registraron bacterias
coliformes fecales utilizando técnicas de cultivo tradicionales. Este estudio
fue ampliado por Sánchez et al. (2014), quienes documentaron niveles elevados
de coliformes totales y termotolerantes que superaban los Estándares de Calidad
Ambiental (ECA) para agua establecidos por el Ministerio del Ambiente (MINAM,
2008). De manera comple-mentaria, investigaciones en ecosistemas similares como
la realizada por Rodríguez et al. (2017), en los Humedales de Ventanilla
(Callao), confirmaron este patrón de elevados niveles de coliformes que superan
los ECA para agua establecidos por el MINAM (2008).
Adicionalmente, el estudio de Sota et al.
(2023) en los Humedales de Huasao (Cusco) encontró altos niveles de
contaminación fecal con coliformes totales, Escherichia coli y Enterococcus
faecalis que superaban los ECA para agua (MINAM,2017).
Si bien los estudios previos se centraron en
indicadores de contaminación como los coliformes, es crucial comprender la
composición completa de las comunidades bacterianas y su dinámica para
implementar estrategias de gestión ambiental. Revisando en la normativa
peruana, ECA para agua (MINAM, 2008, 2017), se observa que se establecen
parámetros para coliformes termotolerantes y otros indicadores microbiológicos,
pero carecen de lineamientos específicos para la caracterización integral de
comunidades bacterianas en ecosistemas artificiales como la Poza La Arenilla e
incluso en los humedales naturales. Esta ausencia de normativa refuerza la
necesidad de desarrollar estudios completos sobre diversidad bacteriana en
estos entornos, ya que los microorganismos no solo son indicadores de
contaminación, sino que cumplen funciones ecológicas esenciales en los ciclos
biogeoquímicos y pueden servir como bioindicadores más precisos del estado
ambiental del ecosistema.
Nuestro estudio representa un avance método-lógico
al implementar tecnologías de secuenciación de nueva generación que permiten
caracterizar no solo indicadores específicos de contaminación, sino la
estructura completa de la comunidad bacteriana y su dinámica estacional. Por ejemplo, las variaciones estacionales
observadas en la diversidad bacteriana de la Poza La Arenilla, muestran
similitudes con los hallazgos de Cakin et al. (2025) quienes documentaron
fluctuaciones en comunidades bacterianas acuáticas en respuesta a factores
ambientales. Estos autores, al igual que nuestro estudio, reportan la
dominancia de Proteobacteria en humedales escoceses donde este filo desempeña
un papel crucial en procesos biogeoquímicos esenciales como la degradación de
materia orgánica y la transformación de nutrientes.
Asimismo, Song et al. (2024), en un estudio
de un humedal costero de China (humedal costero de Tiaozin), confirmaron al
filo Proteobacteria como dominante, gracias a su adaptabilidad a diversos
entornos ecológicos dentro del humedal, como diferentes zonas dominadas por
distintas especies vegetales, suelos con variaciones en salinidad, distintos
rangos de pH, y diferentes contenidos de materia orgánica y nutrientes. Geng et
al. (2020) consideran que las Proteobacterias participan en el ciclo de carbono
y representa el filo bacteriano más común en el mundo y Hartman et al. (2008)
sugirieron su potencial como indicador de calidad ambiental debido a su
respuesta ante cambios en el pH del suelo, contenido de carbono orgánico y
regímenes de humedad. Diversos estudios consideran la importancia de las
Proteobacterias en los procesos de descomposición de materia orgánica y el
ciclo de nutrientes. Por ejemplo, diversos estudios destacan el papel
fundamental de las Proteobacterias en los procesos ecosistémicos de
descomposición de materia orgánica y ciclaje de nutrientes en humedales.
García-Estrada et al. (2024), al investigar las aguadas de la Reserva de la
Biósfera de Calakmul en México, identificaron a Proteobacteria como uno de los
filos dominantes en sedimentos. Su estudio reveló que la diversidad bacteriana
resultó menor en sitios menos perturbados y mayor en áreas con mayor
intervención humana. Particularmente relevante fue la identificación de vías
metabólicas relacionadas con los ciclos del nitrógeno y azufre. En un contexto
urbano similar al de Poza La Arenilla, Castro et al. (2022) estudiaron la
comunidad bacteriana del humedal Santa María del Lago en Bogotá, Colombia, un
sistema de agua dulce, donde identificaron grupos bacterianos abundantes,
incluyendo Proteobacteria, Acidobacteria, Aminicenantes, Bacteroidetes y
Chloroflexi. Aunque Proteobacteria representó uno de los filos más frecuentes,
los autores encontraron que la composición bacteriana se mantuvo relativamente
homogénea entre diferentes zonas del humedal y a lo largo del tiempo. Este
patrón de estabilidad comunitaria resulta interesante en ecosistemas urbanos
que típicamente enfrentan múltiples presiones antropogénicas. La distri-bución
relativamente homogénea de estos grupos entre diferentes áreas de muestreo
sugiere patrones comunitarios consistentes en ecosis-temas urbanos perturbados.
Complementaria-mente, Wei et al. (2021), estudiando humedales de flujo
superficial en Beijing, China, identificaron una comunidad bacteriana
desnitrificante dominada por Proteobacterias no clasificadas (18,16%) y géneros
específicos como Dechloromonas (16,21%), junto con diversos grupos
taxonómicos no clasificados. Este predominio de bacterias asociadas a procesos
de desnitrificación señala la importancia de las comunidades bacterianas en la
regulación del ciclo del nitrógeno en humedales, función particularmente
relevante en ecosistemas sometidos a presiones antropogénicas.
Los filos Campylobacterota y
Desulfobacterota, aunque presentes en baja abundancia, se detectaron
consistentemente en ambos períodos de muestreo. Estos grupos minoritarios
cumplen funciones especializadas en los ciclos del azufre y nitrógeno, lo que
sugiere que mantienen nichos ecológicos específicos dentro del ecosistema. En
contraste, Actinobacteriota mostró mayor abun-dancia y ha sido reportado como
un componente dominante en diversos sistemas acuáticos (Chen et al., 2018;
García-Estrada et al., 2024), participando activamente en la degradación de compuestos
carbonados. La coexistencia de grupos abundantes y minoritarios con funciones
metabólicas comple-mentarias contribuye a la estabilidad del ecosis-tema,
permitiéndole responder adaptativamente a cambios ambientales. Estos filos,
junto con Bacteroidota, han sido registrados en humedales costeros de China,
Estados Unidos, Colombia y Australia (Shen et al., 2023), donde cumplen fun-ciones
ecológicas específicas. Beazley et al. (2012) identificaron a Proteobacteria,
Bacteroidota y Actinobacteriota como degradadores de hidrocar-buros en
humedales estadounidenses, algo que podría también estar ocurriendo en la Poza
La Arenilla, mientras que Morina et al. (2022) correlacionaron la abundancia de
Bacteroidota y Desulfobacterota con mayores concentraciones salinas en
humedales estadounidenses.
La comunidad bacteriana de la Poza La
Arenilla, caracterizada por géneros formalmente descritos como Amylibacter,
Planktomarina y Glaciecola, el género candidato Candidatus_Actinomarina
y grupos filogenéticos marinos como HIMB11, NS11-12_marine_group,
OM43_clade, SAR86_clade y Clade_Ia, representa un ecosistema
microbiano único, cuyo estudio es crucial para comprender la biodiversidad de
este humedal costero artificial peruano. Estos géneros no solo reflejan la
naturaleza transicional del ecosistema entre ambientes marinos y costeros, sino
que desempeñan funciones ecosistémicas clave relacionadas con las condiciones
fisicoquímicas específicas de cada estación (Tabla 3).
Esta diversidad funcional bacteriana
concuerda con lo descrito por García-Estrada et al. (2024), quienes enfatizan
que las comunidades microbianas en humedales no solo reflejan las condiciones
ambientales predominantes, sino que desempeñan roles cruciales en procesos
biogeoquímicos y de biorremediación. Nuestros hallazgos representan una primera
aproximación a la comprensión de un componente microbiano que, como señalan
Gandhi et al. (2018), permanece en gran parte desconocido en muchos
ecosistemas, constituyendo así una contribución científica fundamental para la
caracterización ecológica y el manejo de este humedal costero artificial.
El índice de Shannon reveló un incremento
significativo en la diversidad bacteriana durante septiembre (3,2 – 3,8) en
comparación con junio (2,7 - 3,1), patrón que coincide con lo documentado por
Cakin et al. (2025) en humedales costeros. En contraste, Wei et al. (2021)
documentaron valores del índice de Shannon superiores en humedales naturales de
flujo superficial en China (3,718 en mayo, 4,303 en agosto y 4,432 en octubre).
Esta diferencia podría atribuirse a que nuestro estudio se realizó en un
humedal artificial costero, mientras que Wei et al. analizaron humedales
naturales con mayor heterogeneidad de hábitats. A pesar de esta distinción,
ambos estudios eviden-cian patrones similares de variación temporal en las
comunidades bacterianas, particularmente en la respuesta a factores
fisicoquímicos como tempe-ratura, pH y oxígeno disuelto. Wei et al. (2021)
identificaron una fuerte correlación entre estos parámetros y la abundancia y
diversidad de bacte-rias desnitrificantes, con predominio de Proteo-bacterias
(18,16%) y géneros especializados como Dechloromonas (16,21%) y Rhodanobacter (1,51%). El
Índice de Simpson mostró alta dominancia en ambos períodos, con valores
cercanos a 0.95 en junio, incrementándose ligeramente en septiembre de 0,95 – 1,00
(p < 0,05, test de Mann-Whitney). Esta tendencia refleja cómo Proteobacteria
mantuvo su predominio, mientras Bacteroidota aumentaba su representación en
septiembre, demostrando dinámicas temporales consistentes.
En el presente estudio se documentan cambios
significativos en la diversidad y composición bacte-riana entre junio y
septiembre, con mayor riqueza taxonómica y predominio de diferentes géneros
según la estación. Esto sugiere que, al igual que en el estudio de Wei et al.,
las variaciones estacionales en parámetros ambientales, especialmente tempe-ratura
y oxigenación, influyen decisivamente en la estructura de las comunidades
microbianas, lo que estaría correlacionado con las fluctuaciones esta-cionales
en factores fisicoquímicos que modulan la composición bacteriana, un fenómeno
consistente tanto en ecosistemas naturales como artificiales.
Tabla 3
Filos
bacterianos dominantes en la Poza La Arenilla. Funciones ecológicas potenciales
e importancia de acuerdo a la literatura
|
Filo
|
Funciones
Ecológicas Potenciales
|
Importancia
en Humedales Costeros
|
Referencia
|
|
Proteobacteria
|
•
Descomposición de materia orgánica
•
Participación en ciclos del carbono y nitrógeno
•
Degradación de hidrocarburos
•
Transformación de nutrientes
•
Procesos de Biorremediación
•
Adaptabilidad a diversos gradientes ambientales
|
Filo
más versátil y abundante en humedales costeros. Esencial en el ciclo del
carbono, participando activamente en la descomposición de materia orgánica y
compuestos orgánicos complejos.
|
Song
et al. (2024); Geng et al. (2020); Hartman et al. (2008)
|
|
Bacteroidota
|
•Degradación
de hidrocarburos
•Degradación
de materia orgánica
•
Biorremediación
|
Presente
en diversos tipos de humedales costeros y urbanos. Responde
significativamente a contaminación por hidrocarburos, mostrando incrementos
dramáticos durante eventos de contaminación y posterior disminución al
degradarse los contaminantes.
|
Castro
et al. (2022); Beazley et al. (2012)
|
|
Actinobacteriota
|
•
Degradación de carbono mediante glicósido hidrolasas
•
Producción de metabolitos secundarios
•
Síntesis de antibióticos naturales
•
Amplia cobertura funcional metabólica.
|
Filo
dominante ampliamente distribuido en humedales, con influencia agrícola.
|
Chen
et al. (2018); García-Estrada et al. (2024)
|
|
Desulfobacterota
|
•
Reducción de sulfatos
•
Ciclo del azufre
•
Degradación anaerobia
|
Indicador
de transición salina en humedales costeros. Aumenta progresivamente con
salinización
|
Morina
et al. (2022).
|
La
diversidad microbiana observada en la Poza La Arenilla coincide con patrones
encontrados en otros ecosistemas costeros a nivel mundial, lo que resalta la
necesidad de este tipo de estudios y su replicación en humedales costeros
artificiales para comprender completamente la estructura y función de sus
comunidades bacterianas. Esta información es fundamental para desarrollar
estrategias efectivas de conservación y manejo, como el biomonitoreo molecular
basado en técnicas de metabarcoding de ADN ambiental para la detección
temprana de cambios en la composición microbiana (Robertson, 2024), la
identificación de bioindicadores especí-ficos de calidad ecosistémica que
permitan evaluar objetivamente el impacto de intervenciones (Chen et al.,
2018), y la implementación de aproximaciones de biorremediación microbiana para
degradar contaminantes específicos (Cakin et al., 2024). Además, como sugieren
Esser et al. (2024), la ecogenómica de humedales permite evaluar directamente
los efectos de cambios en la calidad del agua y medir procesos biogeoquímicos
clave como la desnitrificación o la transformación de contami-nantes,
proporcionando información crucial para estrategias adaptativas de gestión que
optimicen los servicios ambientales que este humedal artificial ofrece a la
comunidad circundante.
Los parámetros
fisicoquímicos de la Poza La Arenilla sugieren una relación significativa con
la diversidad bacteriana observada. El pH se mantuvo en rango alcalino con un
ligero incremento entre junio y septiembre, la salinidad permaneció estable (34,5
– 35,4‰) como es característico en ecosis-temas costeros, mientras que el
oxígeno disuelto mostró fluctuaciones estacionales y la temperatura presentó un
descenso sutil entre ambos períodos de muestreo. La literatura científica respalda
la influencia de estos parámetros en comunidades bacterianas. Wei et al. (2021)
documentaron cómo la temperatura y salinidad modifican la estructura microbiana
en humedales chinos, evidenciado por variaciones significativas en el Índice de
Shannon-Wiener (3,718 – 4,432) entre estaciones. Morina et al. (2022)
establecieron correlaciones específicas entre salinidad y abundancia de
Bacteroidota y Desulfobacterota en humedales de Florida, mientras que Geng et
al. (2020) identificaron al pH, humedad y concentración de nutrientes como
factores determinantes en la composición microbiana. Estas evidencias sugieren
que las variaciones fisicoquímicas registradas en la Poza La Arenilla podrían
explicar las fluctuaciones observadas en la abundancia de filos bacterianos,
particularmente Proteobacteria y Bacteroidota.
Desde una perspectiva de
gestión ambiental, los resultados obtenidos sugieren la necesidad de incorporar
el análisis de diversidad bacteriana en los programas de monitoreo de
ecosistemas acuáticos artificiales, complementando los pará-metros
microbiológicos convencionales estable-cidos en la normativa peruana. Esta
información proporcionaría indicadores más sensibles sobre cambios en la
calidad ambiental, permitiendo identificar alteraciones ecosistémicas tempranas
y desarrollar estrategias más efectivas para el control de la contaminación y
la conservación de estos valiosos ecosistemas.
En este estudio se revela
el potencial trans-formador de las tecnologías de secuenciación de nueva
generación para el monitoreo y conservación de humedales costeros artificiales
en Perú. A través del metabarcoding del gen 16S rRNA completo mediante
Oxford Nanopore, hemos identificado una comunidad bacteriana altamente diversa,
con patrones estacionales marcados que responden sensiblemente a variaciones en
parámetros fisicoquímicos clave.
La comunidad microbiana de
este ecosistema, dominada por Proteobacteria (60% - 80%), exhibe una clara
diferenciación entre junio y septiembre, con mayor diversidad y riqueza
taxonómica durante esta última. El oxígeno disuelto emergió como el principal
factor estructurador de la comunidad bacteriana, modulando la distribución de
géneros con funciones ecológicas específicas, desde degradadores de
hidrocarburos hasta microorganismos fotosintéticos y potenciales bioindicadores
de calidad ambiental.
Los perfiles bacterianos
identificados constituyen una herramienta de diagnóstico altamente sensi-ble,
capaz de detectar alteraciones ecosistémicas. Esta sensibilidad permite
anticipar cambios en la calidad ambiental, facilitando intervenciones
preventivas más efectivas y económicas.
La incorporación de los análisis
de diversidad bacteriana en programas regulares de monitoreo ambiental
potenciará significativamente las estrategias de conservación de humedales
costeros, proporcionando indicadores precisos sobre el funcionamiento
ecosistémico, los ciclos biogeoquímicos y los procesos de contaminación antropogénica.
Esta aproximación representa un avance sustancial respecto a los métodos tradi-cionales
basados exclusivamente en coliformes termotolerantes, estableciendo un nuevo
estándar para la evaluación integral de la salud ecosistémica en ambientes
acuáticos.
Futuras
investigaciones deberían enfocarse en tres aspectos fundamentales. Primero,
sería importante realizar análisis funcionales para entender mejor qué procesos
metabólicos realizan estas bacterias, especialmente aquellas relacionadas con
la degra-dación de contaminantes típicos de la zona del Callao. Segundo,
necesitamos datos de períodos más largos que incluyan eventos climáticos
extremos, ya que durante nuestro estudio no pudimos evaluar el impacto de
fenómenos como El Niño. Tercero, sería valioso comparar nuestros resultados con
otros humedales cercanos como Ventanilla o Pantanos de Villa para verificar si
los patrones observados son consistentes. Este tipo de información permitiría
eventualmente desarrollar herramientas de monitoreo más precisas y adaptadas a
nuestra realidad costera, contri-buyendo así a una mejor gestión de estos
importantes ecosistemas.
Los
autores expresan su gratitud al Vicerrectorado de Investigación y Posgrado de
la Universidad Nacional Mayor de San Marcos que a través del proyecto de
investigación PCONFIGI INV B23100181 "adÁn: prototipo de sistema de
monitoreo rápido de biodiversidad usando ADN ambiental (2023)" financió
este estudio. También, reconocemos el apoyo técnico del personal del
Laboratorio de Biología Integrativa de la Facultad de Ciencias Biológicas por
facilitar el acceso a equipamiento y brindar soporte durante la fase
experimental del proyecto.
REFERENCIAS
BIBLIOGRÁFICAS
Anaconda Inc. (2025). Conda
documentation. https://www.anaconda.com/docs/main
Beazley, M. J., Martinez,
R. J., Rajan, S., Powell, J., Piceno, Y. M., Tom, L. M., Andersen, G. L.,
Hazen, T. C., Van Nostrand, J. D., Zhou, J., Mortazavi, B., & Sobecky, P.
A. (2012). Microbial
community analysis of a coastal salt marsh affected by the Deepwater Horizon
oil spill. PLOS ONE, 7(7), e41305. https://doi.org/10.1371/journal.pone.0041305
Benítez-Páez, A., Portune, K.
J., & Sanz, Y. (2016). Species-level resolution of 16S rRNA gene amplicons
sequenced through the MinION™ portable nanopore sequencer. GigaScience, 5(1),
s13742-016-0111-z. https://doi.org/10.1186/s13742-016-0111-z
Bouchiat, C., Ginevra, C.,
Benito, Y., Gaillard, T., Salord, H., Dauwalder, O., Laurent, F., &
Vandenesch, F. (2022). Improving the diagnosis of bacterial infections:
Evaluation of 16S rRNA Nanopore metagenomics in culture-negative samples. Frontiers
in Microbiology, 13, 943441. https://doi.org/10.3389/fmicb.2022.943441
Cakin, I., Marcello, L.,
Morrissey, B., Gaffney, P. P. J., & Taggart, M. A. (2025). Long-term
monitoring of constructed wetlands in distilleries in Scotland - Evaluating
treatment performance and seasonal microbial dynamics. Journal of
Environmental Management, 375, Article 124279. https://doi.org/10.1016/j.jenvman.2025.124279
Cakin, I., Morrissey, B.,
Marcello, L., Gaffney, P. P. J., Pap, S., & Taggart, M. A. (2024). A
comparison between constructed wetland substrates: Impacts on microbial
community and wastewater treatment. Chemosphere, 364, Article 143179. https://doi.org/10.1016/j.chemosphere.2024.143179
Castro, M., Pacheco, M.,
& Restrepo, M. (2022). Bacterial communities in sediments of an urban
wetland in Bogota, Colombia. Universitas Scientiarum, 27(2), 163-185. https://doi.org/10.11144/Javeriana.SC272.bcis
Chen, W., Wilkes, G., Khan,
I. U. H., Pintar, K. D. M., Thomas, J. L., Lévesque, C. A., Chapados, J. T.,
Topp, E., & Lapen, D. R. (2018). Aquatic bacterial communities associated
with land use and environmental factors in agricultural landscapes using a
metabarcoding approach. Frontiers in Microbiology, 9, 2301. https://doi.org/10.3389/fmicb.2018.02301
Cotillo, A., Podestá, J.,
Segura-Cobeña, E., & Cabanillas, G. (2018). Distribución espacial de las
aves playeras limícolas para once zonas descritas en el humedal costero Poza de
la Arenilla La Punta, Callao. The Biologist (Lima), 16(1), 119-137. https://doi.org/10.24039/rtb2018161226
Cuscó, A., Catozzi, C.,
Viñes, J., Sanchez, A., & Francino, O. (2019). Microbiota profiling with
long amplicons using Nanopore sequencing: full-length 16S rRNA gene and the
16S-ITS-23S of the rrn operon [version 2; peer review: 2 approved, 3 approved
with reservations]. F1000Research, 7, 1755. https://doi.org/10.12688/f1000research.16817.2
De Coster, W., D'Hert, S.,
Schultz, D. T., Cruts, M., & Van Broeckhoven, C. (2018). NanoPack:
visualizing and processing long-read sequencing data. Bioinformatics, 34(15),
2666–2669.
https://doi.org/10.1093/bioinformatics/bty149
De la Cruz-Leyva, M. C.,
Zamudio-Maya, M., Corona-Cruz, A. I., González-de la Cruz, J. U., &
Rojas-Herrera, R. A. (2015). Importancia y estudios de las comunidades
microbianas en los recursos y productos pesqueros. Ecosistemas y Recursos Agropecuarios,
2(4), 99-115.
Ercole, E., Adamo, M.,
Lumini, E., Fusconi, A., & Mucciarelli, M. (2022). Alpine constructed
wetlands: A metagenomic analysis reveals microbial complementary structure. Science
of The Total Environment, 822, Article 153640. https://doi.org/10.1016/j.scitotenv.2022.153640
Esser, M., Brinkmann, M.,
& Hecker, M. (2024). Solving freshwater conservation challenges through
next-generation sequencing approaches. Environmental Science Advances, 3(9),
1181-1196. https://doi.org/10.1039/d4va00112e
Gandhi, J. K., Tatu, K.,
& Kamboj, R. D. (2018). A review of studies on bacterial and fungal
diversity in wetland ecosystems. Research & Reviews: A Journal of
Microbiology and Virology, 8(1), 25-38.
García-Estrada, D. A.,
Selem-Mojica, N., Martínez-Hernández, A., Moreno-Hagelsieb, G., &
Escalante, A. E. (2024). Diversity of bacterial communities in wetlands of
Calakmul Biosphere Reserve: A comparative analysis between conserved and
semi-urbanized zones in pre-Mayan Train era. BMC Microbiology, 24, 376. https://doi.org/10.1186/s12866-024-03523-x
Geng, X. D., Zhou, Y.,
Wang, C. Z., Yu, M. H., & Qian, J. L. (2020). Bacterial community structure
and diversity in the soil of three different land use types in a coastal
wetland. Applied Ecology and Environmental Research, 18(6), 8131-8144. https://doi.org/10.15666/aeer/1806_81318144
Hartman, W. H., Richardson,
C. J., Vilgalys, R., & Bruland, G. L. (2008). Environmental and
anthropogenic controls over bacterial communities in wetland soils.
Proceedings of the National Academy of Sciences, 105(46), 17842-17847. https://doi.org/10.1073/pnas.0808254105
Heikema, A. P.,
Horst-Kreft, D., Boers, S. A., Jansen, R., Hiltemann, S. D., de Koning, W.,
Kraaij, R., de Ridder, M. A. J., van Houten, C. B., Bont, L. J., Stubbs, A. P.,
& Hays, J. P. (2020). Comparison of Illumina versus Nanopore 16S rRNA Gene
Sequencing of the Human Nasal Microbiota. Genes, 11(9), 1105.https://doi.org/10.3390/genes11091105
Kai, S., Matsuo, Y.,
Nakagawa, S., Kryukov, K., Matsukawa, S., Tanaka, H., Iwai, T., Imanishi, T.,
& Hirota, K. (2019). Rapid bacterial identification by direct PCR
amplification of 16S rRNA genes using the MinION™ nanopore sequencer. FEBS
Open Bio, 9(3), 548-557. https://doi.org/10.1002/2211-5463.12590
Lapeña, A. (2023).
Spaghetti [Software]. GitHub. https://github.com/adlape95/Spaghetti
Li, H. (2018). Minimap2:
pairwise alignment for nucleotide sequences. Bioinformatics, 34(18),
3094-3100. https://doi.org/10.1093/bioinformatics/bty191
Marijon, P., Chikhi, R.,
& Varré, J.-S. (2020). yacrd and fpa: upstream tools for long-read genome
assembly. Bioinformatics,
36(12),
3894-3896. https://doi.org/10.1093/bioinformatics/btaa262
Ministerio del Ambiente.
(2008). Decreto Supremo Nº 002-2008-MINAM. Aprueban los Estándares Nacionales
de Calidad Ambiental para Agua. El Peruano.
Ministerio del Ambiente.
(2017). Decreto Supremo N° 004-2017-MINAM. Aprueban Estándares de Calidad
Ambiental (ECA) para Agua y establecen Disposiciones Complementarias. El
Peruano.
Minitab, LLC. (2023).
Minitab Statistical Software. https://www.minitab.com/en-us/
Morina, J. C., &
Franklin, R. B. (2022). Intensity and duration of exposure determine
prokaryotic community response to salinization in freshwater wetland soils. Geoderma,
428, 116138. https://doi.org/10.1016/j.geoderma.2022.116138
Narciso-Mezones, A.,
Merma-Mora, L., Córdova-Rodríguez, K., Rocca, C., & otros. (2025).
Foraminiferal assemblages and organic matter relationships in a coastal lagoon
on the arid coast of the Southeast Pacific. Marine Ecology Progress Series, 754, 17-33. https://doi.org/10.3354/meps14773
Oxford Nanopore
Technologies. (s.f.). Experiment companion - MinKNOW. https://nanoporetech.com/es/document/experiment-companion-minknow
Padilla-García, C. Y.,
Camacho-Sánchez, F. Y., & Reyes-López, M. A. (2021). Metabarcoding de DNA
ambiental: un enfoque para el seguimiento de la biodiversidad. CienciaUAT, 16(1), 136-149. https://doi.org/10.29059/cienciauat.v16i1.1509
Posit. (2023). Download
RStudio Desktop. https://posit.co/download/rstudio-desktop/
Quast, C., Pruesse, E.,
Yilmaz, P., Gerken, J., Schweer, T., Yarza, P., Peplies, J., & Glöckner, F.
O. (2013). The SILVA ribosomal RNA gene database project: improved data
processing and web-based tools. Nucleic Acids Research, 41(D1),
D590-D596.
https://doi.org/10.1093/nar/gks1219
Rieder, J., Kapopoulou, A.,
Bank, C., & Adrian-Kalchhauser, I. (2023). Metagenomics and metabarcoding
experimental choices and their impact on microbial community characterization
in freshwater recirculating aquaculture systems. Environmental Microbiome,
18(1),
Article 8. https://doi.org/10.1186/s40793-023-00459-z
Robertson, C. (2024). Beyond the
surface: Enhancing freshwater pond ecosystem assessment through eDNA
metabarcoding [Tesis doctoral no publicada]. Cardiff University.
Rodríguez, R., Retamozo-Chavez,
R., Aponte, H., & Valdivia, E. (2017). Evaluación microbiológica de un cuerpo de agua del
ACR humedales de Ventanilla (Callao, Perú) y su importancia para la salud
pública local. Ecología Aplicada, 16(1), 15-21. https://doi.org/10.21704/rea.v16i1.899
Rodríguez-Pérez, H.,
Ciuffreda, L., & Flores, C. (2020). NanoCLUST: a species-level
analysis of 16S rRNA nanopore sequencing data. Bioinformatics, 37(11), 1600-1601. https://doi.org/10.1093/bioinformatics/btaa900
Sánchez, G., Flores, V.,
& Henostroza, A. (2014). Calidad ambiental del humedal Poza La
Arenilla-Callao, 2008. Informe Instituto del Mar del Perú, 41(1-4).
Santos, A., van Aerle, R.,
Barrientos, L., & Martinez-Urtaza, J. (2020). Computational methods for
16S metabarcoding studies using Nanopore sequencing data. Computational and
Structural Biotechnology Journal, 18, 296-305. https://doi.org/10.1016/j.csbj.2020.01.005
Shen, L., Hu, L., Wu, H.,
Yan, B., & Song, A. (2023). Microorganisms in coastal wetland sediments: A
review on microbial community structure, functional gene, and environmental
potential. Frontiers in Microbiology, 14, 1163896. https://doi.org/10.3389/fmicb.2023.1163896
Smith, A. M., Jain, M.,
Mulroney, L., Garalde, D. R., & Akeson, M. (2019). Reading canonical and
modified nucleobases in 16S ribosomal RNA using nanopore native RNA sequencing.
PLOS ONE, 14(5), e0216709. https://doi.org/10.1371/journal.pone.0216709
Song, B., Wang, T., Wan,
C., Cai, Y., Mao, L., Ge, Z., & Yang, N. (2024). Diversity patterns and
drivers of soil bacterial and fungal communities in a muddy coastal wetland of
China. Journal
of Fungi, 10(11),
770. https://doi.org/10.3390/jof10110770
Sota-Cano, A.,
Cáceres-Vizarreta, A. I., & Anchari-Oblitas, Y. (2023). Caracterización
microbiológica del cuerpo de agua de los humedales de Huasao (Oropesa-Cusco,
Perú). Hatun Yachay Wasi, 2(1), 7-14. https://doi.org/10.57107/hyw.v2i1.31
Szoboszlay, M., Schramm,
L., Pinzauti, D., Scerri, J., Sandionigi, A., & Biazzo, M. (2023). Nanopore is
preferable over Illumina for 16S amplicon sequencing of the gut microbiota when
species-level taxonomic classification, accurate estimation of richness, or
focus on rare taxa is required. Microorganisms, 11(3), 804. https://doi.org/10.3390/microorganisms11030804
Velazco, F., & Solís,
J. (2013). Caracterización de los sedimentos de la Poza La Arenilla - Callao,
marzo 2004. Informe
Instituto del Mar del Perú, 40(3-4).
Wei, J., Cui, L., Li, W.,
Zhang, Y., Wang, J., Lei, Y., Liu, W., Zhang, M., Lei, F., & Wei, Y.
(2021). Denitrifying
bacterial communities in surface-flow constructed wetlands during different
seasons: Characteristics and relationships with environment factors. Scientific
Reports, 11, 4918. https://doi.org/10.1038/s41598-021-82438-3
Wick, R. (2017). Porechop:
adapter trimmer for Oxford Nanopore reads. GitHub. https://github.com/rrwick/Porechop/
Winand, R., Bogaerts, B.,
Hoffman, S., Lefevre, L., Delvoye, M., Braekel, J. V., Fu, Q., Roosens, N. H.,
Keersmaecker, S. C., & Vanneste, K. (2019). Targeting the 16S rRNA gene for
bacterial identification in complex mixed samples: Comparative evaluation of
second (Illumina) and third (Oxford Nanopore Technologies) generation
sequencing technologies. International Journal of Molecular Sciences, 21(1),
298. https://doi.org/10.3390/ijms21010298
Yamashiro, C., Tafur, R.,
Jacinto, M. E., Morón, O., Lostaunau, N., Delgado, C., Gómez, O., &
Arrieta, S. (1997). Determinación
de las condiciones bioambientales de la poza La Arenilla, La Punta, Callao.
Informe Progresivo Instituto del Mar del Perú, 51(1), 3-26.
Yeo, K., Connell, J.,
Bouras, G., Smith, E., Murphy, W., Hodge, J.-C., Krishnan, S., Wormald, P.-J.,
Valentine, R., Psaltis, A. J., Vreugde, S., & Fenix, K. A. (2024). A comparison
between full-length 16S rRNA Oxford nanopore sequencing and Illumina V3-V4 16S
rRNA sequencing in head and neck cancer tissues. Archives of Microbiology,
206(6), 248. https://doi.org/10.1007/s00203-024-03985-7
Yilmaz, P., Parfrey, L. W.,
Yarza, P., Gerken, J., Pruesse, E., Quast, C., Schweer, T., Peplies, J.,
Ludwig, W., & Glöckner, F. O. (2014). The SILVA and "All-species
Living Tree Project (LTP)" taxonomic frameworks. Nucleic Acids Research,
42(D1), D643-D648. https://doi.org/10.1093/nar/gkt1209
Zegarra Tello, I. J.,
Cabrera Carranza, C. F., Tinoco Gómez, O. R., & Moore Torres, R. K. (2025).
Contaminación del aire y suelo del humedal marino costero por material
particulado de fuentes industriales en el Callao, Perú. Revista del
Instituto de Investigación de la Facultad de Minas, Metalurgia y Ciencias
Geográficas, 28(55), e26394. https://doi.org/10.15381/iigeo.v28i55.26394
Zhang, T., Li, H., Ma, S.,
Cao, J., Liao, H., Huang, Q., & Chen, W. (2023). The newest Oxford Nanopore
R10.4.1 full-length 16S rRNA sequencing enables the accurate resolution of
species-level microbial community profiling. Applied and Environmental
Microbiology, 89(10), e0060523. https://doi.org/10.1128/aem.00605-23






